Estudi Genòmic Computacional de la proteïna Kua-UEV
Martina Illa Mestre (martina.illa01@campus.upf.edu) i Anna Julià Valls (anna.julia01@campus.upf.edu)
Facultat de Ciències de la Salut i de la Vida
Universitat Pompeu Fabra
Índex
Resum
La proteïna Kua-UEV és una proteïna de fusió de dos dominis codificada per als gens Kua i UBE2V1, aquest últim també anomenat com a UEV1. Ambdós gens es poden transcriure tan independentment com conjuntament. La co-transcripció d'UBE2V1 juntament amb Kua, que és adjacent en 'upstream', dóna lloc a un transcript no massa freqüent (Kua-UEV), el qual resulta la proteïna de fusió esmentada. Les proteïnes UEV són variants inactives dels enzims conjugats a ubiquitines E2 que regulen l'elongació no canònica de les cadenes d'ubiquitines; són una subfamília de les proteïnes E2. La característica bàsica que les diferencia és la pèrdua d'un residu de cisteïna, cosa que els fa perdre la seva activitat catalítica (com hem comentat, són variants inactives). La proteïna resultant, localitzada a nivell nuclear, pot causar activació transcripcional de l'oncogen FOS i està en controvèrsia la seva possible implicació en l'alteració del cicle cel.lular. D'altra banda, el producte de Kua es troba situat al citoplasma. D'aquest segon gen no es disposa de gaire informació a causa que la seva recent identificació. Sobre la funció de Kua-UEV és important comentar que es manté la funció de UBE2V1 però Kua hi confereix noves propietats biològiques, com per exemple la seva nova localització al citoplasma.
Torna a l'índexResultats i Discussió
A continuació, passarem a comentar els diferents aspectes estudiats. L'estudi ha estat realitzat tan pels dos gens (Kua i UBE2V1) independentment com per la seva transcripció conjunta (Kua-UEV).
Estructura genòmica
La proteïna Kua-UEV que estudiem és una proteïna de fusió de dos gens contigus del cromosoma 20. Concretament es troben a la banda q13.13. Aquests dos gens són Kua i UBE2V1. A la figura 1 podem veure els diferents transcrits que té cada gen amb la seva corresponent representació exònica. Veiem que els gen Kua té 6 exons mentre que UBE2V1 en té 4. Aquests resultats han estat comprovats amb diferents programes de predicció de gens.
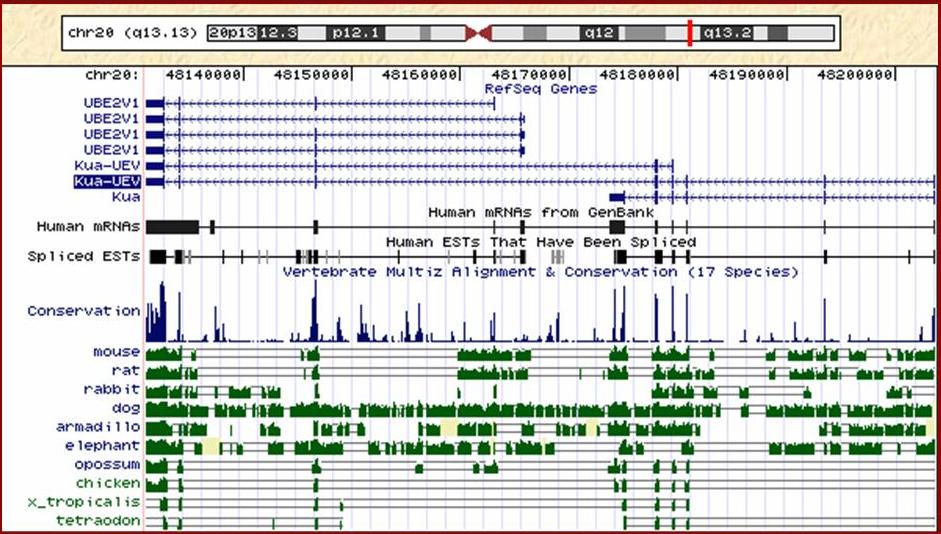
Figura 1. Observem que la proteïna Kua-UEV té dues isoformes; una llarga i una curta. La proteïna Kua té un únic transcrit i la proteïna UBE2V1 en té 4 de diferents.
Durant la transcripció, en alguns casos, i a causa de l'splicing alternatiu, els cinc primers exons de Kua i els tres últims de UBE2V1 formen la isoforma llarga, tal i com podem veure en la figura 2. La isoforma més curta, en canvi, està formada pels exons 4 i 5 de Kua i els tres últims de UBE2V1. Tot i així, el transcrit que es forma més habitualment és el corresponent a la isoforma llarga.
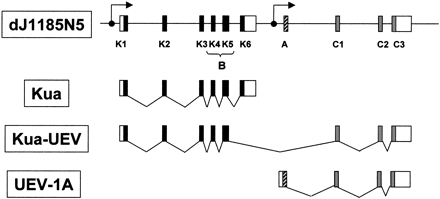
Figura 2. DJ1185N5 és un clon PAC que conté part de la seqüència del cromosoma 20. En aquesta regió es troben els gens Kua i UBE2V1 (UEV-1), els quals tenen un promotor a 'upstream' que els permet transciure's de forma independent. Tot i així, en rares ocasions, la transcripció del promotor de Kua pot donar lloc a un transcrit híbrid (Kua-UEV) dels dos gens a causa d'splicing alternatiu.
Cal destacar que els gens Kua i UBE2V1 es troben a l'strand negatiu de la seqüència d'ADN, per tant primer es produirà la transcripció de Kua i després la de UBE2V1 (veure Figura 3). Així, la proteïna híbrida resultant dels dos gens, conté una primera part provinent de Kua i una segona part provinent de UBE2V1.
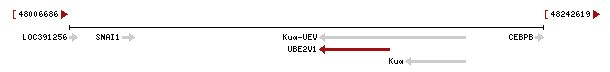
Figura 3.
El transcrit de la proteïna Kua és el següent:
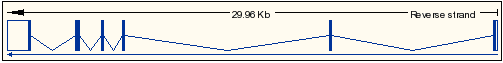
- Referència de la seqüència: NM_199129
- Posició: chr20:48173717-48203667
- Banda: 20q13.13
- Tamany del gen: 29951
- Nombre d'aminoàcids de la proteïna: 270 aa
- Nombre d'exons: 6
- Seqüència exónica
Aquest únic transcrit de Kua conté 6 exons, tots ells codificants. Tot i així, tant el primer com l'últim no ho són totalment, ja que les seves seqüències dels extrems no es codifiquen. Per tant, la proteïna Kua és el resultat de la traducció total dels exons 2-5 i de la traducció parcial dels exons 1 i 6 del gen Kua.
El transcrit 1 de la proteïna UBE2V1 és el següent:
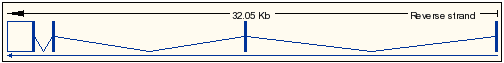
- Referència de la seqüència: NM_001032288.1
- Posició: chr20:48131070-48163117
- Banda: 20q13.13
- Tamany del gen: 32048
- Nombre d'aminoàcids de la proteïna: 147 aa
- Nombre d'exons: 4
- Seqüència exónica
El transcrit 1 del gen UBE2V1 conté 4 exons, dels quals el primer i l'últim són codificants parcialment (la part del extrems no es tradueixen) i els 2 i 3 ho són totalment.
El transcrit 2 de la proteïna UBE2V1 és el següent:
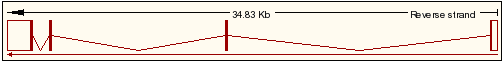
- Referència de la seqüència: NM_021988.3
- Posició: chr20:48131070-48165901
- Banda: 20q13.13
- Tamany del gen: 34832
- Nombre d'aminoàcids de la proteïna: 170 aa
- Nombre d'exons: 4
- Seqüència exónica
El transcrit 3 de la proteïna UBE2V1 és el següent:
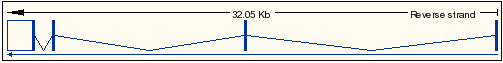
- Referència de la seqüència: NM_199144.1
- Posició: chr20:48131070-48165901
- Banda: 20q13.13
- Tamany del gen: 34832
- Nombre d'aminoàcids de la proteïna: 170 aa
- Nombre d'exons: 5
- Seqüència exónica
Els transcrits 2 i 3 són considerats com a diferents, ja que la seqüència dels seus mRNA no són equivalents, tot i traduir-se a una proteïna amb exactament el mateix número i tipus d'aminoàcids. La diferència recau en què el transcrit 3, té una llargada d'mRNA 78 pb superiors. Aquests nucleòtids no influeixen en la proteïna final, ja que es troben en la part no codificant de l'exó 1. En aquests dos transcrits, el primer exó i l'últim són parcialment codificants i la resta són totalment traduits a proteïna.
El transcrit 4 de la proteïna UBE2V1 és el següent:

- Referència de la seqüència: NM_022442.3
- Posició: chr20:48131070-48165898
- Banda: 20q13.13
- Tamany del gen: 34829
- Nombre d'aminoàcids de la proteïna: 103 aa
- Nombre d'exons: 3
- Seqüència exónica
El transcrit 4 conté 3 exons codificants, dels quals la seqüència dels extrems dels 1 i 3 no es tradueix. L'exó 2 es tradueix totalment.
El transcrit 1 (isoforma llarga) de la proteïna de fusió Kua-UEV és el següent:
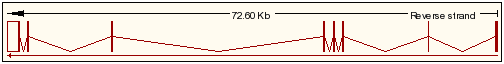
- Referència de la seqüència: NM_199203.1
- Posició: chr20:48131070-48203667
- Banda: 20q13.13
- Tamany del gen: 72598
- Nombre d'aminoàcids de la proteïna: 370 aa
- Nombre d'exons: 8
- Seqüència exónica
El transcrit 2 (isoforma curta) de la proteïna de fusió Kua-UEV és el següent:
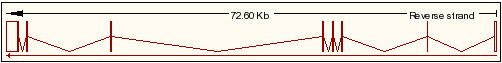
- Referència de la seqüència: NM_003349.4
- Posició: chr20:48131070-48179589
- Banda: 20q13.13
- Tamany del gen: 48520
- Nombre d'aminoàcids de la proteïna: 221 aa
- Nombre d'exons: 5
- Seqüència exónica
La proteïna de fusió Kua-UEV té 2 isoformes (llarga i curta), provinents de dos transcrits diferents. En aquests dos transcirts, UBE2V1 aporta la mateixa seqüència d'aminoàcids provinent dels 3 últims exons dels diferents transcrits d'aquest gen. Per tant, per a l'obtenció de Kua-UEV és indiferent quin transcrit de UBE2V1 es transcrigui. La diferència entre aquestes dues isoformes recau en la diferencial transcripció del gen Kua. En el transcrit llarg de la proteïna híbrida es transcriuen tots els exons de Kua menys l'últim, en canvi en el trancrit curt només ho fan el quart i el cinquuè.
Tal i com podem veure a la figura 4, tan per la isoforma llarga com per la curta, els aminoàcids que provenen de UBE2V1 són els mateixos (marcat en verd), en canvi, els que provenen de Kua són diferent per a cadascuna d'aquestes isoformes de la proteïna híbrida. Per obtenir l'isoforma llarga cal de la traducció dels exons de l'1 al 5 de Kua i dels tres últims de UBE2V1. Així, aquesta isoforma està codificada per 8 exons en total. Per l'expressió de l'isoforma curta (marcat en taronja) es requereix de la traducció dels exons 4 i 5 de Kua i també dels tres últims de UBE2V1. Aquesta isoforma de la proteïna híbrida està codificada per un total de 5 exons.

Figura 4. Seqüència protèica de Kua-UEV per les dues isoformes que presenta. La que es produeix més habitualment és la isoforma llarga de la proteïna de fusió.
Els dos transcrits de Kua-UEV tenen el potencial per codificar per una proteïna amb dos dominis; un prominent de Kua i l'altre de UBE2V1 (els dominis seràn comentats en l'apartat de la funció del gen).
Torna a l'índexEstudi de l'homologia del gen
El fet que tan les proteïnes E2 com totes les seves subfamílies (com la inactiva UEV) relacionades estiguin involucrades en diversos processos cel.lulars fonamentals (veure apartat de l'estudi de la funció) suggereix que tota la via d'ubiquitinació representa un sistema clau i molt antic en la història evolutiva dels eucariotes. Això queda demostrat amb el fet que, com podem observar a les següents taules, existeix una alta homologia dels gens d'estudi del nostre treball. El gen Kua i el UBE2V1 presenten homologia amb gairebé els mateixos animals i el percentatge d'homologia és molt semblant pel que ens atrevim a afirmar que tenen un nivell de conservació molt semblant. Per tal de visualitzar l'evolució que ha patit cada gen, al final de cada taula s'ha realitzat un arbre filogenètic que mostra les distàncies entre els diferents homòlegs de cada gen.
Els homòlegs del gen Kua presenten una similitut molt gran (>88%) en espècies de mamífers. Aquest percentatge d'homologia va disminuint a mesura que l'organisme s'allunya evolutivament de l'Homo sapiens. Sorprenentment, el gos sembla més proper a l'humà que al ratolí. I, com sabem, això no és així. Aquest fet és degut a que la seqüència del gos té algunes llacunes que han estat omplertes amb seqüències humanes.
| Espècie | Nom del gen | Referència del gen | Homolgia DNA (%) | Homolgia proteïna (%) | Imatge | C.familiaris | LOC611401 | XP_854159.1 | 96.4 | 98.3 |  |
| M.musculus | Al840826 | NP_663513.1 | 88.5 | 93.3 |  |
| R.norvegicus | Kua_predicted | XP_342589.3 | 88.0 | 92.3 |  |
| G.gallus | LOC419354 | XP_417513.1 | 69.0 | 76.5 |  |
| D.melanogaster | Kua | NP_610026.1 | 61.3 | 59.5 |  |
| A.gambiae | ENSANGG00000010642 | XP_318759.2 | 63.0 | 63.0 |  |
| C.elegans | Y53C10A.5 | NP_493036.1 | 56.1 | 55.9 |  |

Els homòlegs del gen UBE2V1, tal i com s'ha explicat, presenten una homologia entre les espècies molt semblant a la del gen Kua. La família de les proteïnes UEV està molt conservat tan a nivell de seqüència com a nivell d'estructura en tots els organismes eucariotes. Aquest fet és degut a la seva implicació en processos cel.lulars fonamentals per a la supervivència de l'organisme. A més a més, com es pot veure en aquesta imatge, la família sencera de proteïnes E2 té un ancestre comú.
| Espècie | Nom del gen | Referència del gen | Homolgia DNA (%) | Homolgia proteïna (%) | Imatge |
| P.troglodytes | LOC745497 | XP_001166716.1 | 99.1 | 98.6 |  |
| C.familiaris | LOC477262 | XP_534454.2 | 98.4 | 100.0 |  |
| M.musculus | Ube2v1 | NP_075719.1 | 87.1 | 93.9 |  |
| R.norvegicus | Ube2v1_predicted | XP_215948.4 | 87.3 | 93.9 |  |
| G.gallus | LOC419354 | XP_417514.1 | 91.6 | 98.6 |  |
| D.melanogaster | Uev1A | NP_647959.1 | 59.3 | 66.9 |  |
| A.gambiae | ENSANGG00000019247 | XP_308820.2 | 63.0 | 70.4 |  |
| A.thaliana | AT1G70660 | NP_564994.1 | 56.0 | 53.7 |  |
Els homòlegs del gen híbrid (Kua_UEV) són molt més reduïts en la base de dades consultada. Creiem que aquest fet es deu a què aquesta fusió no ha estat gaire estudiada en diverses espècies.
| Espècie | Nom del gen | Referència del gen | Homolgia DNA (%) | Homolgia proteïna (%) | Imatge |
| P.troglodytes | LOC458329 | XP_514718.2 | 99.8 | 99.7 |  |
| A.thaliana | AT1G23260 | NP_564191.1 | 56.5 | 54.5 |  |
| O.sativa | OSJNBb003 | XP_469523.1 | 55.9 | 50.4 |  |
| P.falciparum | MAL3P2.20 | NP_473184.1 | 54.8 | 49.3 |  |

Caracterització de l'expressió del gen
Per tal de mostrar els llocs d'expressió dels diferents gens, s'ha realitzat una taula on la sobreexpressió dels gens es troba remarcada amb color vermell (com més fosc, més expressió) i la subexpressió es troba amb color verd (com més fosc, menys expressió).
L'expressió del gen Kua es troba molt difosa entre els diferents teixits. Degut a què encara no es coneix amb precisió la funció d'aquest gen, no podem realitzar una explicació clara que raoni aquest fet.
Expressió ++ Teixit Miel.loide, Cèl.lules del Sistema Immunitari, Pulmons Expressió + Cèl.lules sanguínies, Mèdul.la espinal, Tiroides, Múscul llis, Fetge, Placenta, Testicles Expressió - Estructures Cerebrals, Múscul esquelètic, Ovaris, Apèndix, Òs Expressió -- No detectat
L'expressió del gen UBE2V1, igual que el gen Kua, manté una expressió molt difusa. UBE2V1 és un gen l'expressió del qual és requerida per al procés d'ubiquitinació, procés comú en la majoria de tipus cel.lulars. Aquest fet, concorda amb l'expressió tan extesa que existeix en diferents teixits del nostre organisme.
Expressió ++ Ovaris, Tiroides, Paratiroides Expressió + Testicles, Teixit Miel.loide, Cèl.lules del Sistema Immunitari, Placenta, Múscul llis, Pulmons, Mama, Melsa, Cèl.lules sanguínies, Tràquea, Fetge, Mèdul.la espinal Expressió - Pell, Apèndix, Ovaris, Pròstata, Estómac, Tim, Node Limfàtic, Mèdul.la Adrenal, Pàncrees Expressió -- Estructures cerebrals
Pel que fa a l'expressió del gen Kua-UEV tampoc podem dir que sigui específica d'uns pocs tipus cel.lulars. Tot i així, tots els grups de teixits on la proteïna de fusió es troba s'obreexpressada, tenen també uns nivells superiors de Kua o UBE2V1. El mateix passa amb la seva subexpressió. Tots els teixits on té uns nivells per sota del considerat normal, també es troben subexpressats amb Kua o UBE2V1.
Expressió ++ Pulmons, Testicles, Cèl.lules sanguínies, Cèl.lules del Sistema Immunitari, Teixit Miel.loide Expressió + Mèdul.la espinal, Tim, Cor, Fetge, Tràquea, Tiroides, Pròstata, Placenta, Múscul, Melsa Expressió - Estructures Cerebrals, Amígdala, Pàncrees, Ós Expressió -- Apèndix, Ovaris
Caracterització de la regió promotora del gen
En aquest apartat estudiarem quins factors de transcripció s'uneixen a la seqüència promotora de Kua i quins s'uneixen a UBE2V1. Aquest estudi l'hem realitzat a partir de dos mètodes; mitjançant un programa fet per nosaltres amb llenguatge Perl i a partir de PROMO, un programa de la xarxa. La codificació del programa Perl es pot consultar clicant aquí.
Abans de continuar, és important remarcar que els dos mètodes són molt diferents tot i que tenen la mateixa finalitat. El programa que hem dissenyat és més simple i utilitza unes matrius diferents a les que té PROMO. A més a més, PROMO és força més sofisticat i té en compte molts més factors de transcripció. Per tant, els resultats no seran idèntics.
En la figura 5 es mostren els factors de transcripció que s'uneixen al promotor de Kua i que tenen una puntuació inferior a 0'08 de "RE query". Aquest promotor també és el de Kua-UEV, ja que aquests dos gens es troben a l'strand negatiu del DNA i el primer gen que es transcriu és Kua. Només els factors marcats amb color han estat estudiats amb el nostre programa. Els valors que hi ha a la columna PERL corresponen als p-values obtinguts amb el nostre programa. Quan observem el factor RXR-alpha veiem que s'uneix a dos llocs diferents del promotor però el programa perl només ens mostra el lloc on s'uneix amb més afinitat. Aquest correspon a l'entrada de RXR-alpha amb un valor de RE query més baix (el de dalt).
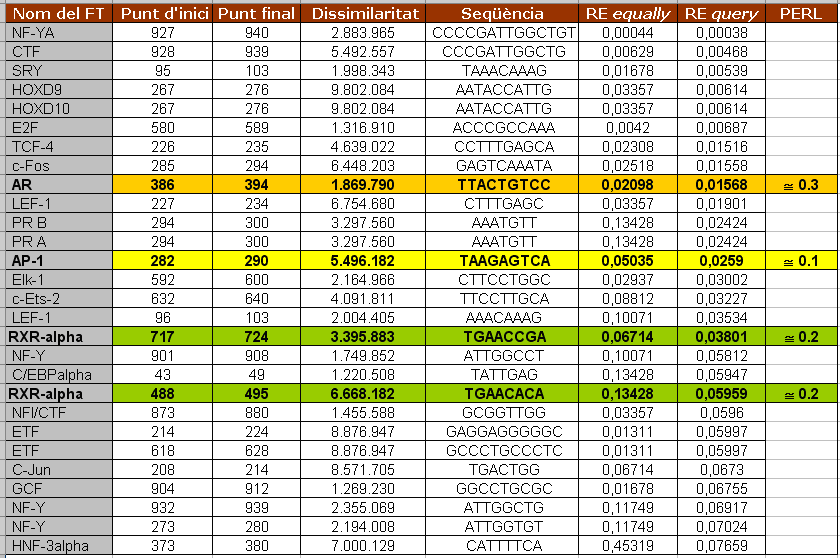
Figura 5. Taula que mostra els factors de transcipció que s'uneixen al promotor de Kua amb un valor de "RE query" inferior a 0'08. També es mostren els valors de p-value obtinguts amb el nostre programa pels facotrs que coincideixen.
Així, hem comprovat que aquests tres factors de transcripció s'uneixin realment al lloc que ens diu el nostre programa. I sí que ho fan. Les posicions, però, no coincideixen exactament, sinó que les del PROMO són més àmplies. Això és degut simplement a que el PROMO utilitza unes matrius més llargues que les que hem utilitzat nosaltres. Creiem que els valors que RE query i p-value obtinguts amb PROMO i amb el nostre programa, respectivament, són coherents. Un p-value baix (o relativament baix), com obtenim per a aquests tres factors, és indicatiu de que és complicat d'obtenir el mateix resultat per atzar. D'altra banda, un RE query tan baix també ens indica que hi ha poca probabilitat d'obtenir el resultat per atzar i que, per tant, aquest té una alta significància estadística.
Passem ara a comentar els resultats obtinguts per al promotor de UBE2V1. Aquests es poden observar a la figura 6. En aquest cas hem sintetitzat la informació en la figura. Simplement hi hem posat aquells factors de transcripció que també havien estat estudiats amb el nostre programa i, únicament les entrades que coincideixen en la posició. Creiem que els resultats tornen a ser coherents i significatius, pel raonament que hem explicat anteriorment. Tot i així, és important comentar que el factor AR obtingut amb PROMO no s'uneix a la mateixa posició que el del nostre programa. Però si ens fixem en el p-value veiem que aquest és força elevat (0'5) i que per tant, no és complicat obtenir aquest resultat a l'atzar. Per això no ens refiaríem d'aquest resultat.

Figura 6. Taula que mostra només els factors de transcipció que s'uneixen al promotor de UBE2V1 amb un valor de "RE query" inferior a 0'08 i que coincideixen també han estat obtinguts amb el nostre programa.
Pel que fa als resultats obtinguts amb el nostre programa Perl, partiem d'un nombre limitat de factors de transcripció a estudiar. Per això, molts factors de transcripció amb un "RE query" en el programa PROMO no han estat pogut contrastats amb els resultats del nostre programa. Cal destacar que el valor del p-value és una aproximació, ja que es calcula a partir de la nostre seqüència promotora reorganitzada a l'atzar.
Aquesta primera taula mostra els resultats del nostre progrma realitzats a partir de la seqüència promotora del gen Kua, que és el mateix que el de la proteïna de fusió Kua-UEV.
| Factor de transcripció | Nucleòtids on s'uneix | Score | p-value | AP-1 | 285-291 | 3,3010 | 0,08 |
| AR | 408-414 | 2,9311 | 0,33 |
| c-Myc | 324-329 | -996,0320 | 0,97 |
| NF-AT1 | 265-271 | -996,0786 | 0,76 |
| NF-KappaB | 672-680 | -995,0872 | 0,32 |
| SRF | 40-48 | -995,0784 | 0,14 |
| YY1 | 1087-1092 | 2,8403 | 0,26 |
| RXR-alpha | 718-723 | 3,1882 | 0,18 |
| HIF-1 | 320-328 | -995,8374 | 0,72 |
| AhR | 1009-1015 | 3,1396 | 0,21 |
| PU.1 | 487-493 | -996,7950 | 0,6 |
| HNF-4 | 686-693 | -996,2413 | 0,98 |
| NRSF | 319-327 | 4,1054 | 0,01 |
La següent taula mostra els resultats obtinguts a partir de la seqüència promotora del gen UBE2V1 mitjançant el nostre programa Perl.
| Factor de transcripció | Nucleòtids on s'uneix | Score | p-value | AP-1 | 114-120 | -995,5520 | 0,72 |
| AR | 547-553 | 2,8190 | 0,38 |
| c-Myc | 201-206 | -995,9544 | 0,82 |
| NF-AT1 | 595-601 | 3,3859 | 0,23 |
| NF-KappaB | 764-772 | 2,3087 | 0,31 |
| SRF | 705-713 | -994,6389 | 0,11 |
| YY1 | 330-335 | 3,0296 | 0,18 |
| RXR-alpha | 249-254 | 3,0608 | 0,21 |
| HIF-1 | 928-936 | -995,6685 | 0,57 |
| AhR | 920-926 | 3,2831 | 0,18 |
| PU.1 | 891-897 | 2.5936 | 0,32 |
| HNF-4 | 446-453 | -995,5672 | 0,47 |
| NRSF | 436-444 | -995,9644 | 0,46 |
Estudi de la funció del gen
Les proteines UEV es poden classificar en en dos tipus, que són les UEV1 (o UBE2V1) i les UEV2 (o UBE2V2, del cromosoma 8). Les primeres, expressades per al gen UEV1 són les úniques que comentarem ja que la proteïna de fusió que estudiem s'expressa únicament a partir d'aquest tipus.
Arribat a aquest punt creiem convenient fer una breu introducció del sistema d'ubiquitinació, ja que la nostra proteïna en forma part. Aquest sistema consisteix en un complex enzimàtic on E2 (prèviament conjugat al residu conservat de cisteïna Ub) catalitza la transferència d'Ub als residus de lisina de les proteïnes diana. Es va descobrir una subfamília d'E2, la de UEV, la qual no posseïa aquest residu de cisteïna. Per aquesta causa, són proteïnes inactives i van ser designades com a variants inactives dels enzims conjugats a ubiquitines.
Les UEV1 estan localitzades al nucli (veure figura 10) i són variants inactives dels enzims conjugats a ubiquitines E2. Concretament, estan considerades com a una subfamília d'aquestes últimes. Això ho demostren diversos estudis d'homologia sobre aquesta família. En aquesta imatge s'observa un arbre filogenètic dels enzims conjugats a Ubiquitines. Les proteïnes UEV1 regulen l'elongació no canònica de les cadenes d'ubiquitines unides per a la lisina de la posicó 63. La seva falta d'activitat catalítica és deguda a la pèrdua d'un residu de cisteïna. Per a la seva activació requereixen de la unió amb UEV2 formant heterodímers. Aquesta funció ve determinada per al seu únic domini, que s'observa a la figura 7. A més a més, protegeixen a la cèl.lula d'agents perjudicials pel DNA.

Figura 7. Es mostren els dominis de la proteïna UEV.
També s'ha estudiat la seva expressió diferencial en cèl.lules en proliferació i en diferenciació així com en teixits tumorals. Aquests estudis suggereixen que les proteïnes UEV1 poden actuar com a reguladores de la proliferació i diferenciació cel.lular. Per a aquest fet, existeixen altres estudis on es demostra que poden causar càncer de colon en humans degut a l'activació de l'oncogen FOS que provoquen.
D'altra banda, no s'ha acabat de determinar la funció de Kua. El que sí que està clar és que Kua aporta el domini transmembrana a la proteïna híbrida i això determina la localització d'aquesta al citoplasma. S'ha de dir que Kua també es pot trobar associada al reticle endoplasmàtic (tot aixó s'observa a la figura 10). Aquest és l'únic domini que té aquesta proteïna i s'observa a la figura 8.
Figura 8. Es mostren els dominis de la proteïna híbrida.
Per últim, Kua-UEV genera dues isoformes (comentades a l'apartat de l'expressió gènica) que mantenen la funció UEV. Les Kua-UEV, com s'observa a la figura 9, són proteïnes de dos dominis; un l'aporta UEV i és el que dóna la funció i l'altre és el de Kua, que com ja hem comentat, simplement aporta la seva localització final. En la figura 10 es pot comprovar com la localització de UEV1 és nuclear (A) mentre que la de Kua (B) i Kua-UEV (C) són citoplasmàtiques. Recordem que Kua també es pot trobar associada al reticle endoplasmàtic i per això podem observar la seva presència més difusa en B.

Figura 9. Es mostren els dominis de la proteïna Kua.
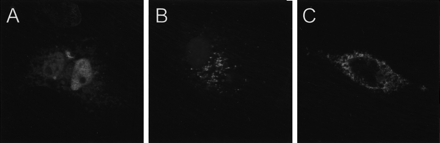
Figura 10. Imatge de les localitzacions cel.lulars de UEV1 (A), Kua (B) i Kua-UEV (C) a partir d'immunofluorescència indirecta amb microscopia confocal.
Fins aquí s'ha comentat la funció dels gens trobada a partir de la literatura científica. A continuació es mostren els resultats trobats mitjançant 'Gene Onthology', que ajuda a entendre la resta de l'informació.
Funció Molecular de UBE2V1:
Procés Biològic on està involucrat UBE2V1:
Funció Molecular de Kua-UEV:
Procés Biològic on està involucrat UBE2V1:
Procés Biològic on està involucrat Kua-UEV:
Mètodes
En aquest punt comentarem com hem pogut realitzar els diferents apartats del treball així com la manera en que l'hem estructurat.
Primer de tot, hem ajuntat els apartats de resultats i discussió, ja que considerem que és més fàcil assimilar la informació després del seu resultat corresponent. Tot i així, hem cregut oportú de posar un apartat de conclusions per a sintetitzar tota la informació obtinguda i "hipòtesis" fetes.
Passant ja als diferents apartats en sí, la informació referent a l'estructura genòmica l'hem obtingut bàsicament del UCSC i del NCBI. De l'Ensembl quasi no n'hem extret ja que, tot i que hi ha informació sobre les dues proteïnes, aquesta està estructurada de forma que tan els dos gens com la proteïna de fusió són transcripts de d'una sola proteïna; de la UBE2V1. De totes maneres, d'aquí és d'on hem extret la informació referent a les parts codificants i no codoficants així com algunes de les imatges. A més a més, ens ha sigut de molta utilitat poder fer alguns aliniaments amb el ClustalW per visualitzar millor les diferències entre els diferents transcrits. Pel que fa a la predicció de gens, l'hem feta amb tots els programes que ens van explicar a les sessions pràctiques per a poder comparar-los. Aquests són el Geneid, el Genscan, el Fgenesh i el Grail. L'únic que s'ajustava bé a la resta d'informació, però, era el Geneid, per la qual cosa només hem adjuntat els resultats d'aquest programa.
Per a l'estudi de la homologia, en canvi, hem extret els percentatges d'homologia del NCBI, de l'apartat de l'HomoloGene. Per a fer la informació més visual i acabar d'aclarir alguns conceptes, però, vam agafar les seqüències de DNA dels animals dels quals havíem trobat homologia i en vam fer una aliniament amb el programa ClustalW per a construir-ne els respectius arbres filogenètics.
A continuació, vam fer la caracterització de l'expressió del gen. I sobre aquest punt ens hem basat en la informació del UCSC; concretament a l'apartat dels microarrays ("microarray expression data").
Els factors de transcripció han estat estudiats amb dos mètodes diferents. Amb el programa Perl dissenyat per a nosaltres mateixes i amb el PROMO. Posteriorment, hem comprovat que els factors de trancripció comuns tinguessin coherència.
I, per últim, la funció del gen, que ha estat extreta bàsicament de la literatura científica però amb el Gene Ontology ens vam poder fer un esquema general. Això ha estat complementat amb una búsqueda dels dominis de les diverses proteïnes, realitzat amb l'Interpro.
Torna a l'índexConclusions
Després d'haver realitzat l'estudi genòmic computacional de la proteïna Kua-UEV, podem concloure que aquesta és fruit de la transcripció contínua de dos gens veïns del cromosoma 20. Aquests dos gens són Kua i UBE2V1 (també anomenat com a UEV1).
El gen UBE2V1 pertany a una subfamília de les proteïnes E2 que es va separar d'aquestes en un moment determinat de la història evolutiva. A partir d'aquest punt cada espècie va evolucionar separadament però existeix que hi ha una gran conservació entre elles. Aquest fet és degut a la implicació d'aquest gen en processos cel.lulars bàsics i fonamentals. Pel que fa al gen Kua s'ha descobert més recentment i no està tan ben estudiat. Tot i això, té una alta homologia entre les espècies estudiades.
Els dos gens estudiats estan situats a l'strand negatiu de la cadena de DNA de la banda q13.13 del cromosoma 20 on Kua es troba 'upstream' de UBE2V1. Kua codifica per a un sol transcrit de sis exons mentre que UBE2V1 té quatre transcrits diferents que dónen lloc a quatre isoformes. Tot i així, aquestes diferències recauen en els exons que no codifiquen per a la proteïna híbrida. Per la qual cosa, és indiferent quin transcrit es transcrigui; la nostra proteïna té la part comuna a tots els transcrits. Ocasionalment, es dóna la co-transcripció d'ambdós gens tot i que es poden transcriure independentment. Com que els gens estan a l'strand negatiu del DNA, la transcripció començarà per Kua. Aquesta co-transcripció es dóna a causa de splicing alternatiu i pot donar lloc a dues isoformes (una de 221 aminoàcids i l'altra de 370) diferents de la proteïna híbrida. Cal remarcar que la diferència en les dues isoformes es basa en la diferencial transcripció del gen Kua. La isoforma llarga, que és la més freqüent, consta dels cinc primers exons de Kua i dels tres últims de qualsevol dels quatre transcrits de UBE2V1. D'altra banda, la isoforma curta està formada pels exons 4 i 5 de Kua i, altra vegada, pels tres últims de UBE2V1.
Pel que fa a l'expressió d'aquests gens, podem concloure que els dos es troben en una gran quantitat de teixits. Pel que fa a Kua, no hem acabat de trobar-hi una expicació plausible ja que la seva funció no està del tot determinada. En canvi, per a UBE2V1, hipotetitzem que es molt lògic que estigui en aquesta gran quantitat de teixits a causa de la seva funció tan bàsica i essencial. Finalment, per a Kua-UEV, podem observar que la seva expressió (tan la sobre com la subexpressió) coincideix amb les dels dos altres gens.
Sobre l'apartat de la caracterització de la regió promotora, podem concloure que els resultats de PROMO són més fiables que els obtinguts per al nostre programa, ja que estudia un nombre limitat de factors i no fa servir un mètode tan acurat. Per això, els factors de transcripció amb més probabilitat d'unir-se a la regió promotora de la nostra proteïna de fusió (que serà la de Kua, ja que la transcripció comença per a aquest gen) són els que es mostren a la figura 5. De totes maneres, els únics factors coincidents per als dos mètodes són AR, AP-1 i RXR-alpha.
Per últim, farem referència a la funció. Com hem anat repetint a la resta del treball, la funció de Kua no està determinada. Així, només en podem concloure que aporta a la proteïna de fusió el domini transmembrana que determinarà la seva localització cel·lular (citoplasmàtica) una vegada traduïda. En canvi, UBE2V1 està implicada en molts processos bàsics i fonamentals, a causa del domini que té conjugat a ubiquitines. Aquests processos són, bàsicament, l'elongació no-canònica de les cadenes d'ubiquitines i la regulació de la proliferació i la diferenciació cel.lular.
Torna a l'índex
Referències
Bibliografia:
Syed NA, Andersen PL, Warrington RC, Xiao W. Uev1A, a ubiquitin conjugating enzyme variant, inhibits stress-induced apoptosis through NF-kappaB activation. 2006 Dec;11(12):2147-57. PMID: 17041755.
Thomson TM, Lozano JJ, Loukili N, Carrio R, Serras F, Cormand B, Valeri M, Diaz VM, Abril J, Burset M, Merino J, Macaya A, Corominas M, Guigo R. Fusion of the human gene for the polyubiquitination coeffector UEV1 with Kua, a newly identified gene. 2000 Nov;10(11):1743-56. PMID: 11076860.
Thomson TM, Khalid H, Lozano JJ, Sancho E, Arino J. Role of UEV-1A, a homologue of the tumor suppressor protein TSG101, in protection from DNA damage. 1998 Feb 13;423(1):49-52. PMID: 9580084.
Rothofsky ML, Lin SL. CROC-1 encodes a protein which mediates transcriptional activation of the human FOS promoter. 1997 Aug 22;195(2):141-9. PMID: 9305758.
Programes:
UCSC: http://genome.ucsc.edu
NCBI: http://www.ncbi.nlm.nih.gov
Ensembl: http://www.ensembl.org/index.html
ClustalW: http://www.ebi.ac.uk/clustalw
Geneid: http://www1.imim.es/software/geneid/geneid.html
Genscan: http://genes.mit.edu/GENSCAN.html
Fgenesh: http://www.softberry.com/berry.phtml
Grail: http://grail.lsd.ornl.gov/grailexp
PROMO: http://alggen.lsi.upc.es
Gene Ontology: http://www.geneontology.org
InterPro: http://www.ebi.ac.uk/InterProScan/
Torna a l'índex