En aquest projecte es realitza un estudi complet del gen quimèric característic de Wld(s), així com del dos gens originals: Ube4b i Nmnat1. Es caracteritza l'estructura genòmica d'aquests, així com l'homologia present en altres espècies, l'estudi del seu patró d'expressió i de la seva regió promotora. Finalment, es fa una revisió en profunditat de la funció de cada gen.
Resultats i Discussions |
La proteïna de fusió Ube4b/Nmnat1 està codificada per l'assemblatge de dues seqüències dels gens Ube4b i Nmnat1. Tots dos gens es troben en la regió qE2 del cromosoma 4 de Mus musculus, separats per una distància aproximada de 70000 pb. Ambdós tenen la mateixa orientació cromosòmica, s'expressen en reverse.
Per tal de caracteritzar l'estructura genòmica de la proteïna de fusió, cal abans un anàlisi acurat dels dos gens originals.
Ube4b
- GENE ID: ENSMUSG00000028960, NM_022022 , MGI:1927086
- Sinònims: 4930551I19Rik, 4933406G05Rik, D4Bwg0973e, mKIAA0684, UFD2, UFD2a.
- Posició: Cromosoma 4 regió qE2: 148,172,216-148,270,549 (reverse strand).
- DNA genòmic en FASTA
- Splicing: Alternatiu.
- Nº de transcrits: 2.
| TRANSCRIT 1: ENSMUST00000084123 | |
|---|---|
|
|
| TRANSCRIT 2: ENSMUST00000103212 | |
|---|---|
|
|
- Nº d' isoformes: Els dos transcrits donen lloc a dues isoformes diferents. En la traducció dels transcrits es segueix el mateix marc de lectura, de manera que la diferència entre les isoformes és tan sols pel que fa la seva longitud.
[Veure alineament de les isoformes]
| ISOFORMA 1: ENSMUSP00000081140 | |
|---|---|
|
VHPLSADEIRRRRLARLAGGQTSQPTTPLTSPQRENPPGPPIAASAPGPSQSLGLNVHNMTPATSPIGAAGVAHRSQSSEGVSSLSSSPSNSLETQSQSL
SRSQSMDIDGVSCEKSMSQVDVDSGIENMEVDENDRREKRSLSDKEPSSGPEVSEEQALQLVCKIFRVSWKDRDRDVIFLSSLSAQFKQNPKEVFSDFKD
LIGQILMEVLMMSTQTRDENPFASLTATSQPIATAARSPDRNLMLNTGSSSGTSPMFCNMGSFSTSSLSSLGASGGASNWDSYSDHFTIETCKETDMLNY
LIECFDRVGIEEKKAPKMCSQPAVSQLLSNIRSQCISHTALVLQGSLTQPRSLQQPSFLVPYMLCRNLPYGFIQELVRTTHQDEEVFKQIFIPILQGLAL
AAKECSLESDYFKYPLMALGELCETKFGKTHPMCNLVASLPLWLPKSLSPGSGRELQRLSYLGAFFSFSVFAEDDAKVVEKYFSGPAITLENTRVVSQSL
QHYLELGRQELFKILHSILLNGETREAALSYMAALVNANMKKAQMQADDRLVSTDGFMLNLLWVLQQLSTKIKLETVDPTYIFHPRCRITLPNDETRINA
TMEDVNERLTELYGDQPPFSEPKFPTECFFLTLHAHHLSILPSCRRYIRRLRAIRELNRTVEDLKNNESQWKDSPLATRHREMLKRCKTQLKKLVRCKAC
ADAGLLDESFLRRCLNFYGLLIQLMLRILDPAYPDVTLPLNSEVPKVFAALPEFYVEDVAEFLFFIVQYSPQVLYEPCTQDIVMFLVVMLCNQNYIRNPY
LVAKLVEVMFMTNPSVQPRTQKFFEMIENHPLSTKLLVPSLMKFYTDVEHTGATSEFYDKFTIRYHISTIFKSLWQNIAHHGTFMEEFNSGKQFVRYINM
LINDTTFLLDESLESLKRIHEVQEEMKNKEQWDQLPRDQQQARQSQLAQDERVSRSYLALATETVDMFHLLTKQVQKPFLRPELGPRLAAMLNFNLQQLC
GPKCRDLKVENPEKYGFEPKKLLDQLTDIYLQLDCARFAKAIADDQRSYSKELFEEVISKMRKAGIKSTIAIEKFKLLAEKVEEIVAKNARAEIDYSDAP
DEFRGNNPLMDTLMTDPVRLPSGTVMDRSIILRHLLNSPTDPFNRQMLTESMLEPVPELKEQIQAWMREKQSSDH
|
| ISOFORMA 2: ENSMUSP00000099501 | |
|---|---|
|
MEELSADEIRRRRLARLAGGQTSQPTTPLTSPQRENPPGPPIAASAPGPSQSLGLNVHNMTPATSPIGAAGVAHRSQSSEGVSSLSSSPSNSLETQSQSL
SRSQSMDIDGVSCEKSMSQVDVDSGIENMEVDENDRREKRSLSDKEPSSGPEVSEEQALQLVCKIFRVSWKDRDRDVIFLSSLSAQFKQNPKEVFSDFKD
LIGQILMEVLMMSTQTRDENPFASLTATSQPIATAARSPDRNLMLNTGSSSGTSPMFCNMGSFSTSSLSSLGASGGASNWDSYSDHFTIETCKETDMLNY
LIECFDRVGIEEKKAPKMCSQPAVSQLLSNIRSQCISHTALVLQGSLTQPRSLQQPSFLVPYMLCRNLPYGFIQELVRTTHQDEEVFKQIFIPILQGLAL
AAKECSLESDYFKYPLMALGELCETKFGKTHPMCNLVASLPLWLPKSLSPGSGRELQRLSYLGAFFSFSVFAEDDAKVVEKYFSGPAITLENTRVVSQSL
QHYLELGRQELFKILHSILLNGETREAALSYMAALVNANMKKAQMQADDRLVSTDGFMLNLLWVLQQLSTKIKLETVDPTYIFHPRCRITLPNDETRINA
TMEDVNERLTELYGDQPPFSEPKFPTECFFLTLHAHHLSILPSCRRYIRRLRAIRELNRTVEDLKNNESQWKDSPLATRHREMLKRCKTQLKKLVRCKAC
ADAGLLDESFLRRCLNFYGLLIQLMLRILDPAYPDVTLPLNSEVPKVFAALPEFYVEDVAEFLFFIVQYSPQVLYEPCTQDIVMFLVVMLCNQNYIRNPY
LVAKLVEVMFMTNPSVQPRTQKFFEMIENHPLSTKLLVPSLMKFYTDVEHTGATSEFYDKFTIRYHISTIFKSLWQNIAHHGTFMEEFNSGKQFVRYINM
LINDTTFLLDESLESLKRIHEVQEEMKNKEQWDQLPRDQQQARQSQLAQDERVSRSYLALATETVDMFHLLTKQVQKPFLRPELGPRLAAMLNFNLQQLC
GPKCRDLKVENPEKYGFEPKKLLDQLTDIYLQLDCARFAKAIADDQRSYSKELFEEVISKMRKAGIKSTIAIEKFKLLAEKVEEIVAKNARAEIDYSDAP
DEFRDPLMDTLMTDPVRLPSGTVMDRSIILRHLLNSPTDPFNRQMLTESMLEPVPELKEQIQAWMREKQSSDH
|
Nmnat1
- GENE ID: ENSMUSG00000028992, NM_133435, MGI: 1913704
- Sinònims: 2610529L11Rik, 5730441G13Rik, D4Cole1e, nicotinamide mononucleotide adenylyl transferase, nmnat.
- Posició: Cromosoma 4 regió qE2: 148,312,409-148,329,096 cadena reversa.
- DNA genòmic en FASTA
- Splicing: Constitutiu.
- Nº de transcrits: 1
| TRANSCRIT 1: ENSMUST00000030845 | |
|---|---|
|
|
- Nº d' isoformes: 1
| ISOFORMA 1: ENSMUSP00000030845 | |
|---|---|
|
MDSSKKTEVVLLACGSFNPITNMHLRLFELAKDYMHATGKYSVIKGIISPVGDAYKKKGLIPAHHRIIMAELATKNSHWVEVDTWESLQKEWVETVKVLR
YHQEKLATGSCSYPQSSPALEKPGRKRKWADQKQDSSPQKPQEPKPTGVPKVKLLCGADLLESFSVPNLWKMEDITQIVANFGLICITRAGSDAQKFIYE
SDVLWRHQSNIHLVNEWITNDISSTKIRRALRRGQSIRYLVPDLVQEYIEKHELYNTESEGRNAGVTLAPLQRNAAEAKHNHSTL
|
Gen quimèric que codifica per Ube4b/Nmnat1
- GENE ID: No es troba identificat com a gen, sinó com a aproximació de la unitat triplicada en tandem [AF260927].
- Sinònims: UFD2/D4COLE1E
- Localització: Cromosoma 4 regió qE2.
El gen quimèric és característic de la soca C57BL/Wld(s). Aquest es forma a partir de la concatenació de les seqüències proximals i distals d'unitats de repetició contigües.
Pel que fa a la transcripció i processament del gen quimèric, només s'ha observat la producció d'un transcrit que segueix el model del transcrit 2 d'Ube4b i l'splicing constitutiu de Nmnat1.
El mRNA obtingut té 6 exons [AF260924]:
- Exó 1 - exó 2: corresponen a una fracció de l'exó 1 i la totalitat de l'exó 2 del transcrit 2 d'Ube4b.
- Exó 3 - exó 6: corresponen als exons 2-5 de Nmnat1.
Les regions codificants del transcrit quimèric són les mateixes que en els transcrits corresponents d'Ube4b i de Nmnat (soca wt), excepte un cas: l'exó 2 de Nmnat es tradueix totalment en el trancrit quimèric, a diferència del transcrit wt en el qual els 55 primers nucleòtids són no codificants. En tot cas, aquesta modificació no fa variar el marc de lectura de la seqüència downstream d'aquesta modificació.
Per un major aclariment, veure seqüències il·lustrades.
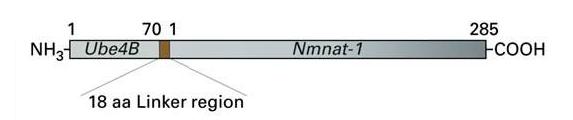
La proteïna quimèrica resultant té 372 residus [AAG17285]:
- 70 aminoàcids de l'extrem N-terminal d'Ube4b
- 302 aminoàcids de Nmnat1; dels quals:
-
- Els primers 18 aminoàcids corresponen a la part no codificant de l'exó 2 de la Nmnat1 wt. A vegades és citada com a seqüència linker entre els dos gens [11].
- Els 285 aminoàcids darrers corresponen a la totalitat de la seqüència proteica de Nmnat1 wt.
Font: Fainzilber M, Twiss JL. Tracking in the Wld(s)--the hunting of the SIRT and the luring of the Draper. Neuron. 2006 Jun 15;50(6):819-21.[11]
La proteïna Ube4b/Nmnat1 té un pes molecular aproximat de 43 KDa [9]. La proteïna de fusió conté el domini característic de Nmnat1, per contra, no conté el domini U-box d'Ube4b. És a dir, que presenta activitat de nicotinamida transferasa, però no pot unir-se a ubiquitines. Nogensmenys, Conforti L et al. (2000) [9] hipotetitzen que els 70 aminoàcids N-terminals d'Ube4b presents en la proteïna de fusió podrien contenir dominis d'interacció amb substrats proteics, relacionant-se amb l'alteració de les rutes d'ubiquitinació. Així, en cas d'expressar-se la proteïna de fusió, cursaria reclutant possibles substrats per a multiubiquitinació que finalment no es multiubiquitinarien per falta del domini U-box en la proteïna de fusió.
Pel que fa a la seqüecia linker de 18 aminoàcids cal dir que fins ara no ha estat associada a cap domini proteic. La seva funció està discutida en l'apartat 5: Estudi de la funció del gen quimèric.
Estudi de la homologia del gen en altres espècies La proteïna de fusió Ube4b/Nmnat1 és exclusiva de la soca de ratolí C57BL/Wld(s) i per això mateix no es troba cap ortòleg de la seva seqüència completa. Per estudiar l'homologia dels gens, ens basem en l'anàlisi per separat dels dos gens que formen el gen quimèric en els ratolins C57BL/Wld(s).
Ube4b
Per altra banda, els resultats que considerem realment informatius són els ortòlegs de Gallus gallus i de Xenopus tropicalis, ja que tot i tractar-se d'espècies llunyanes (pertanyen a classes diferents) tenen respectivament, un 91 i un 85% d'homologia. Amb aquesta observació deduim que és un gen, molt conservat. Per tant, la seva funció és molt important i l'estructura molecular de la proteïna per la que codifica no admet gaires canvis que impliquin alteracions de la seva funció.
Un estudi detallat de l'homologia en les seqüències dels gens ortòlegs mostra que la similaritat és destacadament alta en la regió dels ~100 aminoàcids C-terminal, allà on trobem el domini U-box (UFD2-homology domain) del que depèn la funció, de la proteïna. A més a més, es troben flanquejats per seqüències que sovint representen potencials dominis d'interacció proteïna-proteïna [20].
Nmnat1
Es coneixen 25 gens ortòlegs al gen de Nmnat1 de Mus musculus. Tan sols 8 d'aquests tenen un percentatge d'homologia superior al 70% i un únic ortòleg, superior al 85%. Tots els ortòlegs dels quals es coneix la funció, codifiquen per la Nmnat pròpia de cada espècie. El màxim percentatge d'homologia trobat és del 92% i correspon al gen ortòleg de R. novergicus. Després d'aquest, hi ha una sèrie d'espècies amb una homologia entre 77 i 81% entre les quals trobem només mamífers de diferents ordres. Els ortòlegs d'espècies de diferent classe taxonòmica, és a dir, que no són mamífers, tots tenen percentatges d'homologia inferiors al 62%. Entre aquests trobem sobretot peixos, amfibis, insectes, entre d'altres. D'aquestes observacions deduim que el gen de Nmnat no està especialment conservat en el total d'espècies, més aviat presenta força variació entre ortòlegs. En tot cas, cal destacar que mostra una conservació molt menor que Ube4b.L'homòleg de Nmnat1 en humans fou descrit i mapat al cromosoma 1 regió p36.2 per Fernando FS et al. (2002) [13]. El cDNA humà que codifica per una seqüència aminoacídica de 279 residus, té un 80% d'homologia amb l'ortòleg de ratolí. Demostraren que el cDNA humà i la seqüència proteica són idèntiques a la nicotinamida mononucleòtid adeniltransferasa (Nmnat). L'objectiu del projecte és facilitar l'screening dels exons d'aquest gen humà en pacients amb malalties neurodegeneratives.
Ube4b
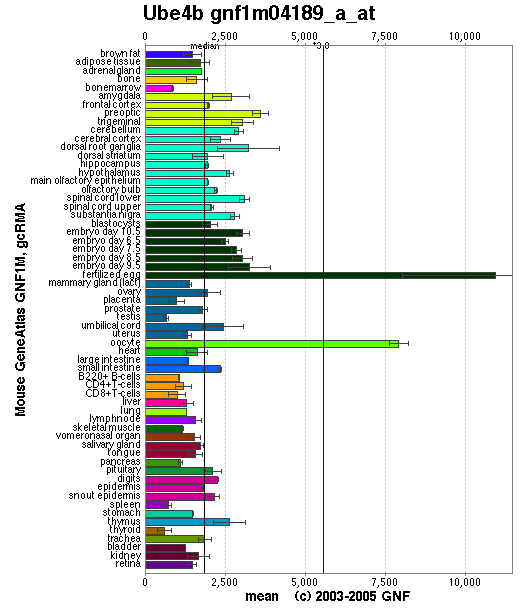 En l'estudi de l'expressió del gen d'Ube4b de Mus musculus observem que hi ha notables diferències entre els teixits dels diversos òrgans i sistemes.
En l'estudi de l'expressió del gen d'Ube4b de Mus musculus observem que hi ha notables diferències entre els teixits dels diversos òrgans i sistemes.Així en primer lloc, observem que en l'animal adult, els teixits del sistema nerviós presenten una magnitud d'expressió superior a la mitjana d'expressió global. De mitjana, entre tots ells, presenten unes 600-700 unitats d'expressió per sobre la mitjana que correspon aproximadament a 1800-1900 unitats d'expressió segons GNF Atlas 2 (2003-2005). Entre tots els teixits del sistema nerviós, els que més destaquen, indicats per ordre decreixen, són el gangli de l'arrel dorsa, el nucli preòptic, l'amígdala, el trigemin, el cerebel i la part d'adat de la medul·la espinal. De totes maneres, l'òrgan que presenta un nivell d'expressió major en els ratolins adults són els oòcits de les femelles, on l'expressió sobrepassa les 7500 unitats d'expressió.
En segon lloc, pel que fa a l'expressió del gen en el desenvolupament embrionari del ratolí, s'observa que aquesta assoleix els nivells més elevats (inclús majors que els màxims en edat adulta) en el primer moment just després de la fecundació de l'oòcit, on assoleix les 11000 unitats d'expressió; seguidament, la seva expressió decreix gradualment amb l'avenç del desenvolupament de l'embrió, mantenint sempre nivells d'expressió de 600-700 unitats, aproximadament, per sobre la mitjana global del gen.
Considerant la funció del gen, aquests resultats són força lògics. Ube4b forma part de la ruta d'ubiquitinació de proteïnes. Donat que aquesta és una de les rutes més important de control de la síntesi proteica, concretament en la ruta principal de degradació de proteïnes mal sintetitzades o mal plegades, és lògic pensar que estarà sobreexpressat en aquells moments en què la síntesi proteica és màxima, com és el cas de les primeres fases i en general de tot el desenvolupament embrionari. Pel que fa als teixits del sistema nerviós, s'observen alts nivells d'expressió d'aquest gen. Es pot hipotetitzar que això és causat perquè es tracta d'un sistema molt delicat, en el qual la mida de les cèl·lules, així com la longitud dels axons està en constant modificació i adaptació; per tant, cal que la les rutes de control de la síntesi proteica estiguin fortament expressades.
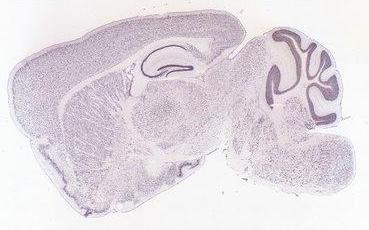 Finalment, adjuntem a continuació una imatge trobada en la base de dades VisiGene en la que es pot apreciar l'elevat índex d'expressió d'Ube4b en el teixit nerviós. Es tracta d'un tall sagital d'encèfal de ratolí mascle de la soca C57BL/6 wt de 5,3 setmanes de vida.
Finalment, adjuntem a continuació una imatge trobada en la base de dades VisiGene en la que es pot apreciar l'elevat índex d'expressió d'Ube4b en el teixit nerviós. Es tracta d'un tall sagital d'encèfal de ratolí mascle de la soca C57BL/6 wt de 5,3 setmanes de vida.
La imatge és original de l'Allen Brain Atlas (ABA), 2006.
Copyright: ©2004-2006 Allen Institute for Brain Science.
Nmnat1
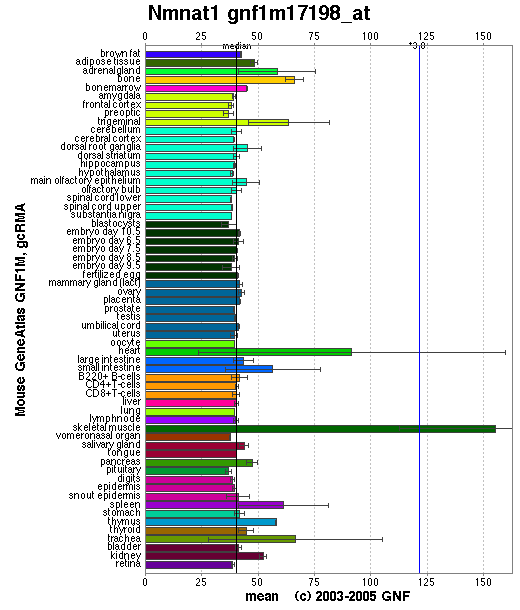 En l'estudi de l'expressió del gen Nmnat1 s'han observat una tendència global molt més unificada entre els diferents teixits, òrgans i sistemes, per comparació amb Ube4b en què s'observaren nivells d'expressió molt diferents entre els teixits. Per a Nmnat1, la majoria dels teixits presenten nivells d'expressió molt ajustats a la mitjana global d'expressió, que en aquest cas ronda les 35-40 unitats d'expressió. Tot i així, trobem alguns casos en què l'expressió assoleix nivells molt superiors a la mitja.
En l'estudi de l'expressió del gen Nmnat1 s'han observat una tendència global molt més unificada entre els diferents teixits, òrgans i sistemes, per comparació amb Ube4b en què s'observaren nivells d'expressió molt diferents entre els teixits. Per a Nmnat1, la majoria dels teixits presenten nivells d'expressió molt ajustats a la mitjana global d'expressió, que en aquest cas ronda les 35-40 unitats d'expressió. Tot i així, trobem alguns casos en què l'expressió assoleix nivells molt superiors a la mitja.Els teixits que més destaquen són el múscul esquelètic, amb més de 150 unitats d'expressió (més del triple de l'expressió mitja), i el teixit cardíac, amb gairebé 90 unitats d'expressió (més del doble de l'expressió mitja).
Seguidament, trobem una sèrie de teixits en què també trobem nivells d'expressió superiors a la mitja (en unes 25-30 unitats), es tracta de la tràquea, el trigèmin i l'òs. Finalment, la glàndula adrenal, l'intestí prim, la melsa i el timus, entre d'altres mostren un nivell d'expressió lleugerament superior, aproximadament en unes 20 unitats major que la mitja.
El gen de Nmnat codifica per una nucleotinamida transferasa, que participa en la síntesi dels cofactors NAD+ i NADH, entre d'altres; esperaríem trobar-ne una elevada expressió en aquells teixits més actius, que tinguessin una taxa metabòlica més important degut a la seva activitat. Els resultats obtinguts coincideixen amb la nostra hipòtesi inicial, ja que, com ja s'ha comentat, els teixits que presenten major expressió són el múscul esquelètic i el teixit cardíac.
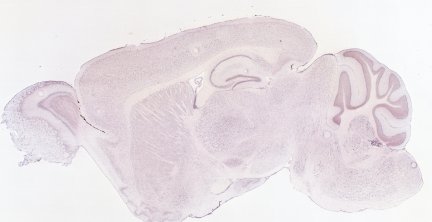
A continuació, adjuntem una imatge trobada en la base de dades VisiGene que demostra l'expressió de Nmnat1 en el teixit nerviós. Es tracta d'un tall sagital d'encèfal de ratolí mascle de la soca C57BL/6 wt de 5,3 setmanes de vida.
La imatge és original de l'Allen Brain Atlas (ABA), 2006.
Copyright: ©2004-2006 Allen Institute for Brain Science.
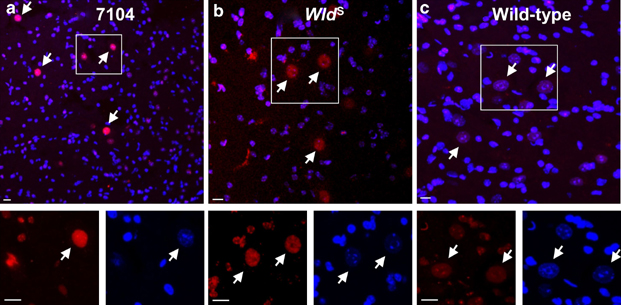 L'estudi de l'expressió de Nmnat1 en teixits neuronals de ratolins Wld(s) és un objectiu freqüent en les investigacions relacionades amb el progrés de la malaltia. Adjuntem a continuació una imatge de l'article de Conforti L et al. (2007) [8] que mostra per tinció inmunohistoquímica les altes concentracions nuclears de Nmnat1 en neurones de la medul·la espinal lumbar en ratolins wt i Wld(s).
L'estudi de l'expressió de Nmnat1 en teixits neuronals de ratolins Wld(s) és un objectiu freqüent en les investigacions relacionades amb el progrés de la malaltia. Adjuntem a continuació una imatge de l'article de Conforti L et al. (2007) [8] que mostra per tinció inmunohistoquímica les altes concentracions nuclears de Nmnat1 en neurones de la medul·la espinal lumbar en ratolins wt i Wld(s).
Gen quimèric Ube4b/Nmnat1
 Degut a la naturalesa de les triplicacions Wld(s), l'expressió del transcrit quimèric que codifica per la proteïna de fusió es troba sota el control del promotor d' Ube4b. Considerant que Ube4b presenta alts nivells d'expressió particularment en teixit nerviós, esperem que la proteïna de fusió presenti un patró d'expressió similar. El transcrit quimèric ha estat detectat a concentracions destacables en cervell, en els ganlis de l'arrel dorsal i en el nervi ciàtic [9]. Aquests resultats demostren que l'expressió de la proteïna quimèrica es dóna en el sistema nerviós, tant central com perifèric.
Adjuntem una imatge de Mack TG et al. (2001) [22] en la que mostren per tinció immunohistoquímica la presència d'Ube4b/Nmnat1 als teixits neuronals.
Degut a la naturalesa de les triplicacions Wld(s), l'expressió del transcrit quimèric que codifica per la proteïna de fusió es troba sota el control del promotor d' Ube4b. Considerant que Ube4b presenta alts nivells d'expressió particularment en teixit nerviós, esperem que la proteïna de fusió presenti un patró d'expressió similar. El transcrit quimèric ha estat detectat a concentracions destacables en cervell, en els ganlis de l'arrel dorsal i en el nervi ciàtic [9]. Aquests resultats demostren que l'expressió de la proteïna quimèrica es dóna en el sistema nerviós, tant central com perifèric.
Adjuntem una imatge de Mack TG et al. (2001) [22] en la que mostren per tinció immunohistoquímica la presència d'Ube4b/Nmnat1 als teixits neuronals.
Caracterizació de la regió promotora del gen La caracterització de la regió promotora s'ha basat en predir els potencials factors de transcripció que es poden unir a la seqüència promotora, així com la posició en aquesta i el seu score d'interacció. Aquest anàlisi s'ha realitzat pels dos gens de manera independent i mitjançant dos mètodes diferents:
- Programa PROMO.
- Programa en Perl creat per nosaltres:
Ube4b
- PROMO: Considerant un índex de dissimilaritat del 15% i un RE query inferior a 0.1, hem obtingut 23 factors de transcripció que s'uneixen potencialment a la seqüència promotora del gen Ube4b. Destaquem factors de transcripció tan importants com aquells que pertanyen a la família E2F, involucrada en la regulació del cicle cel·lular i la síntesi de DNA. Aquests es poden unir a la seqüència promotora d'Ube4b en 4 llocs diferents. Per altra banda, trobem el factor de transcripció olfactori neuronal (COE1), que es poden unir en 6 llocs diferents, o els factors nuclears de cèl·lules T activades (NF-AT) que es poden unir en 3 posicions diferents.- PROGRAMA en PERL: A partir de les matrius de pesos proporcionades i el programa creat, hem obtingut els següents resultats on es mostra el nom del factor de transcripció, la posició en la que s'uneix, el seu score i el paràmetre p-value. Obtenim scores destacadament alts en els següents factors de transcripció: NF-AT1, el receptor alfa d'àcid retinoic (RXR-α), AP-1 (també conegut com Fos/Jun), el receptor d'andrògen (AR) i YY1. El valor de p-value d'aquestes interaccions és força baix, tal i com és d'esperar; per contra, cal dir que en el cas del factor de transcripció YY1, el p-value obtingut és de 0,67, molt més elevat del que s'espera per un score de 5,71. Pensem que pots ser degut a què el motiu de YY1 és tan sols de 6 nucleòtids, i l'atzar afecta molt en seqüències tan curtes.
Tot comparant ambdós resultats, observem que existeixen diferències en les posicions obtingudes. Aquestes podrien ser degudes a les diferències entre les matrius de pesos proporcionades i les que realment utilitza PROMO, i/o a les diferències ja citades entre els factors de transcripció utilitzats en cada cas. Destaquem la predicció dels motius d'unió dels factors de transcripció següents: PU.1 i AP-1, en què les posicions obtingudes pels dos mètodes coincideix respectivament; i NF-AT1, en què la posició tan sols es diferencia per un nucleòtid, i té un RE query de 0 i un p-value de 0,17. Per altra banda ambdós programes prediuen la interacció de YY1 i NF-κB però en tots dos casos, en posicions força diferents.
A partir de l'anàlisi de la regió promotora d'Ube4b de ratolí, Kaneko et al. (2003) [18] van predir que no hi havia cap TATA box en aquesta regió, però que contenir un seqüència consensus Inr en els nucleòtids de -933 a -927 (com sovint ocorre en els promotors defectius en TATA box). La potencial regió promotora inmendiatament upstream de la seqüència consens Inr, conté dos motius d'unió per al factor de transcripció Sp1. Així com potencials llocs d'unió per als factors de transcripció AP2 i AP4. Mitjanšant la generació d'una sèrie de de constructes amb regions del promotor deleccionades, suggereixen que la seqüència essencial per obtenir una expressió basal d'Ube4b (que conté elements cis) va els nucleòtids -1018 a -943. Destaquen a més a més, que aquesta regió conté seqüències tàndem consens per Sp1. Per tant, hipotetitzen que Sp1 és un factor regulador important per la transcripció.
Finalment, destaquem que la nostra predicció en PROMO inclou com un dels millors potencials factors de transcripció (RE query i equally de 0), a Sp1.
Nmnat1
- PROMO: Considerant un índex de dissimilaritat del 15% i un RE query inferior a 0.1, s'han predit 3 factors de transcripció: YY1 del que es prediuen 10 llocs potencials d'interacció diferents; i dos factors de transcripció de la família d'enhancer binding protein (C/EBP) que regulen el creixement i diferenciació de diversos tipus cel·lulars, es tracta de C/EBP α, del que es prediuen 10 llocs d'interacció, i C/EBPβ del que se'n prediuen 19.- PROGRAMA en PERL: Obtenim scores destacadament alts en els següents factors de transcripció: AP-1, AR, c-MYC, NF-AT1, NF-κβ i SRF. La majoria d'ells presenten un p-value baix, però, com en el cas de l'anàlisi d'Ube4b, en trobem alguns dels quals el p-value és més elevat del que s'espera.
Si comparem els dos mètodes, veiem que alguns factors de transcripció obtinguts en el programa en Perl prediuen la mateixa posició que mostra el programa PROMO, tot i que alguns tenen un RE query molt elevat. Així, trobem: AP-1 i PU.1 del que coincideixen les posicions predites per ambdós programes, tot i que PU.1 presenta un RE query molt alt; NF-AT1 i NF-κβ, dels que les posicions es diferencien tan sols en un nucleòtid; i YY1 i AhR, que tots dos programes han trobat com a potencials factors de transcripció, però en posicions força diferents.
Gen quimèric Ube4b/Nmnat1
Donat que el gen quimèric es troba sota el control del promotor d'Ube4b, esperem trobar una regulació transcripcional anàloga a aquest.Estudi de la funció del gen
Ube4b
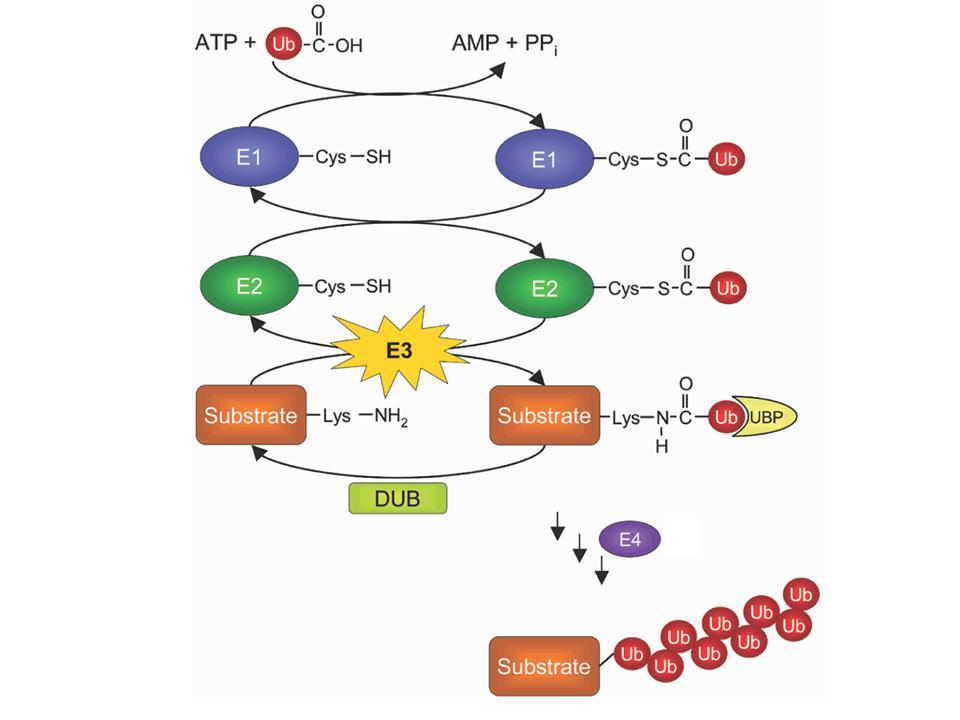 Ube4b, homòleg de UFD2 en S. cerevisiae (ubiquitin factor degradation protein 2) és una proteïna citoplasmàtica que forma part del complex ubiquitina lligasa. Concretament és el factor d'ubiquitinació E4, que s'encarrega de multiubiquitinar als substrats que han estat prèviament monoubiquitinats per E3. El seu mecanisme d'acció és desconegut. Té capacitat d'unió a enzims i a altres proteïnes. Està implicada en processos biològics com l'apoptosi, el plegament de proteïnes, la resposta a UV i els que li donen el seu nom: catàlisi de proteïnes depenent d'ubiquitines.
La degradació selectiva de proteïnes juga un paper molt important en la regulació cel·lular. La progressió en el cicle cel·lular, per exemple, està substancialment regulada per la degradació programada de determinades ciclines, inhibidors de ciclines, etc. en cada moment del cicle. De la mateixa manera, alteracions de programes de transcripció o desenvolupament sovint estan controlats per una degradació programada de proteïnes reguladores. Per altra banda, la degradació selectiva de proteïnes és essencial també per protegir a la cèl·lula en situacions d'estrès degut a què és el sistema més utilitzat per degradar proteïnes aberrants, etc. En eucariotes, la degradació selectiva de proteïnes ocorre majoritàriament per la via ubiquitinació/proteasoma. Els substrats d'aquesta ruta són reconeguts per factors del sistema conjugat d'ubiquitines i marcats per a la degradació amb la unió d'ubiquitines directament a la seva estructura proteica. L'ubiquitinació involucra una sèrie reaccions molt ben definides, que estan catalitzades per una àmplia gamma d'enzims diferents. Destaquem els imprescindibles en la primera fase de la ubiquitinació, el reconeixement del substrat i el marcatge amb la primera ubiquitina. Els factors implicats són els coneguts E1, E2 i E3.
Ube4b, homòleg de UFD2 en S. cerevisiae (ubiquitin factor degradation protein 2) és una proteïna citoplasmàtica que forma part del complex ubiquitina lligasa. Concretament és el factor d'ubiquitinació E4, que s'encarrega de multiubiquitinar als substrats que han estat prèviament monoubiquitinats per E3. El seu mecanisme d'acció és desconegut. Té capacitat d'unió a enzims i a altres proteïnes. Està implicada en processos biològics com l'apoptosi, el plegament de proteïnes, la resposta a UV i els que li donen el seu nom: catàlisi de proteïnes depenent d'ubiquitines.
La degradació selectiva de proteïnes juga un paper molt important en la regulació cel·lular. La progressió en el cicle cel·lular, per exemple, està substancialment regulada per la degradació programada de determinades ciclines, inhibidors de ciclines, etc. en cada moment del cicle. De la mateixa manera, alteracions de programes de transcripció o desenvolupament sovint estan controlats per una degradació programada de proteïnes reguladores. Per altra banda, la degradació selectiva de proteïnes és essencial també per protegir a la cèl·lula en situacions d'estrès degut a què és el sistema més utilitzat per degradar proteïnes aberrants, etc. En eucariotes, la degradació selectiva de proteïnes ocorre majoritàriament per la via ubiquitinació/proteasoma. Els substrats d'aquesta ruta són reconeguts per factors del sistema conjugat d'ubiquitines i marcats per a la degradació amb la unió d'ubiquitines directament a la seva estructura proteica. L'ubiquitinació involucra una sèrie reaccions molt ben definides, que estan catalitzades per una àmplia gamma d'enzims diferents. Destaquem els imprescindibles en la primera fase de la ubiquitinació, el reconeixement del substrat i el marcatge amb la primera ubiquitina. Els factors implicats són els coneguts E1, E2 i E3. En aquest marc teòric, Koegl et al. (1999) [20] descriviren el nou factor d'ubiquitinació Ube4b, anteriorment conegut com a UFD2 en llevat.
Es basaren en demostrar, en primer lloc que la multiubiquitinació és necessària per a què els substrats proteics ubiquitinats siguin degradats via el proteasoma; i suggerien que era imprescindible l'existència d'un nou factor d'ubiquitinació, a més a més de E1, E2 i E3 ja definits, que dugués a terme aquest darrer pas en el marcatge: l'addició de la cadena de multiubiquitines en substrats monoubiquitinats.
Per assajos amb GST-UFD2 per una banda i assajos expressant UFD2 per sistema de baculovirus, demostraren que la síntesi de llargues cadenes de multiubiquitines era directament dependent de la concentració d' UFD2 en el citoplasma cel·lular. UFD2 estimula exageradament la reacció d'ubiquitinació obtenint conjugats de proteïnes marcades amb cadenes destacadament més llargues que les sintetitzades per E1, E2 i E3 sols. E4 no és necessari per a les primeres fases de la ruta d'ubiquitinació, però resulta imprescindible per al darrer pas. E4 no presenta afinitat per substrats que no hagin estat conjugats ja a alguna ubiquitina i sembla interaccionar amb els seus substrats tan sols per la ubiquiitna ja conjugada a aquests.
Per altra banda, Johnson ES et al. (1995) [17] amb estudis amb UFD2 de llevat demostren que la ruta de degradació proteica mitjançant E4 no és essencial en condicions normals, però Koegl et al. (1999) [20] demostren que pren extrema importància quan els llevats són exposats a estrés ambiental (altes temperatures, presència de metalls pesants, etanol, etc.). Així doncs, E4 en llevat, està funcionalment relacionat amb la resistència a l'estrès, és a dir, en la degradació de proteïnes aberrants formades en situacions d'estrès.
Estudis posteriors realitzats amb cèl·lules de mamífer, suggerien que E4 és essencial per al desenvolupament del teixit cardíac així com per la protecció de les neurones espinocerebelars de la degeneració induïda per estrés [19].
Nmnat1
 Nmnat1, altrament anomenat D4COLE1E, és una proteïna nuclear i està implicada en el procés de biosíntesis de NAD+ (nicotinamida adenina dinucleòtid); concretament, és una adeniltransferasa de mononucleòtids de nicotinat i nicotinamida. Té capacitat d'unir-se a magnesi i a proteïnes.
Nmnat1, altrament anomenat D4COLE1E, és una proteïna nuclear i està implicada en el procés de biosíntesis de NAD+ (nicotinamida adenina dinucleòtid); concretament, és una adeniltransferasa de mononucleòtids de nicotinat i nicotinamida. Té capacitat d'unir-se a magnesi i a proteïnes.
Nmnat1 catalitza el darrer pas en la ruta de biosíntesi de NAD+ (dinucleòtid de nicotinamida i adenina), es tracta de la reacció:
NMN (nicotinamida mononucleòtid) + ATP --> NAD+ + Ppi
La molècul·la de NAD+ juga un paper molt fonamental con a cofactor en reaccions redox cel·lulars, també serveix de substrat en reaccions involucrades en regulació com modificacions covalent de proteïnes.
En cèl·lules de mamífers, el NAD+ pot ser sintetitzat a partir de mútlpiles precursors, incloent el triptòfan, l'àcid nicotínic, la nicotinamida i nicotinate riboside(NmR) i mitjançant múltiples passos enzimàtics.
Partint de la propietat axonal-protectora adjudicada al cofactor NAD+ en múltiples estudis, Sasaki J el al. 2006 [26], realitzaren varis assajos per observar si aquesta propietat variava segons la ruta biosintètica de la que provingués el NAD+. Finalment demostraren que Nmnat 1 és l'enzim de biosíntesi de NAD+ que es relaciona amb el major efecte protector (versus la nicotinamida fosforil transferasa i l'àcid nicotínic fosforil transferasa també assajades en l'estudi).
Jia H et al. (2007) [16], han demostrat recentment que el residu crític per la funció de Nmnat1 és una Phe present a l'hèlix A. Per altra banda, observaren que en ratolins Wld(s) amb aquest residu mutat en els dos cromosomes, no té lloc la protecció enfront la degeneració dels axons i per tant, no es retarda la degeneració Walleriana. Així, es confirma que l'activitat de Nmnat1 és imprescindible pel retardament de la degeneració axonal, i que aquesta depèn de la presència del residu de Phe de l'hèlix A de la proteïna.
Gen quimèric Ube4b/Nmnat1
La localització subcel·lular de la proteïna de fusió ha estat extensament estudiada i ha donat lloc a hipòtesis molt divergents. Fins el moment, podríem considerar dues tendències: per una banda, una localització axonal, i per altra banda, nuclear.
La localització axonal de la proteïna de fusió parteix de la base de considerar que aquesta és la responsable del retard en el progrés de la degeneració Walleriana en axons malmesos; i més concretament, que la millora és deguda a un increment de l'activitat de Nmnat1.
Aquesta hipòtesi va ser suggerida per Wang et al. (2005) [28] en observar protecció a curt termini d'axons malmensos després de l'aplicació de NAD+ en altes concentracions. Degut a què altres grups obtingueren resultats oposats [8], s'acabà per deduir que la protecció axonal deguda a NAD+ depenia de certes condicions com l'estrés i la lesió que pateixin els axons. Paral·lelament, es realcionà el retardament de la degeneració Walleriana a les altes concentracions de Ca2+ intracel·lular degudes a la despolarització neuronal [6]; i coneixent que ADP-Riboses cícliques (cADPR) i NADP, metabòlits provinents de NAD+ i NADP respectivament, són responsables de l'alliberament de Ca2+ intracel·lular al citoplasma; s'hipotetitzà que l'increment de Ca2+ intracel·lular podria ser el mecanisme en què es basava la protecció aconseguida amb el suplement de NAD+ [4]. Aquesta consideració, es suggerí que la caiguda de NAD+ trobada per Wang et al. (2005) [28] en axons wt lesionats, podria ser deguda a la conversió d'aquest en cADPR [11]. Cal puntualitzar que Coleman et al. (2005) [7] varen descriure que concentracions excessivament altes de Ca2+ intracel·lular afavorien a la degeneració dels axons. Amb aquesta nova dada, podrem concloure que la regulació dels nivells de Ca2+ intracel·lular resulta realment important en el progrés de processos neurodegeneratius. A més a més, coneixent que el progrés de la degeneració Walleriana avança com una ona des del lloc de lesió, un mecanisme basat en l'increment localitzat de NAD+ en el lloc de lesió, podria ser un mecanisme força efectiu com a primera línia de defensa [11].
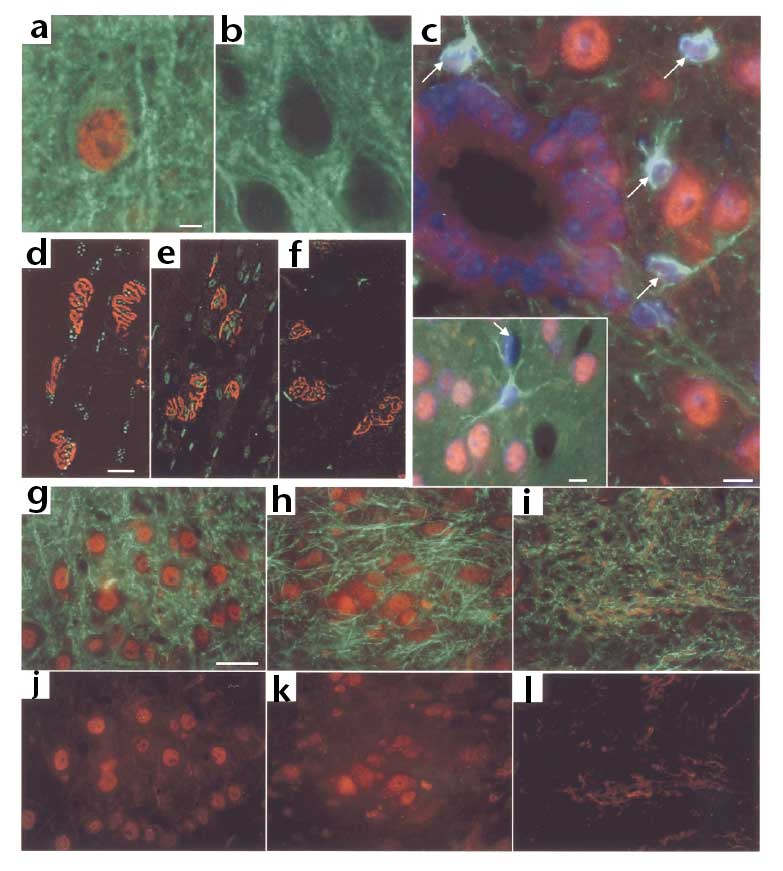
 Pel que fa l'altre hipòtesi, la localització nuclear de la proteïna es basa en observacions de proves inmunohistoquímiques [12][22]. En aquests assajos demostren que tant Nmnat1 en ratolins wt com la proteïna de fusió i Nmnat en ratolins Wld(s) es troben exclusivament en el nucli de les neurones. Creiem oportú adjuntar algunes d'aquestes imatges de microscopi de fluorescència perquè en elles es visualitza molt clarament la demostració de la hipòtesi.
Pel que fa l'altre hipòtesi, la localització nuclear de la proteïna es basa en observacions de proves inmunohistoquímiques [12][22]. En aquests assajos demostren que tant Nmnat1 en ratolins wt com la proteïna de fusió i Nmnat en ratolins Wld(s) es troben exclusivament en el nucli de les neurones. Creiem oportú adjuntar algunes d'aquestes imatges de microscopi de fluorescència perquè en elles es visualitza molt clarament la demostració de la hipòtesi.
La contundència d'aquests resultats semblen desmontar tota la primera hipòtesi, però com en tot en biologia, cal considerar totes les hipòtesis enunciades i intentar arribar a la conclusió més integrativa possible. Els experiments d'inmunohistoquímica esmentats s'han realitzat en situació normal en què les neurones no havien estat exposades a estrés ni tenen els axons malmesos. Així, s'ha especulat que després de produir-se una lesió en els axons, tingui lloc una expressió regulada i transitòria de proteïnes nuclears en els axons per un increment localitzat de la traducció dels transcrits corresponents en el lloc de lesió [15]. Per això, seria necessari que el transcrit de Wld(s) contingui seqüències que el portin específicament a l'axó (axon-targeting). Així doncs, cal realitzar nous experiments que permetin caracteritzar més profundament el transcrit quimèric de Wld(s) per avançar en la demostració de la hipòteisi integrativa enunciada.
En l'estudi de la funció de la proteïna de fusió, destaca la consideració de que és la proteïna responsable del retardament del progrés de la degeneració Walleriana observat en els ratolins de la soca C57BL/Wld(s)[9].
En els estudis de Araki T et al. (2004) [2] i Wang J et al. (2005) [28], s'adjudica al cofactor NAD+ una propietat protectora axonal. Suggerint així que el fenotip dels ratolins Wld(s) venia donat substancialment per la sobreexpresió d'Nmnat1. Ara bé, recentment, Conforti et al (2007) [8] han demostrat que l'efecte protector de NAD+ només es dóna en determinades situacions, i que expressions de Nmnat1 a nivells similars que en Wld(s), no protegien tan efectivament. Les seves conclusions han estat que Nmnat1 i la proteïna de fusió de Wld(s) no són equivalents en la seva capacitat per retardar la degeneració Walleriana; demostrat per assajos ultraestructurals, morfològics i bioquímics in vivo i utilitzant lesions traumàtiques i tòxiques in vitro. Les seves conclusions no neguen la involucració de Nmnat1 en el fenotip Wld(s), sinó que suggereixen que aquesta no és suficient, i que la regió N-terminal, amb una funció encara desconeguda, també es requereix per una forta protecció axonal.
Gillingwater et al. (2006) [14] demostraren que l'expressió de la proteïna de fusió Wld(s) alterava selectivament els nivells d'mRNA d'altres gens en cèl·lules de cerebel de ratolins de la soca C57BL/Wld(s) in vivo i seguidament, en cèl·lules embriòniques de ronyó humà in vitro. Transfeccions amb construccions que contenien tota la longitud de Nmnat1 o el fragment N70-Ube4b demostraren que ambdós tenien efectes selectius diferents: Nmnat1 causava una disminució de l'expressió de 10 vegades de pttg1 (pituitary tumour-transforming gene-1) (10-fold downregulation); i N70-Ube4b causava l'augment de l'expressió 5 vegades de edr1l-EST (structural homologue of erythroid differentiation regulator-1). Gillingwater et al. concluien que la proteïna de fusió Wld(s) té capacitat per regular l'expressió d'un grup de gens tant en neurones de ratolí com en cèl·lules d'humà [14]. Aquests resultats donen suport a la hipòtesi plantejada per Laura Conforti et al. (2007) [8] insistint en què el retardament de la degeneració Walleriana no només deu a la sobreexpressió de Nmnat1 sinó que participa la totalitat de la proteïna quimèrica.
Coneixent que malalties neurodegeneratives cursen amb l'acumulació de substrats multiubiquitinats com les plaques neurítiques en l'Alzheimer [24] o els cossos de Lewy en el Parkinson, es suggereix que la ruta proteolítica per ubiquitinació, juga un paper molt important en la degeneració axonal així com en la neurodegeneració en general. Considerant l'alteració de la ruta d'ubiquitinació suggerida per Conforti et al. (2000) [9], els 70 aminoàcids N-terminal d'Ube4b podrien estar relacionats amb el retardament del progrés de la degeneració Walleriana.
Pel que fa a la seqüència linker de 18 aminoàcids cal dir que la seva funció no ha estat fins ara descrita. S'han realitzat nombrosos estudis que descriuen diverses hipòtesis sobre la seva significaciò i/o efectes. Recentment, Laser et al. (2006) [21] suggereixen que aquesta seqüència és la responsable de la localització nuclear de la proteïna de fusió.
Conclusions |
Els ratolins que presenten la degeneració Walleriana retarda, (C57BL/Wld(s)) (slow Wallerian degeneration), són una soca mutant de ratolí amb una única variació fenotípica dominant: la prolongació de la supervivència dels axons distals després d'una axotomia, és a dir, el retardament del progrés de la degeneració Walleriana en resposta a una lesió axonal.
La mutació causant és una triplicació en tàndem en la regió qE2 del cromosoma 4. Les unitats repetides involucren a tres gens: En les regions proximals i distals de les unitats de repetició es troben, respectivament, fraccions del gen Ube4b, i d'un segon gen, no descrit anteriorment, Nmnat1. En la regió central es troba el tercer gen afectat, Rbp7.
L'existència dels ratolins mutants Wld(s) indica, en primer lloc, que la degeneració del axons és un procés actiu i instrínsec a la naturalsea dels axons; i en segon lloc, que el procés està regulat genèticament i no està forçosament relacionat amb les rutes clàssiques de mort cel·lular en els cossos neuronals. Les experimentacions amb aquesta soca de ratolí mutant ha despertat molt d'interès per a la comunitat científica com a font de possibles novetats terapèutiques per a les malalties neurodegeneratives.
S'han indentificat dos transcrit directament alterats per la triplicació Wld(s): l'mRNA quimèric que codifica per Ube4b/Nmnat1, i el mRNA complert de Rbp7 que és sobreexpressat sense alteracions en la seva seqüència. L'expressió d'Ube4b/Nmnat1 es troba sota el control del promotor d'Ube4b a causa de la naturalesa del mRNA quimèric. El transcrit s'expressa en molts diversos teixits, però la seva expressió és especialment destacable en el teixit nerviós, tant central com perifèric. En canvi, Rbp7 mostra els majors índex d'expressió en teixit adipós i glàndules mamàries. L'anàlisi dels patrons d'expressió d'aquests dos transcrits suggereix clarament que el responsable del fenotip Wld(s) és Ube4b/Nmnat1.
Una altra conseqüència de que la expressió de la proteïna quimèrica estigui sota el control del promotor d'Ube4b, és que esperem que aquesta estigui regulada pels mateixos elements cis i per la unió dels mateixos elements de transcripció. Destaquem el paper regulador del factor de transcripció Sp1.
La formació del mRNA quimèric recau en la fusió de les seqüències dels gens d'Ube4b i Nmnat1 en els límits proximal i distal, respectivament, de les unitats de repetició Wld(s). De tal manera que la proteïna de fusió resultant està formada pels 70 aminoàcids N-terminals d'Ube4b, 18 aminoàcids linkers i la seqüència completa de Nmnat1, 285 aminoàcids. La seqüència linker està codificada per una fracció de l'exó 2 de Nmnat 1 que no codifica normalment en la traducció de Nmnat1 wt.
Respecte l'estudi d'homologia, cal destacar que els gens ortòleg d'Ube4b i Nmnat1 trobats en humans presenten respectivament un 96% i un 80% d'homologia. Fet que resulta molt interesant per poder aplicar la recerca dels gens implicats en l'alteració Wld(s) en terapèutica per a malalties neurodegeneratives.
La proteïna quimèrica té una localització nuclear demostrada per diversos estudis mitjanšant assajos d'inmunohistoquímica in vivo. Considerant que Ube4b és una proteïna citoplasmàtica i Nmnat1 nuclear, s'ha plantejat la possibilitat de que la seqüència linker entre aquests dos sigui la responsable del transport de la proteïna quimèrica al nucli.
Durant molt de temps s'ha pensat que el retardament de la degeneració Walleriana en ratolins Wld(s) venia donat per la sobreexpressió d'Nmnat1, que feia incrementar la síntesi de NAD+ i aquest tenia un efecte protector axonal. Recentment, s'ha demostrat que aquest efecte només ocorre en determinades situacions i que l'expressió de Nmnat a mateixos nivells que Wld(s), no ofereix una protecció tan efectiva. Paral·lelament s'ha demostrat, per una banda, que la proteïna de fusió té capacitat per regular l'expressió d'un conjunt de gens en neurones de ratolí i en cèl·lules humanes i per l'altra banda, que els 70 aminoàcids N-terminals d'Ube4b podrien estar alterant les rutes d'ubiquitinació. Considerant el conjunt d'aquests resultats podem concloure que Nmnat1 no és suficient per causar el retardament de la degeneració Walleriana sinó que el fenotip de Wld(s) està causat per la totalitat de la proteïna de fusió, involucrant també el paper d'altres regions de la proteïna de fusió com són els 70 aminoàcids de la regió N-terminal d'Ube4b.
Seran necessaris més estudis per arribar a conèixer exactament el mecanisme d'acció mitjanšant el qual la proteïna de fusió aconsegueix el retardament de la degeneració Walleriana; coneixement que serà de gran utilitat per aprofondir en el saber de les malalties degeneratives en humans i possibles aplicacions terapèutiques d'aquestes.
Materials i Mètodes |
- Caracterització de l'estructura genòmica del gen
Identificació del gen codificant per la seqüència donada: NCBI tblastn. Utilitzem la seqüència de la proteïna per trobar la seqüència de mRNA corresponent trobada en la base de dades d'NCBI. Dels resultats del tblastn, seleccionem la seqüència que presenta E-value= 0. Així concluïm que es tracta del gen quimèric UFD2/D4COLE1E, altrament dit Ube4b/Nmnat1. Els resultats han estat corroborats amb UCSC blat. La base de dades de la Universitat de Califòrnia no té entrada pel gen ni per la proteïna de fusió com a tals, sino que els resultats del BLAT mostren l'alineament amb els dos gens, Ube4b i Nmnat1, per separat; cadascún amb la regió corresponent de la proteïna de fusió.
Anàlisi dels transcrits i isoformes dels gens: Ensembl. Per estudiar els transcrits dels dos gens identificats, en primer moment fem la cerca en tres bases de dades: l'Ensembl, l'NCBI i l'UCSC. Les tres ens ofereixen informacions molt diferents pel que fa al nombre i naturalesa dels transcrits dels dos gens. La comparació i l'anàlisi dels resultats així com de les diferències polítiques entre les dues bases de dades, ens porta a prendre una decisió. Utilitzem Ensembl perquè coneixem que és molt més restrictiu a l'hora d'identificar com a transcrits les seqüències de mRNA corresponents a un determinat gen. Aleshores, tota la informació pertanyent a l'anàlisi dels transcrits i de les isoformes corresponents, està extreta de la base de dades de l'Ensembl. La cerca l'hem realitzat buscant en primer lloc les pàgines web principals dels dos gens, i seguidament, navegant per les pàgines web enllaçades amb la primera.
Anàlisi de les dues isoformes d'Ube4b: Clustal W. Per estudiar les diferències entre les dues isoformes del gen Ube4b, així com per comparar els dos marcs de lectura, hem alineat les dues seqüències proteiques mitjançant el servidor web del programa Clustal W. L'alineament obtingut ha facilitat la visualització comparativa.
Anàlisi dels dominis de les isoformes: UniProtKB/Swiss-Prot, Pfam, InterPro. Per predir els possibles dominis de la proteïna de fusió Ube4b/Nmnat1 analitzem en primer lloc els dominis presents en les dues proteïnes originals: Ube4b i Nmnat1, així com els residus que abarquen. En segon lloc, estudiem si aquestes regions estan presents en la proteïna de fusió i deduïm els dominis que pot tenir Ube4b/Nmnat1.
Estudi de la proteïna de fusió: NCBI i Ensembl. Donat que NCBI és l'única base de dades en què constava la proteïna de fusió Ube4b/Nmnat1 com a tal, ens hem basat en les dades obtingudes en el tblastn per identificar les fraccions de la proteïna que corresponen a cada gen, etc. Combinant aquests resultats amb la informació dels transcrits extreta de l'Ensembl, descobrim quin model d'splicing (referent a Ube4b) té lloc en el gen quimèric així com les modificacions de les regions no codificants i codificants (referent a Nmnat1).
Altres materials de consulta: MGI, PubMed. La base de dades MGI (Mouse Genome Informatics) ens ha estat útil en tot moment per la cerca d'informació general sobre els gens estudiats així com per trobar enllaços interessants a altres bases de dades i a les referències dels gens, totes elles citades en els resultats.
Estudi de la homologia del gen en altres espècies Estudi de l'homologia dels gens en altres espècies: BIOMART, Ensembl, PubMed. Per a la identificació dels ortòlegs dels dos gens i dels percentatges d'homologia corresponents, ens hem basat en la informació que ofereix l'Ensembl en la pàgina web principal dels gens. La base de dades obtè aquests resultats del programa BIOMART. En la taula d'ortòlegs que mostra Ensembl apareixen dos percentatges d'homologia diferents sota els títols de "Query" i "Target". El primer d'aquests mostra el percentatge d'homologia de la seqüència introduïda respecte la de l'ortòleg trobat, mentre que el segon, el percentatge d'homologia de la seqüència de l'ortòleg trobat respecte la seqüència introduïda. Per això mateix, el valor escollit ha estat el de "Query". Finalment hem contrastat la informació amb la dels articles trobats a PubMed que tractaven la temàtica pertinent.
Caracterització de l'expressió del gen Estudi de l'expressió dels gens: GNF SymAtlas v1.2.4, VisiGene, PubMed. Per estudiar l'expressió d'un determinat gen podem basar-nos en varies bases de dades que ens ofereixen informacions de naturalesa molt diferent. En primer lloc, hem utilitzat les bases de dades específiques d'expressió: El GNF Atlas 2, enllaçat des de UCSC, i el SymAtlas versió 1.2.4. Totes dues es basen en dades obtingudes per experiments amb Mycroarrays. Tot i donar informacions molt similars, la presentació d'aquesta es dóna en dos formats molt diferents. Hem preferit mostrar les dades en el format de gràfic de barres oferit per SymAtlas perquè hem considerat que aquest era molt més entenedor que el format oferit per GNF Atlas 2 (format anàleg a la coloració clàssica de Mycroarrays. En segon lloc, hem cercat en la base de dades de VisiGene oferida per l'UCSC. Aquesta conté imatges reals preses en procediments experimentals que evidencien l'expressió del gen en determinats teixits de l'animal. En tercer lloc, es podrien analitzar els EST (Expressed Sequence Tag) dels gens. Les bases de dades com l'Ensembl i l'UCSC ofereixen informació sobre els EST trobats per a cada gen. Segons el nostre punt de vista, en tots dos casos el format en què mostren la informació de les singularitats de cada EST (la soca, el teixit, el procediment experimental, etc.) és molt poc entenedor, i en conclusió massa heterogeni com per dur-ne a terme un anàlisi acurat. Per aquest motiu, hem preferit no incloure'l en els nostres resultats. Finalment hem contrastat la informació amb la dels articles trobats a PubMed que tractaven la temàtica pertinent.
Caracterizació de la regió promotora del gen Obtenció de la regió promotora en format FASTA: UCSC. A l'opció de Genome Browser de la base de dades d'UCSC hem insertat els noms dels gens: Ube4b i Nmnat1. Ube4b és el gen que conté el promotor que determinará l'expressió de la nostra proteïna de fusió ja que la seqüència genòmica que es fusiona a causa de les repeticions situa en primer lloc una sèrie de nucleòtids d'Ube4b. Un cop realitzada la sel·lecció hem pogut obtenir un document en format FASTA que conté la seqüència genòmica que va des d'1kb upstream del TSS (transcription start point) fins a 100bp downstream d'aquest.
Predicció dels factors de transcripció que s'uneixen a la regió promotora mitjançant un programa en Perl. Partint d'un document que conté les matrius de pesos de 13 factors de transcripció humans, el programa Perl realitzat és capaç de predir les millors posicions d'unió dels 13 factors de transcripció en la seqüència promotora, així com indicar l'score de la interacció i el p-value. L'escriptura del codi Perl s'ha dut a terme seguint les instruccions oferides en l'enunciat del problema. El programa Perl s'ha executat des del terminal i redireccionant els resultats en un document de text. Seguidament convertim aquest document en format de full de càlcul que ens permet col·locar les dades per columnes i ordenar els resultats extrets en funció de l'score de cada interacció. Els resultats obtinguts han estat comparats amb els obtinguts mitjançant PROMO.
Predicció dels factors de transcripció que s'uneixen a la regió promotora mitjançant PROMO. El servidor web del programa PROMO, per defecte, realitza la cerca permetent un 15% de dissimilaritat entre els motius consens del factor de transcripció i els motius trobats en la pròpia seqüència. Nosaltres realitzem diverses cerques variant aquest valor a 10 i 0 (Data not shown). Hem restat amb els valors oferts per defecte. L'anàlisi dels resultats obtinguts l'hem fet mitjançant l'observació dels valors de RE equally i RE query corresponents a cada factor de transcripció i interacció trobades. Per tal de facilitar aquest anàlisi extreiem el document de PROMO, el convertim en format de text i seguidament, en format de full de càlcul. El full de càlcul ens permet col·locar les dades per columnes i ordenar els resultats extrets en funció de RE equally i RE query. De tota la llista obtinguda, que inicialment conté més de 300 factors de transcripció, seleccionem aquells que tenen un RE query inferior a 0,10 (per tant, amb la seqüència promotora de 1100 nucleòtids, la probabilitat de trobar la seqüència del motiu a l'atzar és inferior a 0,009% en tots els casos seleccionats). El programa PROMO ens proporciona una sèrie de variables després d'analitzar la seqüència promotora introduïda a partir de les matrius de pesos de cada factor de transcripció. Ens indica la seqüència on s'uneix el motiu, així com la primera i darrera posició, el grau de dissimilaritat que presenta la nostra seqüència envers la del motiu del factor de transcripció i uns valors de Random Expectation (RE): RE equally i RE query. RE ens mostra el nùmero de vegades que esperem trobar en una seqüència a l'atzar de la mateixa longitud que la seqüència promotora introduïda tenint en compte el grau de dissimilaritat. El valor de RE equally ens informa de la probabilitat de trobar la mateixa seqüència de nucleòtids en una cadena de DNA de mateixa longitud que la insertada però amb una proporció de nucleòtids del 25%. En canvi, RE query indica la mateixa probabilitat però a partir de la mateixa proporció de nucleòtids que la seqüència promotora introduïda. Aquests resultats han estat comparats amb els obtinguts mitjançant el programa en Perl dissenyat.
Informació específica dels factors dels factors de transcripció: TRANSFAC. Hem consultat aquesta base de dades per obtenir més informació sobre els factors de transcripció que hem analitzat.
Informació de la regió promotora del gen quimèric: Pubmed.
Estudi de la funció del gen Estudi de la funció dels gens segons identificadors GO: Gene Ontology i Ensembl. Hem cercat en les bases de dades Gene Ontology i Ensembl els identificadors que atribuïen a cada un del gens en estudi. Hem navegat pel identificadors i hem comparat els resultats d'ambdues bases de dades. Hem seleccionat els identificadors més significatius recollint-los finalment en taules.
Estudi de la funció dels gens a partir de publicacions: PubMed i IHOP. S'ha realitzat una recerca intensiva de publicacions científiques directament en la base de dades PubMed. En paral·lel, s'ha utilitzat també el servidor IHOP (Information Hyperlinked Over Proteins) que a partir de la paraula clau introduïda, proporciona links a articles, mostrant la paraula clau en el context en què apareix en aquests. Amb aquestes dues cerques, hem aconseguit la informació necessària per comprendre adequadament la funció dels gens i la seva implicació en la degeneració Walleriana retardada (WIds).
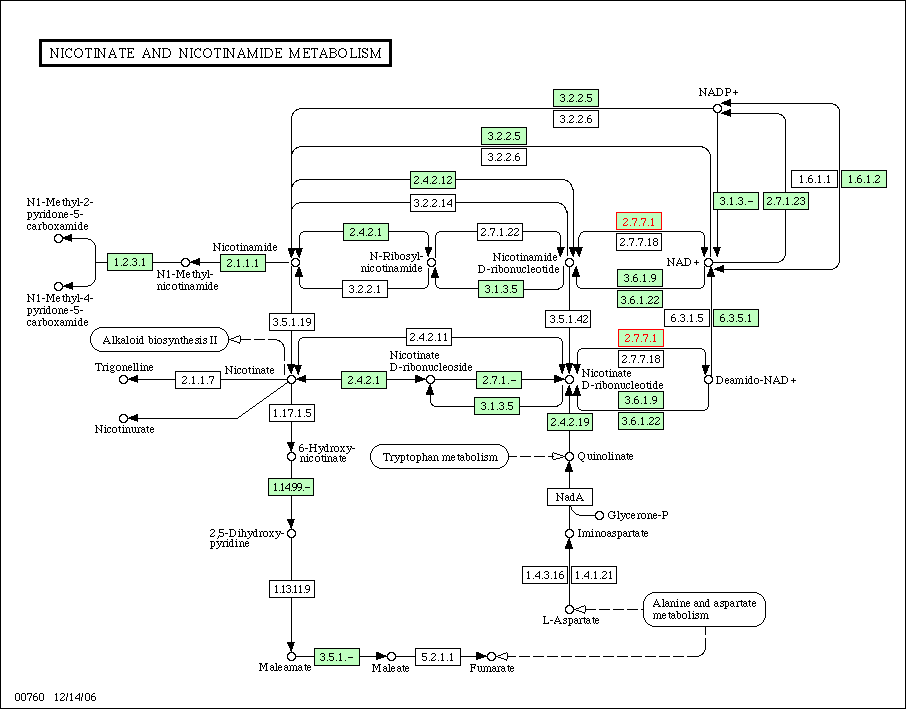
Estudi de la funció de Nmnat1 en el metabolisme: KEGG (Kyoto Encyclopedia of Genes and Genomes). La base de dades KEGG ens ha ofert un esquema que situa la funció de Nmnat1 dins la ruta metabòlica de nicotinat i nicotinamida.
Construcció de la pàgina web Aquesta pàgina web ha estat realitzada mitjançant llenguatge HTML.
Referències |
- ARTICLES
- Adalbert R, Gillingwater TH, Haley JE, Bridge K, Beirowski B, Berek L et al. A rat model of slow Wallerian degeneration (Wld(s)) with improved preservation of neuromuscular synapses. Eur J Neurosci. 2005 Jan;21(1):271-7.
- Araki T, Sasaki Y, Milbrandt J. Increased nuclear NAD biosynthesis and SIRT1 activation prevent axonal degeneration. Science. 2004 Aug 13;305(5686):1010-3.
- Beirowski B, Adalbert R, Wagner D, Grumme DS, Addicks K, Ribchester RR et al. The progressive nature of Wallerian degeneration in wild-type and slow Wallerian degeneration (Wld(s)) nerves. BMC Neurosci. 2005 Feb 1;6(1):6.
- Berger F, Ramirez-Hernandez MH, Ziegler M. The new life of a centenarian: signalling functions of NAD(P). Trends Biochem Sci. 2004 Mar;29(3):111-8.
- Berger F, Lau C, Dahlmann M, Ziegler M. Subcellular compartmentation and differential catalytic properties of the three human nicotinamide mononucleotide adenylyltransferase isoforms. J Biol Chem. 2005 Oct 28;280(43):36334-41.
- Buckmaster EA, Perry VH, Brown MC. The rate of Wallerian degeneration in cultured neurons from wild type and C57BL/Wld(s) mice depends on time in culture and may be extended in the presence of elevated K+ levels.
- Coleman M. Axon degeneration mechanisms: commonality amid diversity. Nat Rev Neurosci. 2005 Nov;6(11):889-98.
- Conforti L, Fang G, Beirowski B, Wang MS, Sorci L, Asress S et al. NAD(+) and axon degeneration revisited: Nmnat1 cannot substitute for Wld(S) to delay Wallerian degeneration.Cell Death Differ. 2007 Jan;14(1):116-27.
- Conforti L, Tarlton A, Mack TG, Mi W, Buckmaster EA, Wagner D et al. A Ufd2/D4Cole1e chimeric protein and overexpression of Rbp7 in the slow Wallerian degeneration (Wld(s)) mouse. Proc Natl Acad Sci U S A. 2000 Oct 10;97(21):11377-82.
- Du L, Zhang X, Han YY, Burke NA, Kochanek PM, Watkins SC et al. Intra-mitochondrial poly(ADP-ribosylation) contributes to NAD+ depletion and cell death induced by oxidative stress. J Biol Chem. 2003 May 16;278(20):18426-33. Epub 2003 Mar 7.
- Fainzilber M, Twiss JL. Tracking in the Wld(s)--the hunting of the SIRT and the luring of the Draper. Neuron. 2006 Jun 15;50(6):819-21.
- Fang C, Bernardes-Silva M, Coleman MP, Perry VH. The cellular distribution of the Wld(s) chimeric protein and its constituent proteins in the CNS. Neuroscience. 2005;135(4):1107-18. Epub 2005 Sep 8.
- Fernando FS, Conforti L, Tosi S, Smith AD, Coleman MP. Human homologue of a gene mutated in the slow Wallerian degeneration (C57BL/Wld(s)) mouse. Gene. 2002 Feb 6;284(1-2):23-9.
- Gillingwater TH, Wishart TM, Chen PE, Haley JE, Robertson K, MacDonald SH et al. The neuroprotective Wld(s) gene regulates expression of PTTG1 and erythroid differentiation regulator 1-like gene in mice and human cells. Hum Mol Genet. 2006 Feb 15;15(4):625-35.
- Hanz S, Perlson E, Willis D, Zheng JQ, Massarwa R, Huerta JJ et al. Axoplasmic importins enable retrograde injury signaling in lesioned nerve. Neuron. 2003 Dec 18;40(6):1095-104.
- Jia H, Yan T Feng Y Zeng C, Shi X, Zhai Q. Identification of a critical site in Wld(s): essential for Nmnat enzyme activity and axon/protective function. Neurosci Lett. 2007 Feb 8;413(1):46-51. nou
- Johnson ES, Bartel B, Seufert W, Varshavsky A. Ubiquitin as a degradation signal. EMBO J. 1992 Feb;11(2):497-505.
- Kaneko C, Hatakeyama S, Matsumoto M, Yada M, Nakayama K, Nakayama KI. Characterization of the mouse gene for the U-box-type ubiquitin ligase UFD2a.Biochem Biophys Res Commun. 2003 Jan 10;300(2):297-304.
- Kaneko-Oshikawa C, Nakagawa T, Yamada M, Yoshikawa H, Matsumoto M, Yada M et al. Mammalian E4 is required for cardiac development and maintenance of the nervous system. Mol Cell Biol. 2005 Dec;25(24):10953-64.
- Koegl M, Hoppe T, Schlenker S, Ulrich HD, Mayer TU, Jentsch S. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 1999 Mar 5;96(5):635-44.
- Laser H, Conforti L, Morreale G, Mack TG, Heyer M, Haley JE et al. The slow Wallerian degeneration protein, Wld(s), binds directly to VCP/p97 and partially redistributes it within the nucleus. Mol Biol Cell. 2006 Mar;17(3):1075-84.
- Mack TG, Reiner M, Beirowski B, Mi W, Emanuelli M, Wagner D et al. Wallerian degeneration of injured axons and synapses is delayed by a Ube4b/Nmnat chimeric gene. Nat Neurosci. 2001 Dec;4(12):1199-206.
- Magni G, Amici A, Emanuelli M, Orsomando G, Raffaelli N, Ruggieri S. Structure and function of nicotinamide mononucleotide adenylyltransferase. Curr Med Chem. 2004 Apr;11(7):873-85.
- Mori H, Kondo J, Ihara Y. Ubiquitin is a component of paired helical filaments in Alzheimer's disease. Science. 1987 Mar 27;235(4796):1641-4.
- Perry VH, Lunn ER, Brown MC, Cahusac S, Gordon S. Evidence that the Rate of Wallerian Degeneration is Controlled by a Single Autosomal Dominant Gene. Eur J Neurosci. 1990;2(5):408-413.
- Sasaki Y, Araki T, Milbrandt J. Stimulation of nicotinamide adenine dinucleotide biosynthetic pathways delays axonal degeneration after axotomy. J Neurosci. 2006 Aug 16;26(33):8484-91.
- Tsao JW, Brown MC, Carden MJ, McLean WG, Perry VH. Loss of the compound action potential: an electrophysiological, biochemical and morphological study of early events in axonal degeneration in the C57BL/Ola mouse. Eur J Neurosci. 1994 Apr 1;6(4):516-24.
- Wang J, Zhai Q, Chen Y, Lin E, Gu W, McBurney MW et al. A local mechanism mediates NAD-dependent protection of axon degeneration. J Cell Biol. 2005 Aug 1;170(3):349-55.
- PROGRAMARI
- PROMO. D. Farré, R. Roset, M.Huerta, J.E.Adsuara, LL.Roselló, M.M.Albà; and X. Messeguer. Identification of Patterns in Biological Sequences at the ALGGEN Server: PROMO and MALGEN:(Abstract)(Full text) Nucleic Acids Research,31 (13):3651-3653
- CLUSTALW. Chenna, Ramu, Sugawara, Hideaki, Koike,Tadashi, Lopez, Rodrigo, Gibson, Toby J, Higgins, Desmond G, Thompson, Julie D. Multiple sequence alignment with the Clustal series of programs. (2003) Nucleic Acids Res 31 (13):3497-500
- UCSC, Blat Alignment Tool. Kent, W.J. BLAT - The BLAST-Like Alignment Tool. Genome Res. 12(4), 656-664 (2002).
- BASES DE DADES
- GENBANK. Benson DA, Karsch-Mizrachi I, Lipman DJ, Ostell J, Wheeler DL. GenBank. Nucleic Acids Res. 34(Database issue), D16-20 (2006)
- ENSEMBL. T. J. P. Hubbard, B. L. Aken, K. Beal1, B. Ballester1, M. Caccamo, Y. Chen et al. Ensembl 2007 Nucleic Acids Res. 2007 Jan 1;
- UCSC Genome Browser. Kent, W.J., Sugnet, C. W., Furey, T. S., Roskin, K.M., Pringle, T. H., Zahler, A. M., and Haussler, D. The Human Genome Browser at UCSC. Genome Res. 12(6), 996-1006 (2002).
- UCSC Genome Browser database/data. Karolchik, D., Baertsch, R., Diekhans, M., Furey, T.S., Hinrichs, A., Lu, Y.T., Roskin, K.M., Schwartz, M., Sugnet, C.W., Thomas, D.J., Weber, R.J., Haussler, D. and Kent, W.J. The UCSC Genome Browser Database. Nucl. Acids Res 31(1), 51-54 (2003).
- UCSC Table Browser. Karolchik, D., Hinrichs, A.S., Furey, T.S., Roskin, K.M., Sugnet, C.W., Haussler, D. and Kent, W.J. The UCSC Table Browser data retrieval tool. Nucl. Acids Res. 32(Suppl 1), D493-D496 (2004).
- UCSC Proteome Browser. Hsu, F., Pringle, T.H., Kuhn, R.M., Karolchik, D., Diekhans, M., Haussler, D., and Kent, W.J. The UCSC Proteome Browser. Nucl. Acids Res. 33(suppl 1), D454-D458 (2004).
- GNF SymAtlas. Su AI, Cooke MP, Ching KA, Hakak Y, Walker JR, Wiltshire T et al. Large-scale analysis of the human and mouse transcriptomes. Proc Natl Acad Sci U S A. 2002 Apr 2;99(7):4465-70. Epub 2002 Mar 19.
- MGI: Mouse Genome Informatics.
- MGD: Mouse Genome Database. Eppig JT, Bult CJ, Kadin JA, Richardson JE, Blake JA, and the members of the Mouse Genome Database Group. 2005. The Mouse Genome Database (MGD): from genes to mice community resource for mouse biology. Nucleic Acids Res 2005; 33: D471-D475.
- GXD: Gene Expression Data. Hill DP, Begley DA, Finger JH, Hayamizu TF, McCright IJ, Smith CM, Beal JS, Corbani LE, Blake JA, Eppig JT, Kadin JA, Richardson JE, Ringwald M. 2004. The mouse Gene Expression Database (GXD): updates and enhancements. Nucleic Acids Res 32: D568-D571.
- TRANSFAC.
Agraïments
We are grateful to...
- Simon Furley! Thank you for your help in programming, because like you know... It's Peeerl !! And thank you for your "empty if" and your "miniwhile"!!
- Charles Chapple! Perquè el verd i el vermell no van sempre com els semàfors...
- Domènec Farré! Per instruir-nos una mica més en el màgic món del PROMO...
- (Laia) Agraeixo moooolt a la Natàlia les seves rialles, el seu positivisme constant i aquesta confiança i companyerisme que m'ha demostrat en tooot moment. Mademoiselle, xsui ravie d'avoir bossé si bien avec vous! ;)
- (Natàlia) Je remercie beaucoup à Laia, ša a été un projet dur mais aussi très amusant!!! À très bientôt!!!
