Para la caracterización de la secuencia utilizaremos un programa realizado en Perl y después, compararemos los resultados obtenidos en nuestro programa con la base de datos de la web PROMO.
La secuencia promotora de nuestros genes la obtenemos a partir de su secuencia nucleotídica proporcionada por la base de datos UCSC. En el enlace correspondiente a la información del gen, descargaremos la secuencia genómica, seleccionando la opción de presentarnos la secuencia promotora (1000 bases upstream). Una vez obtenida, nos quedaremos con los 1100 primeros nucleótidos (1000 de la región promotora + 100 primeros nucleótidos).
Las secuencias promotoras obtenidas para nuestros genes son las siguientes:
A continuación mostramos los resultados obtenidos mediante nuestro programa de indentificación de sitios de unión a promotor en Perl para cada uno de los genes:
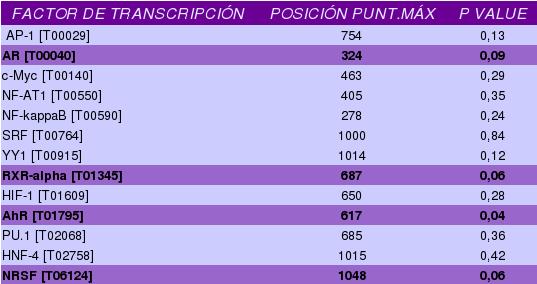
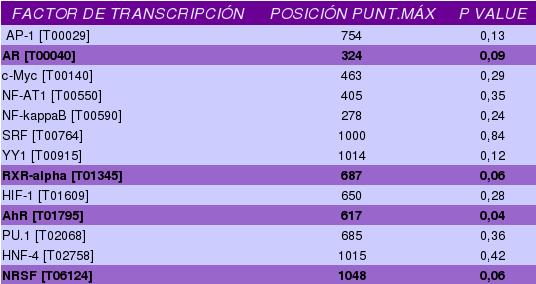
En los siguientes hipervínculos podemos ver los resultados obtenidos mediante la base de datos PROMO tanto para un porcentaje de similitud del 5% como del 15%.
Como podemos observar la comparación entre nuestros resultados y los de PROMO apenas coinciden. En las tablas se encuentran marcados los factores de transcripción que aparecen tanto en las matrices proporcionadas para el programa como en PROMO, aunque el archivo de matrices inicial parece no estar bien adquirido de la base de datos PROMO (no coinciden los datos).