
El gen que codifica para la proteína PML se encuentra en el cromosoma 15 en la localización 72,074,067 - 72,127,204 (15q24.1).

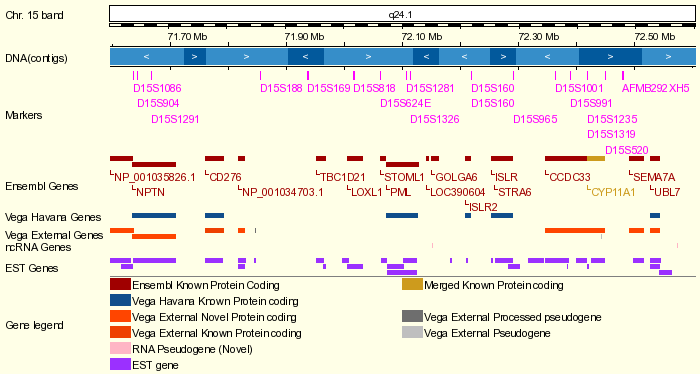
Según la base de datos Ensembl, el gen PML consta de 4 transcritos diferentes:
| TRANSCRITO 1 | 9 exones | 7 exones CD | 0 exones NCD | 2 exones CD/NCD
| 8 exones | 7 exones CD | 0 exones NCD | 1 exón CD/NCD
| TRANSCRITO 3 | 7 exones | 6 exones CD | 0 exones NCD | 1 exón CD/NCD
| TRANSCRITO 4 | 5 exones | 4 exones CD | 0 exones NCD | 1 exón CD/NCD
CD = codificante
NCD = no codificante
CD/NCD = porción codificante y porción no codificante
Como podemos observar en las imágenes anteriores, todos los exones de los transcritos son codificantes exceptuando los exones 1 y 9 que poseen una región codificante y una región no codificante. Ésta última parte se encuentra en los extremos de los transcritos, lo que nos lleva a pensar que puede tratarse de regiones UTR.
En un principio, el número de transcritos determina el número máximo de isoformas a las que puede dar lugar un gen. Por lo tanto, según Ensembl, el gen PML da lugar a 4 isoformas correspondientes a cada uno de sus 4 transcritos.
Hemos querido corroborar estos datos con UCSC Genome Browser, la cual nos asocia 11 isoformas a este gen. Los enlaces de 7 de las isoformas descritas por UCSC nos llevan a la misma secuencia proteica, por lo que de 11 isoformas proporcionadas en un principio, sólo obtenemos 3 proteínas diferentes.
Al no coincidir los datos de UCSC y Ensembl miramos en la base de datos UniProt, donde se describen 5 isoformas para el gen PML producidas por splicing alternativo y con los siguientes nombres: PML-1, PML-2,PML-3, PML-3B y PML-X.
Después de tanta variedad de datos, decidimos realizar un alineamiento múltiple mediante ClustalW, comparando las secuencias pproteicas producidas por los 4 transcritos de Ensembl con las 5 isoformas de UniProt.
Los resultados muestran como el transcrito 1 del Ensembl se corresponde con la isoforma PML-1 y el transcrito 2 con la isoforma PML-3. La secuencia de aminoácidos de los otros dos transcritos y de las otras dos isoformas son totalmente diferentes.
Así, en total (consultando estas tres bases de datos) hemos encontrado 7 variantes proteicas del gen PML. Los resultados de las diferentes bases de datos resultan incongruentes y, por tanto, no son concluyentes ya que en un principio sólo podrían existir 4 isoformas diferentes partiendo de la existencia de 4 transcritos. Esto es lo que nos ha llevado a escoger a Ensembl como la base de datos más fiable a la hora de escoger nuestros resultados.
Como cada una de las isoformas derivan de cada uno de los transcritos, las diferencias se deben a splicing alternativo de algunos de los exones, tal y como hemos descrito en las imágenes anteriores en las que se comparaban los exones que coinciden y los que se han eliminado. Debido a que los exones que se modifican son codificantes, se cambia la pauta de lectura y en cada isoforma se obtiene una secuencia aminoacídica diferente:
El gen que codifica para la proteína RARalpha se encuentra en el cromosoma 17 en la localización 72,074,067 - 72,127,204 (15q24.1).

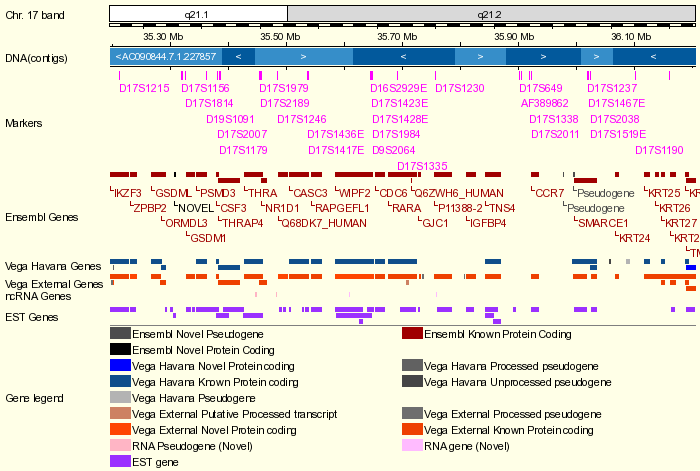
Según la base de datos Ensembl, el gen RARalpha cosnta de 2 transcritos:
| TRANSCRITO 1 | 9 exones | 6 exones CD | 1 exón NCD | 2 exones CD/NCD |
| 11 exones | 9 exones CD | 0 exones NCD | 2 exones CD/NCD |
Al igual que en el caso de PML, al tener 2 transcritos diferentes podemos esperar la generación de 2 isoformas, hecho que corroboran los datos de proteína de Ensembl. Después de buscar en UCSC las isoformas para el gen RARalpha, obtuvimos también isoformas: isoforma a e isoforma b.
Quisimos realizar un alineamiento mediante ClustalW para verificar que tanto las isoformas proporcionadas por Ensembl como las proporcionadas por UCSC eran las mismas. Respecto a la primera isoforma de Ensembl podemos decir que coincide con la isoforma a (UCSC), mientras que la segunda de Ensembl no coincide con la isoforma b, siendo ésta mucho más larga.
Finalmente hemos decidido basarnos de nuevo en la base de datos Ensembl. Si comparamos las isoformas proporcionadas por esta base, observamos que se producen cambios en exones codificantes, por lo que la secuencia de aminoácidos será diferente para cada una de las isoformas, es decir, se alterará la pauta de lectura.