ANÁLISIS COMPUTACIONAL DE LA PROTEÍNA DE FUSIÓN PML-RARalpha
Ainoa Muñoz (ainoa.munoz01@campus.upf.edu) y Soraya Ortiz (soraya.ortiz01@campus.upf.edu)
Facultad de Ciencias de la Salud y de la Vida
Universidad Pompeu Fabra
Resumen
En este trabajo presentamos la caracterización de la estructura exónica, homología, función y expresión de los genes humanos PML y RARalpha, a partir de una secuencia proteíca dada correspondiente a la fusión del producto de ambos genes.
Existe una translocación anómala entre el cromosoma 15 y 17 que da lugar a la leucemia promielocítica aguda. En esta enfermedad se produce una fusión entre un probable factor de transcripción (PML) y el receptor de ácido retinoico (RARalpha), obteniendo como resultado una proteína de fusión:

Este tipo de leucemia presenta tres subtipos (V, L y S) en función del producto de splicing alternativo de las proteínas que se fusionan. Por tanto, según la isoforma de PML (bcr-1, bcr-2 o bcr-3) y de RARalpha que formen la proteína de fusión, se poducirá un subtipo u otro de leucemia, como podemos ver en el siguiente enlace:
subtipos de leucemia promielocítica en función del splicing alternativo en la proteína de fusión
Materiales y métodos
Para la realización de este trabajo,nos hemos basado en la utilización de varias bases de datos para investigar sobre los aspectos generales de cada gen, las funciones que realiza y su mecanismo de acción, así como para extraer información sobre la estructura genética (exones, transcritos, isoformas...). Éstas bases de datos son:




Para llevar a cabo el estudio de homología utilizamos la base de datos de Ensembl y comparamos los resultados mostrados con los obtenidos en BIOMART.
En cuanto al estudio de la expresión de nuestros genes en tejidos, consultamos la base de datos Genome Browser y Gene Sorter (dentro de UCSC), así como las imágenes de VisiGen.
A la hora de caracterizar la región promotora de PML y RARalpha utilizamos un programa en lenguaje de programación PERL, compilado en el editor de texto Emacs. Para corroborar los datos obtenidos al ejecutar el programa, recurrimos a la base de datos PROMO, donde rastrearemos todos los posibles promotores de nuestros genes.
Finalmente, para el estudio de la función de los genes, buscaremos información en la base de datos de Gene Ontology así como artículos en Pubmed.
Resultados y discusión
Caracterización de la estructura genómica de los genes
Gen PML
El gen que codifica para la proteína PML se encuentra en el cromosoma 15 en la localización 72,074,067 - 72,127,204 (15q24.1).

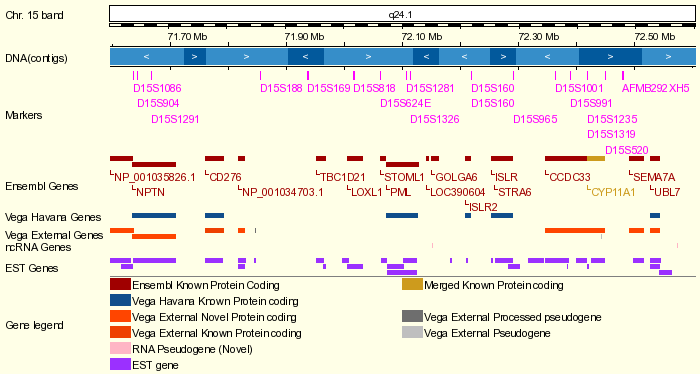
Según la base de datos Ensembl, el gen PML consta de 4 transcritos diferentes:
| TRANSCRITO 1 |
9 exones |
7 exones CD |
0 exones NCD | 2 exones CD/NCD

TRANSCRITO 2
| 8 exones |
7 exones CD |
0 exones NCD | 1 exón CD/NCD

| TRANSCRITO 3 |
7 exones |
6 exones CD |
0 exones NCD | 1 exón CD/NCD

| TRANSCRITO 4 |
5 exones |
4 exones CD |
0 exones NCD | 1 exón CD/NCD

CD = codificante
NCD = no codificante
CD/NCD = porción codificante y porción no codificante
Como podemos observar en las imágenes anteriores, todos los exones de los transcritos son codificantes exceptuando los exones 1 y 9 que poseen una región codificante y una región no codificante. Ésta última parte se encuentra en los extremos de los transcritos, lo que nos lleva a pensar que puede tratarse de regiones UTR.
En un principio, el número de transcritos determina el número máximo de isoformas a las que puede dar lugar un gen. Por lo tanto, según Ensembl, el gen PML da lugar a 4 isoformas correspondientes a cada uno de sus 4 transcritos.
Hemos querido corroborar estos datos con UCSC Genome Browser, la cual nos asocia 11 isoformas a este gen. Los enlaces de 7 de las isoformas descritas por UCSC nos llevan a la misma secuencia proteica, por lo que de 11 isoformas proporcionadas en un principio, sólo obtenemos 3 proteínas diferentes.
Al no coincidir los datos de UCSC y Ensembl miramos en la base de datos UniProt, donde se describen 5 isoformas para el gen PML producidas por splicing alternativo y con los siguientes nombres: PML-1, PML-2,PML-3, PML-3B y PML-X.
Después de tanta variedad de datos, decidimos realizar un alineamiento múltiple mediante ClustalW, comparando las secuencias pproteicas producidas por los 4 transcritos de Ensembl con las 5 isoformas de UniProt.
Los resultados muestran como el transcrito 1 del Ensembl se corresponde con la isoforma PML-1 y el transcrito 2 con la isoforma PML-3. La secuencia de aminoácidos de los otros dos transcritos y de las otras dos isoformas son totalmente diferentes.
Así, en total (consultando estas tres bases de datos) hemos encontrado 7 variantes proteicas del gen PML. Los resultados de las diferentes bases de datos resultan incongruentes y, por tanto, no son concluyentes ya que en un principio sólo podrían existir 4 isoformas diferentes partiendo de la existencia de 4 transcritos. Esto es lo que nos ha llevado a escoger a Ensembl como la base de datos más fiable a la hora de escoger nuestros resultados.
Como cada una de las isoformas derivan de cada uno de los transcritos, las diferencias se deben a splicing alternativo de algunos de los exones, tal y como hemos descrito en las imágenes anteriores en las que se comparaban los exones que coinciden y los que se han eliminado. Debido a que los exones que se modifican son codificantes, se cambia la pauta de lectura y en cada isoforma se obtiene una secuencia aminoacídica diferente:
- ENSP00000268058
- ENSP00000268059
- ENSP00000315434
- ENSP00000353004
Gen RARalpha
El gen que codifica para la proteína RARalpha se encuentra en el cromosoma 17 en la localización 72,074,067 - 72,127,204 (15q24.1).

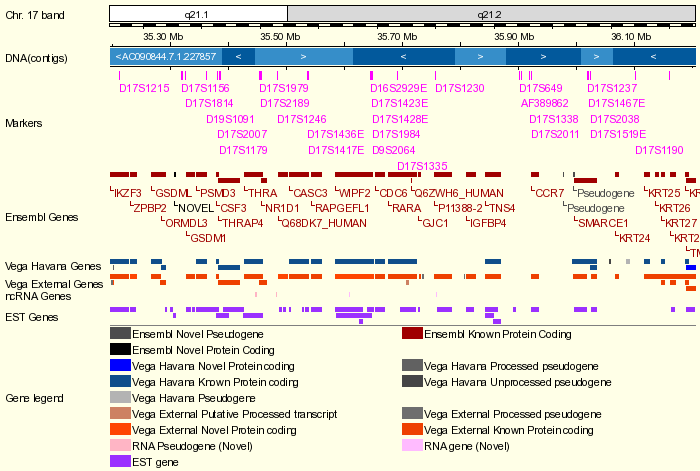
Según la base de datos Ensembl, el gen RARalpha cosnta de 2 transcritos:
| TRANSCRITO 1 |
9 exones |
6 exones CD |
1 exón NCD |
2 exones CD/NCD |

TRANSCRITO 2
| 11 exones |
9 exones CD |
0 exones NCD |
2 exones CD/NCD |

Al igual que en el caso de PML, al tener 2 transcritos diferentes podemos esperar la generación de 2 isoformas, hecho que corroboran los datos de proteína de Ensembl. Después de buscar en UCSC las isoformas para el gen RARalpha, obtuvimos también isoformas: isoforma a e isoforma b.
Quisimos realizar un alineamiento mediante ClustalW para verificar que tanto las isoformas proporcionadas por Ensembl como las proporcionadas por UCSC eran las mismas. Respecto a la primera isoforma de Ensembl podemos decir que coincide con la isoforma a (UCSC), mientras que la segunda de Ensembl no coincide con la isoforma b, siendo ésta mucho más larga.
Finalmente hemos decidido basarnos de nuevo en la base de datos Ensembl. Si comparamos las isoformas proporcionadas por esta base, observamos que se producen cambios en exones codificantes, por lo que la secuencia de aminoácidos será diferente para cada una de las isoformas, es decir, se alterará la pauta de lectura.
Estudio de homología del gen en otras especies
A continuación se muestra una tabla de homología en la que se indican las especies que poseen un gen ortólogo para los genes humanos PML y RARalpha. Los datos mostrados en las tablas han sido obtenidos a partir de las base de datos Ensembl y comparados con los datos de homología de BioMart. Las especies se encuentran ordenadas por % query, que representa la homología de las especies ortólogas respecto a Homo sapiens:
Gen PML
Como podemos observar en la tabla de homología hay un total de 16 especies que poseen un gen ortólogo para PML, lo cual es un indicador de que este gen se encuentra conservado a lo largo del ciclo evolutivo y su función debe ser esencial para muchos organismos. Las especies que comparten una mayor similitud respecto a PML humano son Pan troglodytes y Macaca mulatta, hecho que parece bastante coherente ya que pertenecen al mismo grupo filogenético que Homo sapiens.
A pesar de existir muchas especies homólogas para este gen, la gran mayorí no superan el 70% de similitud lo cual nos puede llevar a pensar que se trate del mismo gen funcional pero que ha acumulado numerosas mutaciones sinónimas.
Observamos como en el caso de Gallus gallus existe homología 1-to-many, lo que quiere decir que se ha producido un proceso de duplicación en el gen PML de esta especie a parte del proceso de especiación que ha dado lugar a este ortólogo. El resto serían homologías 1-to-1, ya que es el hecho más probable al tratarse de una comparación entre una sola copia de cada especie.
Gen RARalpha
La tabla anterior muestra los resultados de homología para el gen RARalpha. Podemos observar un total de 22 especies homológas, que como en el caso del gen PML, puede indicarnos la necesidad de conservación de este gen debido a la importancia de su función.
En este caso, las especies más similares respecto a este gen son de nuevo Macaca mulatta y Pan troglodytes, ya que como hemos dicho anteriormente, pertenecen también a la familia de los homínidos.
También podemos observar una gran cantidad de especies con un porcentaje de homología bastante bajo, en base a la idea de que la funcionalidad del gen se conserva pero presenta modificaciones silentes.
Respecto al gen RARalpha, encontramos 4 especies (Danio rerio, Takifugu rubripes, Ciona savignyi y Ciona intestinalis) que presentan un tipo de homología 1-to-many, que por tanto, tendrán varias copias para el gen RARalpha en su genoma.
Caracterización de la expresión de los genes
Con tal de observar la expresión; de nuestros genes en los diferentes tejidos del cuerpo, hemos analizado los resultados mostrados por UCSC (Gene Browser y Gene Sorter). En esta base de datos se muestran ensayos de microarrays realizados con diferentes marcadores. Las imágenes que se muestran en este apartado son los resultados de estos ensayos, en los que las diferentes tonalidades de rojo haran referéncia a los tejidos donde habrá una alta expresión, mientras que los tonos de verde corresponderían a una baja expresión.
Gen PML
En la siguiente imagen proporcionada por Gene Sorter (UCSC) podemos ver los resultados de microarray(Atlas 2) en una versión simplificada en cuanto a los tejidos analizados.

En esta imagen podemos ver una alta expresión de PML en el hígado. También se observa expresión aunque en menor grado en todo el cerebro.
Si estudiamos la expresión en una selección más amplia de tejidos y usando otros marcadores para realizar los microarrays, podemos observar una alta expresión en músculo esquelético, útero, bazo y corazón además de en los tejidos ya mencionados en la primera imagen. En menor expresión se encuentra además en órganos del sistema urogenital (tanto masculino como femenino) y en regiones del estómago y del intestino.
Gen RARalpha
En la siguiente simplificada de Gene Sorter (microarray Atlas 2) observamos una alta expresión en pulmones, testículos y en menor grado en amígdala, piel y cerebro (tanto fetal como adulto).

Para el gen RARalpha en una versión más extendida de tejidos podemos ver una alta expresión sobretodo en la sangre, así como en las células sanguíneas. También hay una gran expresión en pulmón, diafragma, vesícula biliar, ovario, bazo, páncreas y en algunas estructuras cerebrales. A pesar de que la expresión más relevante se encontraría en los tejidos ya mencionados, también se expresa en otros, como podemos ver en las siguientes imágenes.
Caracterización de la región promotora de los genes
Para la caracterización de la secuencia utilizaremos un programa realizado en Perl y después, compararemos los resultados obtenidos en nuestro programa con la base de datos de la web PROMO.
Programa identificacio_de_lufs.pl
La secuencia promotora de nuestros genes la obtenemos a partir de su secuencia nucleotídica proporcionada por la base de datos UCSC. En el enlace correspondiente a la información del gen, descargaremos la secuencia genómica, seleccionando la opción de presentarnos la secuencia promotora (1000 bases upstream). Una vez obtenida, nos quedaremos con los 1100 primeros nucleótidos (1000 de la región promotora + 100 primeros nucleótidos).
Las secuencias promotoras obtenidas para nuestros genes son las siguientes:
Secuencia promotora del gen PML
Secuencia promotora del gen RARalpha
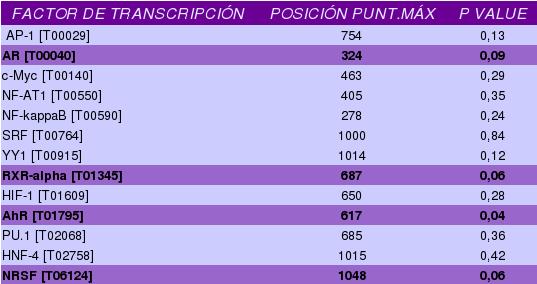
A continuación mostramos los resultados obtenidos mediante nuestro programa de indentificación de sitios de unión a promotor en Perl para cada uno de los genes:
Gen PML
Gen RARalpha
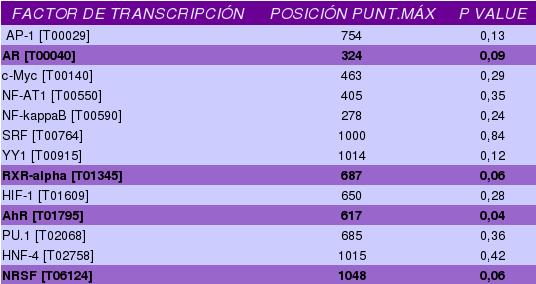
En los siguientes hipervínculos podemos ver los resultados obtenidos mediante la base de datos PROMO tanto para un porcentaje de similitud del 5% como del 15%.
Gen PML
PML 5%
PML 15%
Gen RARalpha
RARalpha 5%
RARalpha 15%
Como podemos observar la comparación entre nuestros resultados y los de PROMO apenas coinciden. En las tablas se encuentran marcados los factores de transcripción que aparecen tanto en las matrices proporcionadas para el programa como en PROMO, aunque el archivo de matrices inicial parece no estar bien adquirido de la base de datos PROMO (no coinciden los datos).
Estudio de la función de los genes PML y RARalpha
Gen PML
El gen PML es el gen que codifica para la proteína PML,que consta de 882 AA y tiene un peso molecular de 97,5 kDa. Este gen también es conocido con los nombres TRIM19(tripartite motif-containing protein 19) o RING finger protein 71.
Se localiza en el núcleo celular, en una especie de corpúsculos llamados cuerpos nucleares PML y su función principales la supresión del crecimiento celular,además posee muchas otras funciones relacionadas con la regulación de genes e implicación en las vias de supresión tumoral y, por tanto, de la inhibición del crecimiento celular:
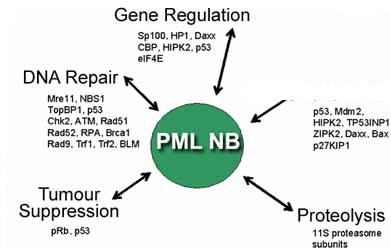
La proteína PML se regula mediante ubiquitinación, mediada por SIAH1 o SIAH2 que le proporcionarán la señal precisa para su degradación en el proteasoma.
PML presenta los siguientes dominios, de los cuales destaca la presencia de un Zn finger y dos B-Box:

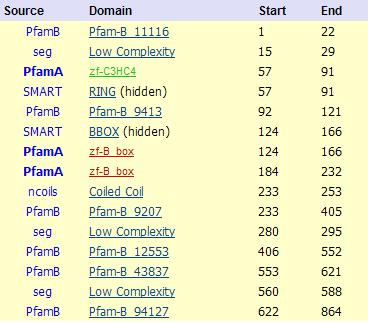
Se define como un probable factor de transcripción que puede jugar un papel importante en el reclutamiento de ELF4, interaccionando con su extremo C-terminal, dentro de los cuerpos nucleares PML.Elf4 o MEF (myeloid elf-1-like factor) es un miembro de la familia de los factores de transcripción ETS y se encarga de la up-regulation de la expresión basal del gen de la lisozima en celulas epiteliales, gracias a la unión con el factor PML.
El gen PML realiza interacciones con varios factores, tales como SIRT1, TOPBP1, TRIM27 y TRIM69, además de interaccionar con la proteína Z del virus Lassa.
También se ha comprobado como PML es capaz de interaccionar con una proteína nucleolar conocida como Mdm2, bajo condiciones de stress celular y/o daño al DNA. Diferentes ensayos de inmunofluorescéncia muestran como la interacción se produce in vivo además de in vitro, donde se ha observado como PML es capaz de formar complejos junto Mdm2 y p53, sugiriendo que la unión de PML a Mdm2 favorece a PML para la activación y estabilización de p53 en respuesta al stress celular.
PML colocaliza a su vez con la forma no fosforilada de retinoblastoma, estabilizando la via de acción molecular implicada en el crecimiento celular:
PML pathway
Gen RARalpha
El gen RARalpha codifica para un subtipo de receptor de ácido retinoico (receptor de ácido retinoico alpha),que consta de 462 AA y tiene un peso molecular de 50,7 kDa.Este receptor es homologo de otros tipos de receptores nucleares como los receptores de hormonas esteroideas, de hormonas tiroideas o el receptor de vitamina D.
El ácido retinoico es un metabolito que tiene un importante efecto sobre el desarrollo de los vertebrados, actuando como morfógeno y potente teratógeno. Éste es el ligando de RARalpha y se une a él con una alta afinidad.
Como función principal, este receptor controla la función celular partiendo de la regulación directa del la expresión génica,siguiendo los mismos mecanismos moleculares implicados en el desarrollo embriológico, diferenciación y crecimiento de celulas tumorales anteriormente descritos en otros miembros de la familia de este receptor nuclear.
Su regulación tiene lugar por procesos de fosforilación (se encuentra fosforilado durante el ciclo celular). La Ser77 es un aminoácido clave para su papel como regulador transcripcional.
Este receptor esta compuesto por tres dominios fundamentales:un extremo N-terminal de función moduladora, un dominio de unión a DNA y un dominio C-terminal encargado de la unión de esteroides.Podemos ver sus dominios con mas detaye en la siguiente imágen:

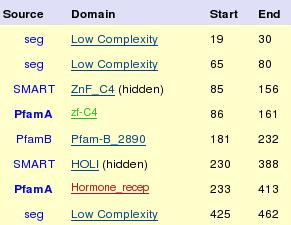
RARalpha interacciona con CDK7 por similaridad, además de unirse y coactivar a NCOA3 y NCOA6, liderando así un fuerte incremento de la transcripción de genes diana en este proceso.Interacciona también con NCOA7 inducido por su ligando (ácido retinoico).
Cuando RARalpha se encuentra fusionado con PML, da lugar a diferenciación hematopoyética (leucemia promielocítica), ya que en este caso, la proteína de fusión PML-RARalpha tiene la capacidad de inactivar genes diana mediante el reclutamiento de histonas deacetilasas (HDACs) y actividad metiltransferasa. PML-RARalpha secuestra los complejos HDAC-MBD1 necesarios para el correcto funcionamiento de la vía de generación de células hematopoyéticas, contribuyendo así al establecimiento y mantenimiento de un estado silente en la cromatina:
PML-RARalpha vs. HDACs
RARalpha, además de participar en esta aberración cromosó, tambié se encuentra en otras translocaciones junto a los genes ZBTB16/PLZF o NPM dando lugar a distintas patologías.
Conclusiones
Después de haber realizado este trabajo podemos concluir que la utilización de bases de datos para estudiar un gen determinado resulta bastante contradictorio, ya que hay incongruencias en los datos proporcionados por éstas.>
Referencias
Kurki S, Latonen L, Laiho M. Cellular stress and DNA damage invoke temporally distinct Mdm2, p53 and PML complexes and damage-specific nuclear relocalization.J Cell Sci. 2003 Oct 1;116(Pt 19):3917-25.
Bernardi R, Scaglioni PP, Bergmann S, Horn HF, Vousden KH, Pandolfi PP.PML regulates p53 stability by sequestering Mdm2 to the nucleolus. Nat Cell Biol. 2004 Jul;6(7):665-72.
Alcalay M, Tomassoni L, Colombo E, Stoldt S, Grignani F, Fagioli M, Szekely L, Helin K, Pelicci PG.The promyelocytic leukemia gene product (PML) forms stable complexes with the retinoblastoma protein.Mol Cell Biol. 1998 Feb;18(2):1084-93.
Pearson M, Carbone R, Sebastiani C, Cioce M, Fagioli M, Saito S, Higashimoto Y, Appella E, Minucci S, Pandolfi PP, Pelicci PG.PML regulates p53 acetylation and premature senescence induced by oncogenic Ras.Nature. 2000 Jul 13;406(6792):207-10.
Suico MA, Yoshida H, Seki Y, Uchikawa T, Lu Z, Shuto T, Matsuzaki K, Nakao M, Li JD, Kai H.Myeloid Elf-1-like factor, an ETS transcription factor, up-regulates lysozyme transcription in epithelial cells through interaction with promyelocytic leukemia protein.J Biol Chem. 2004 Apr 30;279(18):19091-8.
Villa R, Morey L, Raker VA, Buschbeck M, Gutierrez A, De Santis F, Corsaro M, Varas F, Bossi D, Minucci S, Pelicci PG, Di Croce L.The methyl-CpG binding protein MBD1 is required for PML-RARalpha function. Proc Natl Acad Sci U S A. 2006 Jan 31;103(5):1400-5.
Petkovich M, Brand NJ, Krust A, Chambon P.A human retinoic acid receptor which belongs to the family of nuclear receptors.Nature. 1987 Dec 3-9;330(6147):444-50















