
El nostre treball es centra en la proteïna de fusió AML1-MTG8. Aquesta es genera a partir d'una translocació t(8;21)(q22;q22) que és una de les translocacions cromosòmiques més comuna en la leucèmia mieloide aguda. El resultat d'aquesta translocació és una fusió entre la regió N-terminal de la proteïna AML1 (codificada pel gen RUNX1), i la regió C-terminal d'un correpresor transcripcional anomenat MTG8 (codificat pel gen RUNX1T1).[2][3][4]
De cadascun dels gens implicats en aquesta translocació hem determinat l'estructura genòmica, l'homologia en altres espècies, l'expressió, els possibles factors de transcripció que s'uneixen a la regió promotora, i per últim la funció.
La proteïna de fusió AML1-MTG8 provoca que AML1 canviï la seva funció d'activador transcripcional per una de repressor transcricpional de les seves dianes. Per estudiar els efectes d'aquesta proteïna de fusió s'ha expressat exògenament en un tipus cel·lular concret (32D) i s'ha vist que afecta en la diferenciació granulocítica i en la proliferació cel·lular. [1]
La seqüència de la proteïna de fusió AML1-MTG8, està formada per 177 aminoàcids corresponents a AML1 i 574 aminoàcids corresponents a MTG8.
En la proteïna de fusió hem pogut veure que hi ha una asparagina que uneix les dues proteïnes. Aquest aminoàcid està codificat per l'últim nucleòtid de l'exò 3 del gen RUNX1 junt amb els 2 primers nucleòtids de l'exò 2 del gen RUNX1T1, els quals formen el codó AAT.
Clica aquí per veure més informació sobre les seqüències genòmiques:
 |
| Seqüència protèica de AML1-MTG8 |
El gen codificant per AML1, és el gen RUNX1 (run-related transcription factor 1 (acute myeloid leukemia 1; AML1 oncogene)) que es troba en el cromosoma 21 en el braç llarg q22.12.
El gen codificant per MTG8, és el gen RUNX1T1 (run-related transcription factor 1; translocated to, 1 (cyclin D-related)) que es troba en el cromosoma 8 en el braç llarg q21.3.
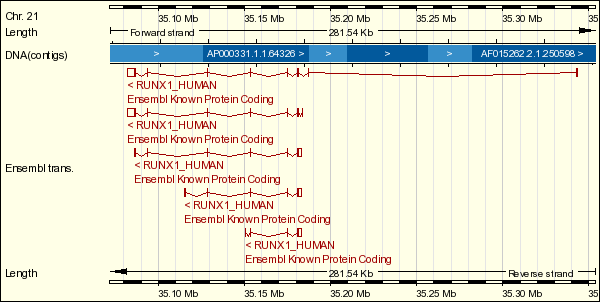
Si ens centrem primerament amb el gen RUNX1 observem com aquest té un total de 8 exons, dels quals només 3 apareixen a la nostra proteïna de fusió que són l'exò ENSE00001380483, el ENSE00001247303 i el ENSE00001044058. La resta d'exons no apareixen. Concretament si entrem en el trànscrit del gen RUNX1 que dóna lloc a la proteïna AML1 en Ensembl, obtenim la següent seqüència.
Clica aquí per veure la seqüència complerta del trànscrit:
A més cal destacar que si fem un BLAST en UCSC es mostren els exons del gen RUNX1 que codifiquen per una part de la nostra proteïna de fusió. Aquests exons els hem identificat en la seqüència del trànscrit anterior.
El gen RUNX1 té associats 5 trànscrits (si ens basem en la base de dades d'Ensembl). Però si ens fixem en la base de dades Uniprot/Swissprot hem trobat 11 isoformes diferents del gen RUNX1. Vist que aquestes dades no concorden, ens hem basat en Ensembl per fer l'estudi dels trànscrits i les isoformes. A més, cal destacar que Ensembl és una base de dades molt més restrictiva, i per tant més fiable.
En aquest cas destaquem que els trànscrits inclouen conjunts d'exons diferents degut a l'splicing alternatiu.
| Trànscrits: RUNX1_HUMAN | Nombre d'exons | ENST00000300305 | 8 |
| ENST00000325074 | 7 |
| ENST00000342083 | 4 |
| ENST00000344691 | 8 |
| ENST00000358356 | 5 |
Si comparem els exons dels diferents trànscrits observem que hi ha exons que són comuns en tots els trànscrits, i a més aquests es troben en la nostra proteïna de fusió (mirar l'alineament fet amb ClustalW de la regió marcada en verd corresponent a la part AML1 de la nostra proteïna). També hi ha exons que difereixen entre els trànscrits, que són codificants o mig codificants (no n'hi ha cap de no codificant per complert), i fan que hi hagi 5 isoformes diferents.
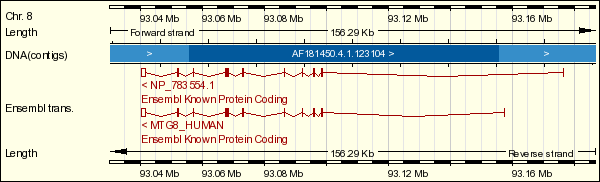
Si ens fixem en el gen RUNX1T1 veiem que té 11 exons en total, i tots ells apareixen a la nostra proteïna de fusió excepte el primer exò. El trànscrit del gen RUNX1T1 que dóna lloc a la proteïna MTG8 té la següent seqüència.
Clica aquí per veure la seqüència complerta del trànscrit:
A més cal destacar que si fem un BLAST en UCSC es mostren els exons del gen RUNX1T1 que codifiquen per una part de la nostra proteïna de fusió. Aquests exons els hem identificat en la seqüència del trànscrit anterior.
El gen RUNX1T1 té associats 2 trànscrits. En aquest cas tots dos trànscrits tenen 11 exons. En un dels trànscrits tots els exons són codificants excepte el 1 i el 11 que són mig codificants perquè contenen regions UTR. I en l'altre trànscrit els exons del 3 al 10 són codificants, el 2 i el 11 són mig codificants, i el primer és no codificant. Per tant, a causa de l'splicing alternatiu tenen seqüències codificants diferents i en conseqüència donen lloc a dues isoformes que són MTG8-A i MTG8-B.
Donat que en aquest gen només té associats 2 trànscrits i dues isoformes, és molt més senzill comparar-les a simple vista i no cal realitzar cap alineament múltiple. De manera que tal i com s'observa a continuació hem comparat visualment els diferents exons dels trànscrits i les diferents seqüències aminoacídiques a les que donen lloc (dues isoformes)

Els dos trànscrits no tenen en comú els exons 1 i 2. Però tenen en comú tota la resta d'exons (del exò 3 al 11).
La isoforma que produeix el primer trànscrit (MTG8_HUMAN) és:
 |
La isoforma que produeix el segon trànscrit (NP_783554.1) és:
 |
Així doncs, observem que la diferència entre les dues isoformes generades a partir dels dos trànscrits del gen RUNX1T1, és deguda al fet que la primera isoforma inclou dos exons codificants (exò 1 i 2) i la segona inclou un exò no codificant (exò 1) i un exò codificant (exò 2). Això però no afecta a la pauta de lectura, ja que a partir del tercer exò es correspon de manera exacta entre les dues isoformes.
En aquest aparatat amb la seqüència codificant (cds) de la proteïna de fusió, hem obitngut únicament gens ortòlegs de diferents espècies de vertebrats (NCBI).
 |
D'altra banda hem buscat a partir dels dos gens RUNX1 i RUNX1T1 per separat, els gens ortòlegs d'altres espècies i el seu percentatge d'identitat obtingut a través d'un alineament global fet amb ClustalW. Els percentatges d'identitat són respecte els gens d'humà de la nostra proteïna de fusió (target % ID). Aquests percentatges són els següents:
| Espècie | RUNX1 | RUNX1T1 |
| Aedes aegypti | 52% | 29% |
| Anopheles gambiae | 48% | 29% |
Bos taurus 
|
98% | 99% |
| Caenorhabditis elegans | 29% | |
Canis familiaris 
|
97% | 92% |
| Cavia porcellus | 94% | |
| Ciona intestinalis | 27% | |
| Ciona svignyi | 64% | |
| Dario rerio | 88% | |
| Dasypus novemcinctus | 46% | 65% |
| Drosophila melanogaster | 31% | 20% |
| Echinops telfairi | 86% | 76% |
| Erinceus europaeus | 77% | |
Felis catus 
|
89% | 99% |
Gallus gallus 
|
77% | 97% |
| Gasterosteus aculeatus | 48% | 83% |
| Loxodonta africana | 74% | 88% |
Macaca mulatta 
|
99% | 99% |
Monodelphis domestica 
|
90% | 97% |
Mus musculus 
|
95% | 97% |
| Ornithorhynchus anatinus | 65% | 95% |
Oryctolagus cuniculus 
|
98% | 94% |
| Oryzias latipes | 56% | 80% |
Pan troglodytes 
|
100% | 100% |
| Rattus norvegicus | 94% | 86% |
| Takifugu rubripes | 55% | 85% |
| Tetraodon nigroviridis | 80% | 80% |
Tupaia belangeri 
|
77% | 98% |
| Xenopus tropicalis | 84% | 96% |
Els percentatges marcats en verd mostren una elevada similitud entre el gen humà i el gen ortòleg corresponent. D'altra banda, els percentatges marcats en vermell mostren una baixa similitud.
El gen RUNX1 té el següent patró d'expressió:
| Teixit o Tipus cel·lular | Timus | BM-CD33+ mieloide | BM-CD105+ endotelial | BM-CD34+ | Cèl·lules dendrítiques | Monòcits | Cèl·lules NK | Cèl·lules T | Cèl·lules B | Leucèmia limfoblàstica | Adenocarcinoma colorectal | Múscul llis | Tràquea | Pulmó |
| RUNX1 |

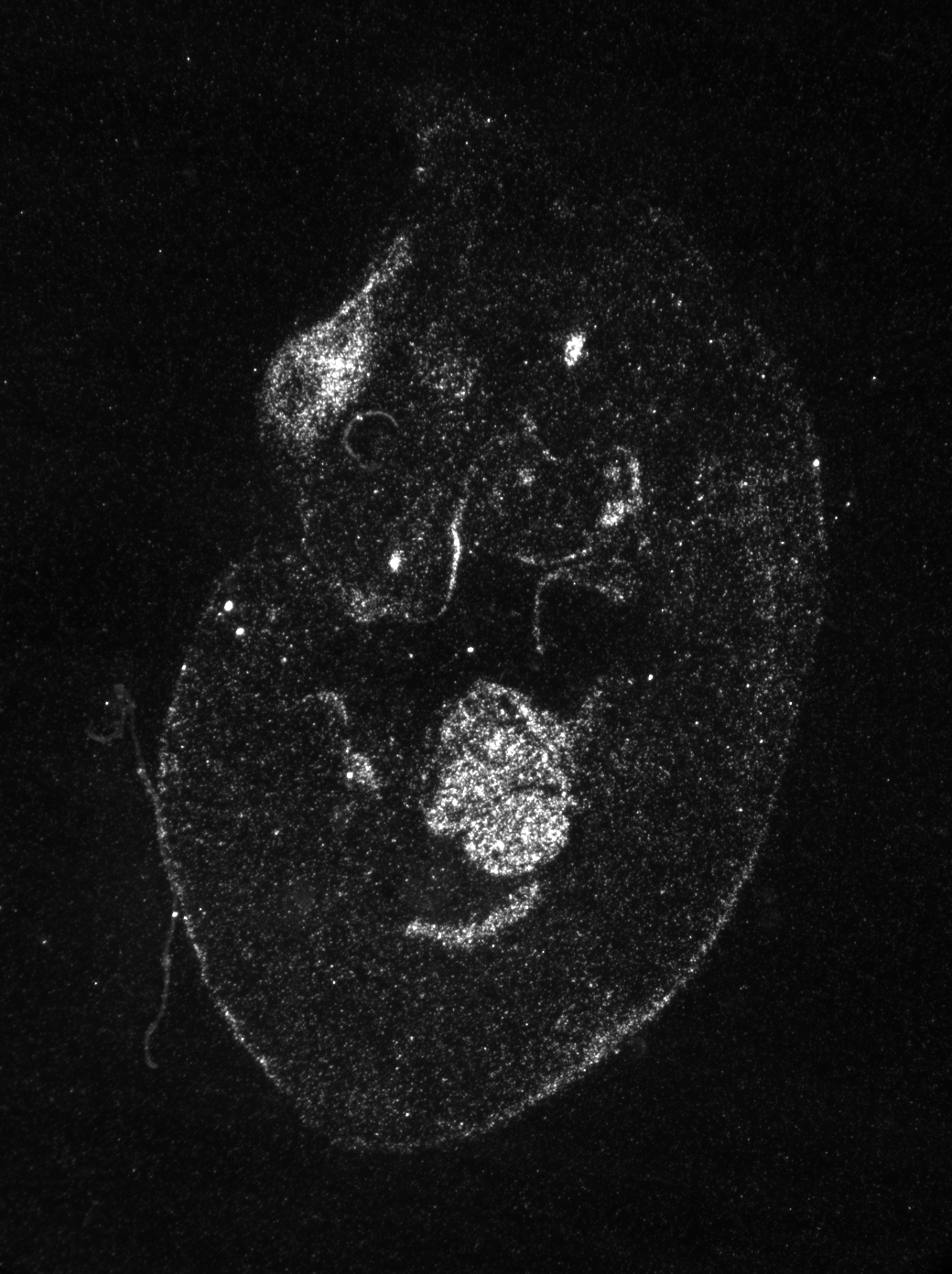
|
Marcatge del gen RUNX1 en embrió de ratolí |
Si ara ens basem en el gen RUNX1T1, podem observar el següent patró d'expressió:
| Teixit o tipus cel·lular | Cervell fetal | Cerebel | Monòcits | Cèl·lules NK | Cèl·lules B | Limfoblasts | Adenocarcinoma colorectal | Úter | RUNX1T1 |

La regió promotora del gen RUNX1 (l'hem agafat del trànscrit ENST00000344691):
 |
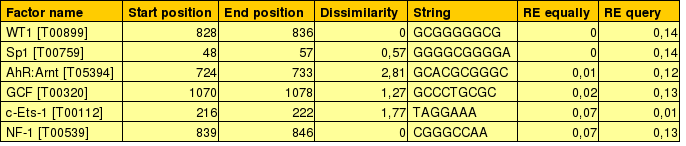
A partir d'aquí hem buscat amb el programa PROMO els FTs més probables que s'uneixen a la regió promotora del gen RUNX1, que són:
La regió promotora del gen RUNX1T1 (l'hem agafat del trànscrit ENST00000265814):
 |
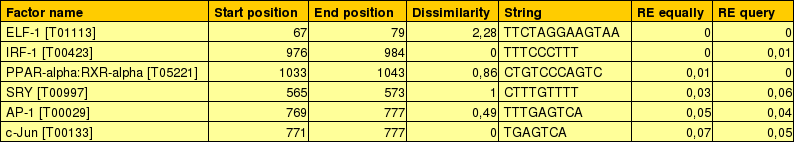
A més a més, igual que el cas anterior, també hem buscat amb PROMO el conjunt de FTs més probables que s'uneixen a la regió promotora del gen RUNX1T1 que són:
D'altra banda, mitjançant el desenvolupament d'un programa Perl hem realitzat la mateixa cerca dels possibles factors de transcripció que es podrien unir als promotors de RUNX1 i RUNX1T1.
Per poder desenvolupar aquest programa, ho hem fet amb quatre passos diferents. I per executar-lo cal introduir-li primer una de les dues seqüències promotores, i a continuació un seguit de matrius (aquestes corresponen a diferents factors de transcripció).
Programa PerlEn executar el programa amb el fitxer de matrius que se'ns ha proporcionat i les seqüències promotores dels gens, s'obtenen un seguit de FTs, dels quals, els més significatius (score màxim més elevats, i p-value més baixos) estan resumits en les següents taules:
| Factors de Transcripció | Score màxim | Posició | p-value |
| NF-AT1 [T00550] | 6.727 | 712 | 0.2 |
| YY1 [T00915] | 6.197 | 941 | 0.6 |
| HIF-1 [T01609] | 8.950 | 970 | 0.05 |
| AhR [T01795] | 6.666 | 700 | 0.31 |
| PU.1 [T02068] | 7.256 | 378 | 0.28 |
| Factors de Transcripció | Score màxim | Posició | p-value |
| AP-1 [T00029] | 9.262 | 771 | 0.08 |
| AR [T00040] | 8.185 | 528 | 0.0 |
| NF-AT1 [T00550] | 7.410 | 348 | 0.13 |
| YY1 [T00915] | 5.941 | 832 | 0.57 |
| RXR-alpha [T01345] | 4.974 | 553 | 0.81 | PU.1 [T02068] | 6.776 | 291 | 0.14 |
| HNF-4 [T02758] | 10.216 | 194 | 0.01 |
En aquestes dues taules, els FTs marcats en nergeta, són els més probables a unir-se a les regions promotores d'ambdós gens.
| Funcions moleculars del gen RUNX1 | Descripció |
| ATP binding | Domini que interacciona selectivament amb l'ATP |
| Protein binding | La proteïna AML1 interacciona amb les proteïnes NERF1 i NERF2. Amb NERF2 es provoca la transactivació d'un promotor (Blk), i amb NERF1 fa l'efecte contrari (repressió). AML1 controla molts dels promotors de gens de cèl·lules hematopoiètiques [5][6][7] | Transcription factor activity | Factor de transcripció capaç d'unir-se a diferents enhancers i promotors (d'un receptor de cèl·lules T, d'un virus de leucèmia murina.etc...). La subunitat alfa d'aquest FT s'uneix al DNA i té un paper important en la regulació dels gens essencials d'hematopoiesi [8] |
| Transcriptional activator activity | AML1 és un factor transcripcional activador [5][6] |
Processos biològics:
| Funcions moleculars del gen RUNX1T1 | Descripció |
| Transcription factor activity | S'ha vist que és un FT regulador que s'expressa al teixit adipós, i que segurament juga un paper molt important en el desenvolupament d'adipòcits o en el control metabòlic [9] |
La caracterització de l'estructura genòmica d'ambdós gens ha estat complicada de realitzar. Perquè la informació disponible en les diferents bases de dades (Ensembl, UCSC, NCBI, UniProt,...) de vegades no coincideix (nombre de trànscrits, isoformes,...). Per això ens hem basat en una única base de dades (Ensembl) per fer aquest punt del treball.
En el desenvolupament d'aquest punt, hem comprovat com l'splicing alternatiu afecta tant al gen RUNX1 com al gen RUNX1T1, generant diferents trànscrits que codifiquen per diferents isoformes, però sempre conservant el marc de lectura. Aquest fet podria estar relacionat amb la importància de mantenir aquells exons que es troben més conservats, i que per tan podrien tenir un paper significatiu en la funció de la proteïna.
El fet d'obtenir gens ortòlegs de la seqüència codificant de AML1-MTG8 només en organismes vertebrats ens pot indicar que la translocació que provoca l'aparició de la nostra proteïna de fusió probablement només s'ha estudiat en vertebrats, ja que es tracta d'una proteïna que es troba associada a un tipus de malaltia (leucèmia mieloide).
A diferència del cds de AML1-MTG8, els gens RUNX1 i RUNX1T1 es troben en organismes invertebrats. Això ens pot indicar que són gens força antics. Els dos gens també es troben en vertebrats amb un percentatge d'identitat força alt. I per tant, considerem que encara no ha passat prou temps com perquè s'acumulin mutacions.
En produir-se la translocació cromosòmica t(8;21) es produeix la fusió dels dos gens. Aquest fet provoca l'aparició de la leucèmia mieloide aguda, que és un tipus de càncer produït en les cèl·lules de la línia mieloide dels leucòcits. Es caracteritza pel creixement descontrolat de cèl·lules leucèmiques anormals que s'acumulen a la medul·la òssia [10]. De manera que podriem pensar que aquestes cèl·lules anormals poden expressar la nostra proteïna de fusió.
Podriem hipotetitzar que la translocació també altera a l'expressió de RUNX1, perquè els tipus cel·lulars diferenciats on s'expressa es veurien diminuits.
Si comparem els resultats dels possibles FTs que s'uneixen a les regions promotores dels dos gens obitnguts amb PROMO i amb el programa Perl, veiem que hi ha molt poca coincidència. Únicament hi ha un factor de transcripció (AP1) que coincideix en el cas del gen RUNX1T1. Això pot ser degut a que s'utilitzen matrius diferents en cada cas.
Els FTs obtinguts amb PROMO són força significatius, però hauríem de determinar si aquests factors són específics o no per a la nostra regió promotora.
Vistes les funcions dels gens involucrats en la translocació t(8;21), hem volgut predir quina és la funció de AML1-MTG8 generada per aquesta translocació.
Si ens fixem en els dominis que presenta la nostra proteïna de fusió:

Veiem que es conserva el domini Runt de la proteïna AML1 (RUNX1), sabem que aquest domini permet que AML1 s'uneixi al DNA i reguli els seus gens diana. De manera que hipotetitzem que la nostra proteïna de fusió manté aquesta característica. Però amb la diferència que la funció passa de ser activadora a represora transcripcional.
Destaquem que els tres dominis restants es consereven de la proteïna MTG8 (RUNX1T1).