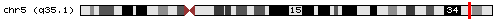
El gen NPM1 es troba situat al cromosoma 5, concretament a la regió q35 (orientació telomèrica) i prensenta un total de 12 exons. Codifica per la nucleofosmina, una fosfoproteïna nuclear, localitzada sobretot a la regió granular del nuclèol, que juga un paper important en l'assemblatge preribosomal, en el transport de ribonucleoproteïnes entre compartiments cel·lulars, en el tràfic citoplasmàtic-nuclear, en la duplicació del centrosoma i en la regulació de p53.
Figura 1: Representació del gen NPM1 al cromosoma 5
El nombre de transcrits associats a aquest gen presenta algunes discrepàncies entre bases de dades diferents: Genome Browser, Ensembl i Gen bank.
Segons la informació trobada al Genome browser i al Gen Bank, el gen NPM1 presenta 3 transcrits, cadascun d'ells amb una estructura exònica diferent. Per contra, en la base de dades de l'Ensembl només consten 2 transcrits. Així doncs, l'anàlisi s'ha fet a partir del Genome Browser, considerada com a més significativa.
El TRANSCRIT 1, corresponent a la 1a isoforma, consta de 12 exons, tot i que no tots són codificants. Representa la primera nucleofosmina descrita, a càrrec de Chan et al. al 1989.
El primer exó és no codificant en la seva totalitat.
El segon exó presenta una primera regió no codificant de 16 nucleòtids (en continuació del primer exó), i una segona regió codificant de 58 nucleòtids. La regió no codificant que abarca aquests dos exons correspon a la regió 5'UTR.
De l'exó 3 a l'exó 11, ambdós incluits, són tots codificants.
L'últim exó presenta els 59 primers nucleòtids codificants i els restants 318 no codificants, els quals representaran la regió 3'UTR.
El TRANSCRIT 2, corresponent a la 2a isoforma, consta d'11 exons, i de la mateixa manera que el transcrit 1, no tots són codificants.
Aquest transcrit presenta un exó menys, concretament l'exó número 9, que es perd pel fenòmen d'splicing alternatiu. Aquest és un fet puntual, ja que la resta de la seqüència es conserva igual com també la pauta de lectura, que es manté intacta.
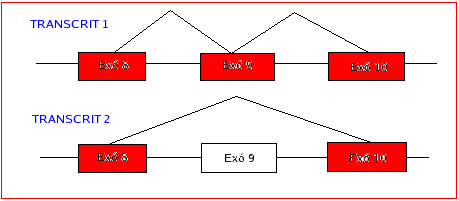
Figura 2: Representació de l'splicing alternatiu que pateix l'exó 9
Aquests dos primers transcrits són també presents a la base de dades de l'ensembl però amb l'existència d'alguna discrepància respecte el Genome Browser i el Gen Bank. Mentre que la regió exònica codificant i la regió 3'UTR coincideix en totes 3 bases, no passa el mateix amb la regió 5'UTR, la qual és totalment diferent entre l'ensembl i les restants. Aquest fet podria explicar-se considerant la possibilitat de que pot haver-hi més d'un inici de la transcripció per a la seqüència d'aquest gen.
La nucleofosmina resultant d'aquest transcrit consta de 265 aminoàcids i de 29,46 Kda de pes molecular.
El TRANSCRIT 3, correspon a la 3a isoforma, última en ser descrita, a càrrec de Dalenc et al. al 2002, i consta d'11 exons, alguns d'ells també no codificants.
Observant-la veiem que els 10 primers exons són idèntics al transcrit 1, mentre que l'exò 11 és totalment diferent. Consta de 329 nucleòtids, dels quals els 9 primers són codificants i els 320 finals són no codificants. Així doncs, aquests últims representen la regió 3'UTR. L'explicació trobada per aquesta diferència és purament funcional, com es descriurà en l'últim apartat.
En aquest cas, la nucleofosmina presenta 259 aminoàcids i pesa 28,4 kDa.
El gen ALK es troba situat al cromosoma 2, concretament a la regió p23 (orientació centromèrica) i presenta un total de 30 exons. Codifica per una glicoproteïna tirosina kinasa amb funció de receptor cel·lular associat a la membrana (molt similar als de la subfamília dels receptors d'insulina), l'expressió del qual està limitada a les cèl·lules nervioses. L'explicació d'aquesta localització específica es troba en el fet que la proteïna juga un paper important en el desenvolupament de l'encèfal i exerceix els seus efectes en neurones específiques del sistema nerviós.

Figura 3: Representació del gen ALK al cromosoma 2
Igual com en el gen NPM1, existeixen diferències entre les bases de dades de l'Ensembl, el Genome Browser i el Gen Bank. En aquest cas, tan sols en la primera trobem més d'un transcrit. Tot i tenir en compte la base de dades del Genome Browser també s'ha volgut analitzar els altres transcrits que apareixen únicament a l'Ensembl.
Segons l'Ensembl, el gen ALK porta associats 3 transcrits, dos dels qual són exactament iguals. Això s'ha volgut corroborar alineant (BLAST) els dos transcrits iguals per verificar aquesta total equivalència. Aquest fet no és massa lògic ja que l'existència de diversos transcrits acostuma a anar associada a la possibilitat de que d'un mateix gen es puguin expressar diferents isoformes d'una proteïa, i per tant s'analitzaran només aquests 2 transcrits.
En el TRANSCRIT 1 s'han trobat 30 exons (tots codificants), mentre que en el TRANSCRIT 2 se n'hi observen 29 (també tots codificants).
La diferència rau en què en el transcrit 1 hi ha una part intrònica de 5 nucleòtids que es codifica donant el resultat de 30 exons. A més a més l'exó 8 d'aquest transcrit presenta 96 nucleòtids, mentre que el transcrit 2 en mostra 101. Al ser el mateix nombre de nucleòtids, és a dir 5, que difereixen entre els exons 8 de cada transcrit, podem assegurar que la pauta de lectura es conserva. Tot i així, els 5 nucleòtids que formen l'exó 9 del transcrit 1 no són els mateixos que té de més l'exó 8 del transcrit 2, sinó que com s'ha dit, corresponen a part intrònica. L'esquema corresponent al procés que ha tingut lloc és el següent:
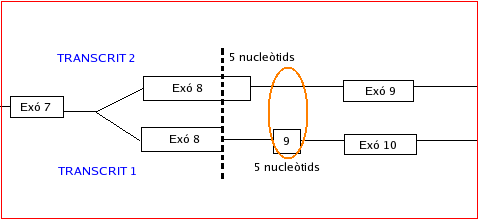
Figura 4: Esquema que representa l'splicing explicat
Per altra banda, l'únic transcrit trobat tan al Genome Browser com al Gene Bank coincideix amb el segon transcrit de l'Ensembl, que presenta 29 exons. La petita discrepància es troba en la presència de regions no codificants. Aquest transcrit del Genome Browser es pot observar en aquest enllaç, on s'hi poden observar els 29 exons codificants i les regions 5'UTR formada de 907 nucleòtids i, 3'UTR formada per 448 nucleòtids.
La formació de la proteïna de fusió és conseqüència d'una translocació entre les dues regions dels dos cromosomes (t(2;5)(p23;q35)), com es pot observar en la següent imatge:
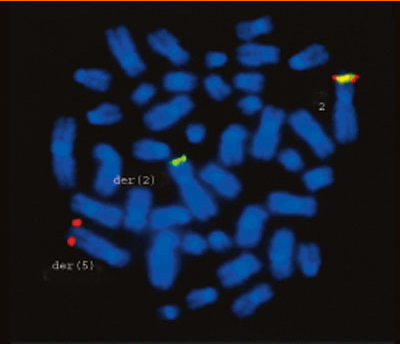
Figura 5: Hibridació in situ de fluorescè que mostra el cariotip anòmal.
El nou gen que es crea conté a la part 5' els primers 4 exons corresponents a la proteïna NPM1, que codifiquen per 116 aminoàcids que representaran l'extrem N-terminal de la proteïna. A partir d'aquí s'hi enganxen els 10 últims exons de la proteïna ALK, que codificaran per una seqüència de 563 aminoàcids que correspondrà a l'extrem C-terminal. Per tant, la proteïna resultant conté un total de 680 aminoàcids, amb un pes molecular de 80 kDa.
La unió d'exons es fa de manera totalment precisa, sense que es produeixi la pèrdua de cap nucleòtid. A nivell proteïc, aquesta fusió involucra tota la porció citoplasmàtica d'ALK, que inclou el seu domini catalític, i el segment amino terminal de NPM1, que és essencial com a domini d'oligomerització i responsable tant de l'activació constitutiva d'ALK com del seu canvi de localització. Així doncs, els dominis extracel·lular i transmembrana d'ALK no hi són presents, de manera que la proteïna de fusió no té la capacitat d'unir-se a les membranes cel·lulars, i passa a localitzar-se en el territori de NPM1, i activada de forma permanent. Aquesta és la característica que causa la transformació dels limfòcits en cèl·lules malignes. La proteïna resultant es considera així una oncoproteïna.

Figura 6: Respresentació de la proteïna de fusió amb els dominis d'oligomerització (NPM1) i tirosina-kinasa (ALK).
Per l'estudi de l'homologia, al tenir en compte que ni l'Ensembl ni el Genome Browser reconeixen la proteïna quimèrica, el que primer s'ha fet és un BLASTN de la seqüència de nucleòtids mitjançant la base de dades de l'NCBI. A partir d'aquí s'ha obtingut una llista de proteïnes (o parts de proteïnes amb homologia significativa) d'Homo sapiens i de les diferents espècies que la base de dades ha trobat. Per representar millor els resultats d'aquesta cerca, es mostren les dades en forma d'arbre filogènic:

Figura 7: Arbre filogènic obtingut per la proteïna quimèrica a partir de l'NCBI
Tal com es pot observar, la major part d'homologies trobades corresponen a proteïnes similars del mateix Homo sapiens, majoritàriament relacionades pel domini ALK. De fet, de totes les seqüències que surten representades en els resultats, només es pot relacionar també el domini NPM1 en alguna variant de la proteïna quimèrica que s'estudia i en el mateix Homo sapiens; en cap altre cas s'hi ha trobat semblança. També cal destacar que la majoria d'homologies en altres espècies corresponen a seqüències PREDICTED, i per tant representa que el resultat de la cerca no és del tot significatiu o real.
En principi es podria especular que la causa d'això és que potser el domini d'NPM1 fusionat es troba molt poc conservat entre les espècies i que per tant al moment en què el BLAST fa l'alineament no troba semblances prou significatives enlloc. Si fos així, voldria dir que el domini d'oligomerització d'NPM1 no representa un paper massa important en la proteïna, de manera que no haurà patit pressió selectiva per mantenir-se constant al llarg de l'evolució. Per poder aprofundir una mica més amb aquesta hipòtesi, s'han intentat buscar homologies a partir de tota la seqüència d'NPM1:
Per una banda, s'ha fet un altre BLASTN amb la seqüència completa de nucleòtids d'NPM1. S'ha agafat el transcrit 1 com a representant de les tres isoformes, al ser el primer que es va descriure i per la semblança força general amb els altres dos transcrits. Els resultats obtinguts ratifiquen en principi l'explicació donada anteriorment, ja que pràcticament totes les proteïnes trobades corresponen a variants d'Homo sapiens, i a més ens permeten afegir que no només el domini d'oligomerització no es troba conservat, sinó que en general no ho està cap domini de la nucleofosmina. De representants d'altres espècies només se'n troben en Macaca mulatta, Pan troglodytes, Pongo pygmaeus i Canis familiaris, i tan sols es tracta de seqüències PREDICTED.
Per altra banda, també s'ha buscat a partir de l'Ensembl, però no s'han obtingut resultats ni de gens ortòlegs ni de paràlegs, la qual cosa coincideix en part amb el BLAST anterior, ja que en cap dels dos casos s'obté una llista significativa.
En tercer lloc hem utilitzat l'HomoloGen, també de l'NCBI, els resultats del qual surten reflectits en la següent imatge:
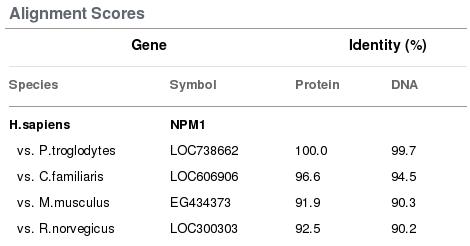
Figura 8: Resultats obtinguts amb l'HomoloGen per la proteïna NPM1
En principi podria semblar que realment existís una alta homologia entre les seqüències de les espècies de la taula, però s'ha de tenir en compte que cap dels gens de les espècies que s'estan comparant amb l'NPM1 humana surt descrit com a gen de la nucleofosmina, excepte en el R.norvegicus, tot i que només n'indica similaritat estructural. En el cas de Pan troglodytes i Canis familiaris, corresponen a proteïnes similars a la humana però no en surt explicada la seva funció. En Mus musculus s'està considerant un gen PREDICTED.
Finalment, a partir de l'HomoloGen, enllaçant amb els resultats d'UNIGEN, s'ha vist que tant R.norvegicus com M.musculus contenen realment un gen ortòleg per la nucleofosmina, estructuralment molt semblant. La descripció funcional que se'n fa coincideix en gran part amb la funció que té la NPM1 humana.

Figura 9: Resultats obtinguts amb l'UNIGEN per la proteïna NPM1
Per acabar de completar els resultats, també s'han analitzat els ortòlegs d'ALK, sobretot després de que en el primer BLAST fet a la seqüència del gen de fusió es mostressin vàries semblances d'aquesta proteïna en diverses espècies.
Com en el cas anterior, s'ha fet inicialment un BLASTN amb la seqüència sencera. Aquesta vegada també s'ha agafat només un dels transcrits com a representant de l'altre que s'ha descrit, sobretot perquè les diferències que hi ha entre ells no afecten al domini catalític, que és el ens interessa comprovar si està conservat tant per la implicació que té en la proteïna de fusió com pel seu gran tamany. A més, aquest és el que provocava la llista d'homologies trobades en el primer BLAST.
Com es pot apreciar en la l'arbre filogènic, es ratifica que la majoria d'homologies es basen en la semblança del domini catalític, que és el que dóna activitat tirosina kinasa al receptor ALK. Les diverses homologies que apareixen corresponents a Homo sapiens de fet, són altres proteïnes tirosina-kinases, encara que diferents d'ALK, demostrant així que el domini que els dóna activitat està conservat en totes les proteïnes de la mateixa família. Pel que fa a les altres espècies, també s'observen homologies tant en gens que codifiquen per ALK (fet que indicaria que són ortòlegs) com en gens d'altres tirosina kinases (fet que tindria la mateixa explicació que s'ha donat per Homo sapiens). La grandària del domini també explicaria que les diverses proteïnes s'agrupin dins el mateix resultat d'homologia.
Per aprofundir amb els resultats, s'ha buscat també a l'Ensembl, i aquesta vegada sí que s'ha obtingut una llista de possibles ortòlegs, que es mostra en la següent taula, on les espècies s'han ordenat per ordre descendent respecte el percentatge d'identitat de la seqüència peptídica. També s'ha inclòs la descripció que es dóna del gen en cada cas, per comprovar si es tracta d'ALK o d'altres tirosines kinases:
| Espècie | % Homologia | Descripció del gen |
| Maccaca mulata | %id: 97 | Receptor tirosin-kinasa |
| Rattus norvegicus | %id: 88 | Kinasa limfoma anaplàstic (fragment) |
| Mus musculus | %id: 87 | Kinasa limfoma anaplàstic (fragment) |
| Pan troglodytes | %id: 87 | No descrit |
| Felis catus | %id: 83 | No descrit |
| Bos taurus | %id: 77 | No descrit |
| Tupaia belangeri | %id: 74 | No descrit |
| Canis familiaris | %id: 70 | No descrit |
| Dasypus novemcinctus | %id: 68 | No descrit |
| Loxodonta africana | %id: 66 | No descrit |
| Echinops telfairi | %id: 65 | No descrit |
| Oryctolagus cuniculus | %id: 63 | No descrit |
| Monodelphis domestica | %id: 62 | Receptor tirosin-kinasa |
| Cavia porcellus | %id: 58 | No descrit |
| Gallus gallus | %id: 57 | Receptor tirosin-kinasa |
| Ornithorhynchus anatinus | %id: 56 | No descrit |
| Erinaceus europaeus | %id: 55 | No descrit |
| Gasterosteus aculeatus | %id: 36 | No descrit |
| Oryzias latipes | %id: 34 | No descrit |
| Xenopus tropicalis | %id: 34 | Receptor tirosin-kinasa |
| Tetraodon nigroviridis | %id: 29 | No descrit |
| Danio rerio | %id: 28 | No descrit |
| Takifugu rubripes | %id: 28 | Receptor tirosin-kinasa |
| Anopheles gambiae | %id: 21 | No descrit |
| Drosophila melanogaster | %id: 21 | Alk CG8250-PA |
| Aedes aegypti | %id: 20 | Receptor tirosin-kinasa leucócit |
| Caenorhabditis elegans | %id: 17 |
Taula 1 Estudi dels gens ortòlegs del gen ALK
Es pot observar, com en el cas de l'NPM1, una alta similaritat entre humans, primats i rosegadors com M.musculus i R.norvegicus. Tot i així, tampoc es pot passar per alt la gran quantitat i diversitat d'espècies que es descriuen com ortòlegs del gen ALK. De totes maneres, al veure que la majoria de vegades, a l'estudiar l'homologia, es té en compte tan sols l'activitat kinasa de la proteïna per la gran semblança dels dominis catalítics de totes elles (només 8 casos dels 27 surten descrits com a ALK), s'ha intentat comprovar d'alguna manera que això és més que una simple observació.
A partir del programa informàtic MEGA, s'ha realitzat un alineament múltiple mitjançant el ClustalW amb totes les seqüències peptídiques d'ortòlegs obtingudes amb l'Ensembl, i s'han marcat els aminoàcids que es trobaven conservats en la totalitat (o quasi) de totes elles.
Analitzant els resultats hem comprovat que efectivament existeix una gran conservació interespecífica del domini catalític de les tirosina kinases, i que per aquest motiu existeixen tants homòlegs del gen d'ALK.
En aquest cas a més, també s'han comprovat els resultats obtinguts des de l'Ensembl amb els que proporciona el BIOMART, fet que ens ha servit per veure la total coincidència entre les dues bases de dades.
Per tal de seguir els mateixos passos que en l'NPM1, a continuació s'ha estudiat la informació obtinguda amb l'HomoloGen, que es resumeix en la següent taula:
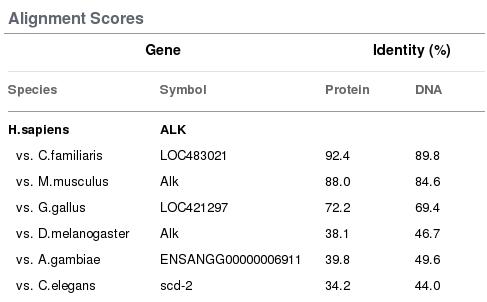
Figura 10: Resultats obtinguts amb l'HomoloGen per la proteïna ALK
En aquest cas, la llista d'espècies amb seqüències homologues és molt menor (tot i que els mostrats aquí surten representats tots a la taula de l'Ensembl). Cal dir a més, que el percentatge d'homologia peptídica obtinguda difereix entre les dues bases de dades, en excepció a M.musculus, en què pràcticament surt la mateixa. El que sí coincideix en els dos casos és la seqüència que s'està comparant, ja que la descripció que fa l'HomoloGen de cadascun dels gens que representen és idèntica a la de l'Ensembl.
Per últim, amb l'UNIGEN, s'obté la següent llista, amb la corresponent homologia de seqüències d'altres espècies i d'una altra tirosina kinasa humana respecte l'ALK:

Figura 11: Resultats obtinguts amb l'UNIGEN per la proteïna ALK
Tornant amb el que s'ha anat explicant fins ara en quant a l'ALK, veiem clarament que les homologies obtingudes en les bases de dades són degudes sobretot a la semblança entre dominis catalítics de les tirosina kinases. Aquesta llista ens mostra que tan sols en un cas la seqüència que s'està comparant correspon a un gen que codifica per ALK (en el M.musculus). Els altres casos, corresponen a altres proteïnes kinases.
El perfil d'expressió del gen NPM1 s'ha obtingut del Genome Broswer, en dos xips de microarray, U133A i GNF1H del GNF Expression Atlas 2. A continuació es mostra una taula amb els nivells d'expressió obsevats en els diferents teixits o tipus de cèl·lula:
| Teixits o tipus de cèl·lula | Nivell d'expressió | Teixits o tipus de cèl·lula | Nivell d'expressió | Teixits o tipus de cèl·lula | Nivell d'expressió |
| Encèfal | Node limfàtic | Leucèmia promielocítica | |||
| Lòbul temporal | Amígdala | Limfoma Burkitts Daudi | |||
| Lòbul occipital | Cèl·lula endotelial BM-CD105+ | Leucèmia mielògena crònica | |||
| Cerebel | BM-CD34+ | Mieloide BM-CD33+ | |||
| Peduncles cerebelosos | Sang | Pell | |||
| Nucli subtalàmic | Cèl·lula dendrítiques PB-BDCA4+ | Tiroide | |||
| Glòbus pàlid | Monòcits | Cor | |||
| Protuberància | Cèl·lules NK PB-CD56+ | Miòcits | |||
| Bulb raquidi | Cèl·lules T PB-C4+ | Múscul esquelètic | |||
| Gangli ciliar | Cèl·lules T PB-C8+ | Múscul llis | |||
| Gangli trigeminal | Cèl·lules B PB-CD19+ | llengua | |||
| Gangli cervical superior | Leucèmia limfoblàstica | Pulmó fetal | |||
| Gangli de l'arrel dorsal | Limfoblastes B 721 | Cèl·lules epitelials broquials | |||
| Glàndula pituitària | Limfoma Burkitts Raji | Fetge |
Taula 2: Perfil d'expressió del gen NPM1
Podem veure que el gen NPM1 s'expressa en tot el conjunt del sistema circulatori, incluïnt cèl·lules limfàtiques i cèl·lules sanguínies.
Els alts nivells d'expressió d'aquest gen es reflexen en diferents parts del sistema limfàtic com són el node limfàtic, l'amígdala i els tres tipus de limfòcits (T,B i NK).
Tanmateix, aquests alts nivells d'expressió també els podem observar en cèl·lules sanguínies madures. No obstant, ni en la mèdul·la òssea encarregada de la generació d'aquestes cèl·lues, ni en els primers estadis de formació dels eritròctis trobem l'expressió de NPM1.
A més a més, obtenim una informació força significativa ja que s'aprecia expressió de la proteïna en diversos tipus de càncers sanguínis (leucèmies i limfomes). Tot i que el tipus de limfoma estudiat en aquest treball no hi surt detallat sí que trobem representants de limfomes tipus no-Hodgkin (dos subtipus de limfoma Burkitts).
Per altra banda, l'expressió del gen ALK en diferents teixits o tipus cel·lulars s'ha obtingut també del Genome Browser, en aquest cas en un xip de microarray, Uffy U95 del GNF Expression Altas 1.
| Teixits o tipus de cèl·lula | Nivell d'expressió | Teixits o tipus de cèl·lula | Nivell d'expressió | Teixits o tipus de cèl·lula | Nivell d'expressió |
| Encèfal | Médul·la espinal | WSU | |||
| Cerebel | Melsa | A2058 | |||
| Nucli caudat | THY- | Fetge fetal | |||
| Amìgdala | Miel`gen K562 | Páncrees | |||
| Tálam | Limfoma Burkitts Daudi | Testicle | |||
| Glàndula pituitària | Limfoma Burkitts Raji | Ovari |
Taula 3 Perfil d'expressió del gen ALK.
El resultat de l'expressió d'aquest gen és la proteïna ALK, la qual exerceix els seus efectes en neurones específiques del sistema nerviòs, fet que coincideix amb l'expressió del seu gen. Com podem observar, aquesta té lloc al sistema nerviòs central i perifèric. Respecte el SNC l'expressió es concentra en parts específiques com són el cerebel, el nucli caudat, l'amígdala, el tàlam i la glàndula pituïtària. Tot i així, en el sistema nerviòs perifèric l'expressió és molt més general ja que n'observem en tot el recorregut de la mèdul·la espinal. El gen ALK no s'expressa en cèl·lules limfàtiques sanes però en canvi sí s'obté un alt nivell d'expressió en limfomes, concretament de tipus no-Hodgkin.
Per altra banda, s'ha fet una cerca de l'expressió del gen al VisiGene d'on s'han pogut extreure les següents imatges, les quals reflexen el que s'ha vist anteriorment, una alta expressió d'ALK a diferents parts específiques del sistema nerviós central i a gran part del perifèric.
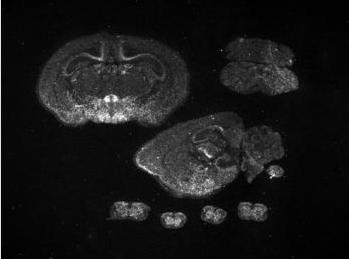 |
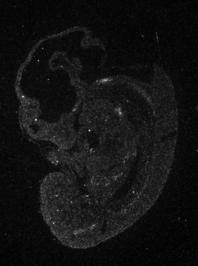 |
Figura 12: Expressió del gen ALK en un embrió de ratolí.
La regió promotora del gen ALK-NPM1 estudiat correspon a la regió promotora del gen NPM1, ja que com s'ha comentat anteriorment la regió 5' del gen de la proteïna de fusió pertany a la regió N-terminal de NPM1. Tot i així com que l'estudi genònic s'ha realitzat per cada un dels gens, també s'han caracteritzat les regions promotores d'ambdós. Aquestes regions formades per 1kb upstream del lloc d'inici de la transcripció (TSS) i 100 bp downstream del TSS s'han extret del Genome Browser, base de dades que en un inici es va considerar més significativa. Les respectives regions promotores de NPM1 i ALK es mostren a continuació, indicant en vermell les 1000 pb upsteam i en gris les 100 bp downstream del TSS.
| Regió promotora NPM1 | Regió promotora ALK |
>tgtaagaaataatgccttggggtgggggtgtcttcctttctgaggct atcatttgtattcctcacttcttaaatttgtttgatatgttgaggctt aaaaaaaaaagattacgggtcttcagaacgccccaatgcccgcggggt gctggggtgcctcggagtttgtcaaggtgtagcttaaaacgttaattc ccatgctcgctcccaaatattcgaattaaggcttctgtagttcttaca agtcacccgctttctttcaggaggaatgttaaagattaagagcggcaa gaagtcagagtcggcccaccctccgagctctcttagggcgatgtcctt gctaatttggagactgattcagtccccttttggcccccaagttacgta aagataaggactttggagatgttttctcaggaaggacagagctgaaaa acaaagttccgtaagtctggccggtaactacatctttccttcctaaca aagaataagccgcaattcactctctctggcacctgaactttggggtaa cgattaactgcgatccgaagccttcaggcaagtaaaaaattcctgaag tgattacgcctgtttggaggcttgcagggcactaggggatggggaaag gtgaatcgaggtgctctctggctcattcgcagccggctaacccgccat atcttacacagctaagggctgccgacgccattttgcagggtgggctgc gcagactcttggcgggaggccggcgcgcttgagcgggagaggacgtgg aagggttggaggtggggagtgcgcgttacgactggaaagcacgcgtgc gcacaggcgacagcagcggaggggtggggccagtgtacgagtgcgcgt gctcggtgggagcccgcggagtacctggaaggaggtgggggcgaggta gaaaggagtggggttgaaaagcgcttgcgcaggacggctacggtacgg gggtgggagggcttcggagcacgcgcgcggaggcgggacttGGGAAGC GCTCGCGAGATCTTCAGGGTCTATATATAAGCGCGGGGAGCCTGCGTC CTTTCCCTGGTGTGATTCCGTCCTGCGCGGTTGTTCTCTGGAGCA |
>tgtctgaggatctagtgaaccacttgttataaaaacagctattatga gttctgtgttggcagctcaggagagacgaaaggaaagggagaggagag gtacagccattacaggtgagtaaaaaaggcctaaggttctgaaccctc attcccaagattgtgggcaaacaattaaatgctctgcaactcagtttc tgcatctgtaaatctggaattaaaatgtttgccttacagagactaggg gaggttacacatgttcagacaccattctgagaaaacagagcgactgac aggggtctgaaaggtatttgttgtagctgcagaacaactctgccagac caagaccatccatccctctctgcccccctattcccaaattctcctgtg tggacggcaggactcctaagctcccaggaatgcattcaaataatagat gggtcagaaaatattctgtctcagggccttaatacaagctgttctcag atttgccagtgtcgcgctgccaccctctccccacttcctcctcccttc ccactcccccctcccttcccctctcctccagttttattctggaacctg tttttccgaagtcggacccgtttaatctcttaaatgtataattaggga gagtgcttgattgcaaaggcctcttccagttctcacatttgctccctt tcacactgcagagaaatagggcagggaatctagaggaggggaagaaca agagactggagagggaacagagggagggtggggcgggctcactccttt tctcaatgaatgccgaggcctctgcagatttgcataggagccgatcga gccacgccattggttggagggtgcgggtggggcggggcgaggccggac tgcgtgggtcgggcagcagcgcggagttggcttgtgagccccgccccc tccgggccccgccccctccctgcgcgcgctcgcgcggctcagccagct gcaagtggcgggcgcccaggcagatgcgatccagcggctctGGGGGCG GCAGCGGTGGTAGCAGCTGGTACCTCCCGCCGCCTCTGTTCGGAGGGT CGCGGGGCACCGAGGTGCTTTCCGGCCGCCCTCTGGTCGGCCACC |
A partir de 13 matrius de factors de transcripció humans diferents que podrien unir-se a aquestes regions promotores s'ha realitzat un programa Perl per tal de determinar la puntuació que s'obté per cada factor de transcripció al llarg de la regió promotora així com la posició que la proporciona. A més a més també s'estima el pvalue, freqüència amb la qual s'observaria a l'atzar la puntuació màxima anterior i així saber el grau de significància dels resultats obtinguts.
Paral·lelament les regions promotores també es van caracteritzar utilitzant el servidor web del programa PROMO, on es van seleccionar possibles factors de transcripció que podrien estar regulant l'expressió de cadascún dels gens. Aquesta selecció s'ha realitzat amb un valor de dissimilaritat del 15% i els resultats obtinguts es poden trobar en els següents enllaços, NPM1 i ALK. D'aquest gran nombre de FT s'han seleccionat els que presentaven un RE query > 0,09 i una dissimiliritat > 2 obtinguent així els millors candidats que poden estar regulant l'expressió de cadascún dels gens, NPM1 i ALK.
Seguidament s'han comparat els resultats obtinguts per cadascún dels gens en els diferents mètodes emprats, Perl i PROMO. Cal comentar que la coincidència entre ambdós programes no és molt alta, lògicament perquè les matrius utilitzades no són les mateixes i a més a més el nombre de factors de transcripció que es disposava pel programa Perl era limitat.
Gen NPM1
| Factor name | Max Score | Start position | End position | String | p-value |
| AP1 [T00029] | 2,52 | 287 | 293 | GAAGTCA | 0,29 |
| AR [T0040] | 2,12 | 513 | 519 | GAACTTT | 0,77 |
| c-Myc [T00140] | -995,91 | 508 | 513 | CACCTG | 0,82 |
| NF-AT1 [T00550] | 2,64 | 617 | 623 | GGAAAGG | 0,55 |
| NF-kappa B [T00590] | -995,85 | 562 | 570 | AAAATTCCT | 0,68 |
| SRF [T00764] | -994,8 | 336 | 344 | CTAATTTGG | 0,19 |
| YY1 [T00915] | 1,92 | 613 | 618 | ATGGGG | 0,95 |
| RXR-alpha [T001345] | 2,92 | 512 | 517 | TGAACT | 0,34 |
| HIF-1 [T01609] | -996,23 | 551 | 559 | GCTGCAAGT | 0,95 |
| AhR [T01795] | 3,09 | 809 | 815 | GCGTGGG | 0,19 |
| PU1 [T02068] | 3,1 | 410 | 416 | CAGGAAT | 0,26 |
| HNF-4 [T02758] | -995,47 | 512 | 219 | TGAACTTT | 0,34 |
| NRSF [T06124] | -996,17 | 823 | 831 | GACAGCAGC | 0,47 |
Taula 4: Resultats obtinguts en Perl.
L'anterior taula mostra els resultats obtinguts pel gen NPM1 mitjançant el programa Perl realitzat. Els factors de transcripció amb una alta probabilitat d'unir-se a la seqüència promotora del gen NPM1 són els que presenten una puntuació més alta i un valor de pvalue més petit. Tenint en compte això es poden predir els diferents factors de transcripció que podrien estar regulant l'expressió del gen a estudi: AP1 [T00029] amb una puntuació de 2,52 i un valor de pvalue de 0,29; RXR-alpha [T001345], amb un score de 2,92 i un pvalue de 0,34; AhR [T01795] presentant un socre de 3,09 i un pvalue de 0,19 i PU1[T0208] amb un 3,1 i 0,26 de puntuació i pvalue respectivament. Tots ells per tant, amb un baix valor de pvalue i un alt score.
| Factor name | Start position | End position | Dissimilarity | String | RE equally | RE query |
| PXR-1:RXR-alpha [T05671] | 512 | 519 | 1,64 | TGAACTTT | 0,03 | 0,02 |
| AR [T00040] | 416 | 424 | 4,24 | GGACAGAGC | 0,04 | 0,04 |
| RXR-alpha [T01345] | 921 | 927 | 1,88 | GGGTTGA | 0,07 | 0,09 |
| NF-AT1 [T00550] | 617 | 625 | 7,74 | GGAAAGGTG | 0,11 | 0,1 |
| AP-1 [T00029] | 285 | 293 | 13,42 | AAGAAGTCA | 0,13 | 0,12 |
| YY1 [T00915] | 613 | 616 | 0 | ATGG | 4,3 | 4,37 |
Taula 5: FT selecionats des del programa PROMO.
Aquesta taula mostra únicament els factors de transcripció obtinguts del PROMO que coincideixen amb els del programa Perl. Així es poden comparar els resultats obtinguts mitjançant els dos mètodes emprats.
Es pot veure també com els factors de transcripció NF-AT1 [T00550] i YY1[T00915] coincideixen tant en posició d'inici com en seqüència. Tot i així, el valor de RE query de YY1 [T00915] és molt alt i per tant, aquesta coincidència és poc significativa. Per contra, el RE query de NF-AT1 [T00550] és baix i el valor de dissimiliritat és acceptable, podent ser per tant, un possible candidat a regular l'expressió del gen en qüestió com també l'expressió de la nostra proteïna de fusió. Pel factor de transcripció AP-1 [T00029] s'obté la mateixa posició d'inici en ambdós mètodes, però no es pot dir el mateix amb la seqüència. Cal comentar també que el factor de transcripció PXR-1:RXR-alpha [T05671] trobat mitjançant el PROMO coincideix en seqüència i posició amb el RXR-alpha [T001345] present al Perl, i tots dos amb resultats força significatius. El factor de transcripció restant, AR[T00040] no coincideix ni en posició d'inici ni en seqüència tot i que el seu RE query és significatiu.
Per últim dir que els valors de p-value i RE query obtinguts mitjançant els programes PERL i PROMO respectivament no coincideixen.
| Factor name | Max Score | Start position | End position | String | p-value |
| AP1 [T00029] | -995,57 | 111 | 117 | TGAGTAA | 0,68 |
| AR [T0040] | 3,46 | 733 | 740 | GAACAGA | 0,04 |
| c-Myc [T00140] | -996,02 | 20 | 25 | CACTTG | 0,85 |
| NF-AT1 [T00550] | 2,87 | 78 | 84 | GGAAAGG | 0,39 |
| NF-kappa B [T00590] | -996,52 | 477 | 485 | AGATTTGCC | 0,97 |
| SRF [T00764] | -99,15 | 38 | 46 | CTATTATGA | 0,28 |
| YY1 [T00915] | 1,78 | 429 | 35 | ATTGGGT | 0,97 |
| RXR-alpha [T001345] | 3,1 | 15 | 20 | TGAACC | 0,27 |
| HIF-1 [T01609] | -995,62 | 956 | 964 | GCTGCAAGT | 0,47 |
| AhR [T01795] | 2,67 | 864 | 870 | GCGTGGG | 0,69 |
| PU1 [T02068] | 5,58 | 407 | 413 | CAGGAAT | 0,38 |
| HNF-4 [T02758] | -995,54 | 205 | 212 | TGGAATTA | 0,58 |
| NRSF [T06124] | -996,12 | 874 | 882 | GGCAGCAGC | 0,54 |
Taula 6: Resultats obtinguts en Perl
L'anterior taula mostra els resultats obtinguts pel gen NPM1 mitjançant el programa Perl realitzat. Els factors de transcripció amb una alta probabilitat d'unir-se a la seqüència promotora del gen NPM1 són els que presenten una puntuació més alta i un valor de pvalue més petit. Tinguent en compte això es poden predir els diferents factors de transcripció que podrien estar regulant l'expressió del gen a estudi: AP1 [T00029] amb una puntuació de 2,52 i un valor de pvalue de 0,29; RXR-alpha [T001345], amb un score de 2,92 i un pvalue de 0,34; AhR [T01795] presentant un socre de 3,09 i un pvalue de 0,19 i PU1[T0208] amb un 3,1 i 0,26 de puntuació i pvalue respectivament. Tots ells per tant, amb un baix valor de pvalue i un alt score.
| Factor name | Start position | End position | Dissimiliraty | String | RE equally | RE query |
| PU,1 [T02068] | 509 | 521 | 3,72 | CCACTTCCTCCTC | 0 | 0 |
| NF-AT1 [T00550] | 78 | 86 | 9,18 | GGAAAGGGA | 0,13 | 0,12 |
| RXR-alpha [T01345] | 134 | 140 | 0 | TGAACCC | 0,13 | 0,13 |
| YY1 [T00915] | 429 | 432 | 0 | ATGG | 4,3 | 4,2 |
Taula 7: FT selecionats des del programa PROMO.
Aquesta taula mostra únicament els factors de transcripció obtinguts del PROMO que coincideixen amb els del programa Perl. Així es poden comparar els resultats obtinguts mitjançant els dos mètodes emprats. Els factors de transcripció NF-AT1 [T00550] i YY1 [T00915] coincideixen en posició i seqüència. Però el valor de RE query de YY1 [T00915] és molt alt i per tant, poc significatiu. En canvi NF-AT1 [T00550] presenta uns resultats a considerar en tots dos mètodes. Observant el factor de transcripció RXR-alpha [T001345] veiem que coincideix en posició d'inici però no en seqüència. PU,1 [T02068] tot i tenir un RE query molt significatiu de valor 0 no coincideix ni en la posició d'inici ni amb la seqüència obtingudes amb el programa Perl. Per últim esmentar que els valors de p-value i RE query obtinguts mitjançant els programes PERL i PROMO respectivament no coincideixen.
Per determinar la funció de l'oncogen format també s'ha treballat amb els dos gens originals per separat. A partir de dades obtingudes de Gen Ontology, de l'OMIM (NCBI) i d'articles del PubMed, s'ha descrit la funció de cada gen en wild type i finalment s'han relacionat per donar una explicació al mecanisme fisiològic de la proteïna de fusió.
ALK ha estat descrita bàsicament com a proteïna que en condicions normals està implicada en el desenvolupament del sistema nerviós i de l'encèfal, que en el Gen Ontology es relaciona amb els termes GO:0007399 i GO:0007420 respectivament. Com ja s'ha comentat en apartats anteriors, es tracta d'un receptor transmembrana amb activitat tirosina kinasa, i per tant la seva funció, no descrita en detall encara, però suposadament comuna amb tots els altres receptors similars, seria la de senyalització cel·lular a través de fosforilacions (ATP + tirosina = ADP + tirosina fosfat).
A nivell de publicacions, ALK es descriu sobretot per la seva implicació en diversos tipus de càncers, degut a la fusió amb altres gens formant oncoproteïnes.
L'NPM1 ha estat associada a múltiples funcions moleculars i processos biològics associats a varis termes GO en la base de dades de Gen Ontology. Després d'intentar ampliar la informació proporcionada amb la cerca d'articles relacionats amb la proteïna, s'han seleccionat aquelles funcions més destacades en els diversos estudis publicats o que més interès tenen per relacionar amb la proteïna de fusió i la patologia que causa.
Chan et al. (1989), els qui van descriure la proteïna, van especular que probablement estava implicada en l'assemblatge de proteïnes ribosomals en els ribosomes, fet que coincideix amb el terme GO:0042255. Estudis amb microscopi electrònic van corroborar aquesta funció al mostrar que la nucleofosmina està majoritàriament concentrada en la regió granular del nucleol, on té lloc l'assemblatge ribosomal. Aquesta localització es pot veure a la figura següent, extreta del VisiGene.
|
| Figura13: Imatge de la localització nucleolar de NPM1 visualitzada per microscopia electrònica. |
Dotze anys més tard, Okuda et al. (2000) van demostrar l'important paper de l'NPM1 en el cicle cel·lular. Van identificar aquesta proteïna com a substrat del complex CDK2/Ciclina E en la duplicació del centrosoma. Concretament, NPM1 s'uneix al centrosoma en la fase G1 impedint que es dupliqui; llavors, coincidint amb l'entrada a la fase S del cicle cel·lular, s'hi dissocia per una fosforilació mitjançada pel complex CDK2-Ciclina E, kinasa associada als factors d'iniciació de la replicació cel·lular. NPM1 torna a fosforilar-se durant la fase M gràcies a un altre complex CDK/ciclina, concretament el CDK1/ciclina B.Diversos estudis amb NPM1s no-fosforilades (per mutació o marcatge d'anticòs especíc que inhibia la fosforilació) van servir per veure que d'aquesta manera se suprimia la duplicació centrosòmica in vivo. Així, la conclusió és que NPM1 actua com a diana de la CDK2/ciclina E en l'inici de la duplicació del centrosoma, permentent que aquesta es pugui realitzar. Aquesta funció està relacionada amb el terme GO:0007098.
NPM1 wild type a més s'ha vist que té un important paper en la regulació de p53 (inductor d'apoptosi), unint-se a ell i a les seves proteïnes associades (ARF i MDM2), i així incrementant-ne la seva estabilitat. L'objectiu de l'NPM1 és activar la transcripció de p53 després de danys irreversibles a l'ADN causats per estrés (per exemple, per exposició a UV), o induïr senescència prematura en cèl·lules embrionàries amb el genoma malmès (GO:0006950). Aquí és on s'ha vist la importància de l'isoforma 3, ja que s'ha comprovat que la seva presència en el citoplasma (més que en el nucli, en aquest cas) estava relacionada amb la resistència cel·lular enfront les radiacions.
El mecanisme fisiològic de la proteïna quimèrica encara no està del tot estudiat. Cal dir però que l'interès que existeix en intentar comprendre'l és actualment màxim, i una mostra d'això és la més que significativa quantitat d'articles publicats en els últims mesos referents al tema.
Fins fa poc se sabia que la proteïna quimèrica autofosforilada aconseguia la capacitat de transformar les cèl·lules per l'activació persistent de vàries vies de senyalització cel·lular. Es va veure que algunes d'aquestes vies estaven relacionades amb proto-oncogens, però no existia una descripció detallada d'altres que poguessin estar implicades.
Per tant doncs, bàsicament l'atenció es centra en la recerca del coneixement d'aquestes vies activades a conseqüència de la proteïna fusionada. Un article publicat a la revista Nature al febrer d'aquest mateix any demostra la participació de la via de les MAPkinases ERK i MEK, que al ser activades constitutivament per la proteïna NPM-ALK es creu que juguen un paper molt important en la transformació maligna de les cèl·lules, fent que s'estimuli la proliferació i la supervivència de les mateixes. De fet, se sap que en aproximadament un 30% dels càncers humans aquesta via està activada de forma constitutiva.
La importància d'aquest coneixement, segons el mateix article, rau en la possibilitat de pensar que la intervenció directa (inhibint o bloquejant) en els funcionaments de les vies anormalment activades per NPM-ALK podria servir com a solució contra la transformació cel·lular, i d'aquesta manera impedir el creixement tumoral. Aquests resultats a més servirien per complementar-ne uns altres publicats uns mesos abans, que asseguraven que s'havia identificat un potent i selectiu inhibidor de la proteïna ALK, l'NVP-TAE684, que actua inhibint la fosforilació de la proteïna de fusió i per tant els seus successius en les vies de senyalització; en conseqüència la cèl·lula entra en apoptosi i es bloqueja aixií el creixement de les línies cel·lulars canceroses.