
Estudi genòmic computacional del gen de fusió MOZ-ASXH2
Índex
- Resum
- Introducció
- Mètodes
- Materials
- Resultats i discussió
- Estructura genòmica
- MOZ
- ASXH2
- Informació de la proteïna quimèrica
- Conservació en altres espècies
- Caracterització de l'expressió
- MOZ
- ASXH2
- Caracterització de les regions promotores
- Dominis i funció de la proteïna
- Referències
En aquest treball presentem la caracterització de l'estructura exònica, homologia, funció i expressió del gen de fusió humà MOZ-ASXH2, provinent d'una translocació. La proteïna quimèrica (o de fusió) codificada per aquest gen és una acetilhistona. Aquesta translocació està implicada en la leucèmia mieloide, ja que juga un paper important en l'hematopoiesi. El paper habitual del gen MOZ (que codifica per una histona acetil transferasa coneguda com ZNF220 o MYST3 i és membre de la família MYST - acetiltransferasses- es veu alterat a causa de la translocació i la traducció conjunta amb ASXH2, que pertany al grup dels gens Polycomb, requerits per mantenir estable la repressió dels gens homeòtics i altres loci i que normalment s'uneix al DNA per tal de regular el metabolisme de les nucleobases, nucleòsids, nucleòtids i àcids nucleics. Mentre que MOZ consta d'un sol transcript que dóna lloc a una sola isoforma, el gen ASXH2 té 2 transcripts diferents, amb un total de 2 isoformes.

La leucèmia mieloide aguda és una neoplàsia clonal del teixit hematopoiètic caracteritzada per la proliferació de cèl·lules blàstiques anormals d'estirp mieloide a la medul·la òssia i menor producció de cèl·lules hemàtiques normals, condicionant anèmia i trombopènia. Es produeix per danys genètics adquirits al DNA de les cèl·lules en desenvolupament de la medul·la òssia.
Es tracta del tipus més comú de leucèmia en adults i és també coneguda com leucèmia mielògena aguda, leucèmia mieloblàstica aguda, leucèmia granulocítica aguda i leucèmia no limfocítica aguda.
En aquesta malaltia, les cèl·lules mare de la medul·la generen un tipus de glòbuls blancs immadurs i anormals (mieloblasts). També pot succeïr que un excessiu nombre de cèl·lules mare es desenvolupin i converteixin en glòbuls vermells o plaquetes anormals. Aquests grups de cèl·lules no poden dur a terme les seves funcions habituals i s'acumulen a la medul·la i la sang, de manera que disminueix la capacitat per allotjar cèl·lules sanes i, d'aquesta manera, es possibilita el desenvolupament d'infeccions, anèmies i hemorràgies. Les cèl·lules leucèmiques es poden propagar fora de la sang fins altres parts de l'organisme com el sistema nerviós central, la pell o la geniva.
Hi ha diferents graus de Leucèma Mieloblàstica Aguda en funció del grau de maduresa de les cèl·lules canceroses i com es diferencïen de les normals.
Els factors de risc que augmenten les possibilitats de patir LMA són:
- Sexe masculí
- Tabaquisme
- Tractaments de químioteràpia o radioteràpia
- Exposició a radiació ionitzant
- ...
Els signes de la malaltia inclouen febre, cansament i formació fàcil d'hematomes o hemorràgies entre altres. Per tal de realitzar el diagnòstic es realitzen diferents proves: examen físic, consideració dels antecedents, recompte sanguini complet, estudis de la química de la sang, biòpsia de la medul·la òssia...
La incidència és d'1.5 casos per 100.000 habitants/any. La seva freqüència augmenta amb l'edat. Comprèn el 80% de les leucèmies agudes en adults i 15-20% de les d'infants. És la leucèmia més freqüent en nounats.
Com a tractament es duu a terme una teràpia de suport (mantenir l'hemoglobina en les concentracions necessàries, administració de plasma, ...) juntament amb un tractament específic (quimioteràpia citotòxica, transplant de medul·la òssia, ..).
Fent una cerca de la proteïna quimèrica en la base de dades del Pubmed, hem obtingut un sol article relacionat amb una malaltia provocada per la translocació t(2;8)(p23;p11). En data de publicació d'aquest article (Oct. 2003) no hi havia cap referència publicada d'aquesta translocació, de manera que es va arribar a la conclusió que aquest va ser el primer cas trobat o almenys reportat.
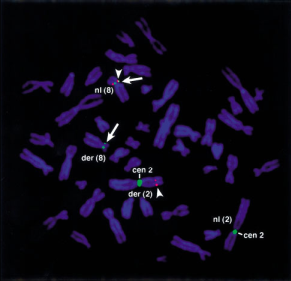 En aquest article, s'estudia el cas d'un infant amb sindrome mielodisplasic el qual va començar a desenvolupar els símptomes 17 mesos després de ser tractat per una leucèmia mieloblàstica aguda. Un cop es va fer el diagnòstic de la síndrome, és va fer un anàlisi del cariotip, i es va detectar una nova translocació t(2,8)(p23;p11.2). A més a més, es va fer un FISH el qual va suggerir un rearranjament del gen del zing finger de la leucèmia monocítica (MOZ) localitzat a la regió 8p11 del cromosoma 8, però no es va poder identificar el gen de la regió 2p23 del cromosoma 2.
En aquest article, s'estudia el cas d'un infant amb sindrome mielodisplasic el qual va començar a desenvolupar els símptomes 17 mesos després de ser tractat per una leucèmia mieloblàstica aguda. Un cop es va fer el diagnòstic de la síndrome, és va fer un anàlisi del cariotip, i es va detectar una nova translocació t(2,8)(p23;p11.2). A més a més, es va fer un FISH el qual va suggerir un rearranjament del gen del zing finger de la leucèmia monocítica (MOZ) localitzat a la regió 8p11 del cromosoma 8, però no es va poder identificar el gen de la regió 2p23 del cromosoma 2.
El FISH, és va realitzar fent una hibridació amb les regions 3' i 5' del gen MOZ marcades amb SpectrumOrange i SpectrumGreen respectivament. Per tal d'identificar el cromosoma 2, és va marcar una molècula amb FITC específica per la detecció del centròmer del cromosoma 2.
Com podem veure en la figura de la dreta extreta de l'article, veiem que la molècula marcada amb FITC ens indica on hi ha els 2 cromosomes 2. A més a més, veiem les senyals obtingudes pel marcatge de les regions 5' i 3' del gen MOZ. Observant detalladament les imatges, veurem que hi ha senyal del gen MOZ en els 2 cromosomes 8 tal i com es d'esperar, però el que crida l'atenció i demostra la translocació del gen MOZ, és el senyal que s'obté en un cromosoma 2, de la regió 3' del gen MOZ.

Per tal de saber de quina proteïna es tracta la seqüència que ens van cedir per fer aquest treball, vam utilitzar el programa tblastn de la pàgina del BLAST. El tblastn és un programa que compara una seqüència proteica amb una base de dades. El resultat serà una pàgina amb els diferents alineaments significatius de la proteïna amb diverses seqüències genòmiques. Mitjançant aquest resultat i valorant els scores obtinguts, podrem saber en quin gen (o quins, en aquest cas) està codificada la proteïna. A partir d'aquí obtindrem la seqüència genòmica.
Un cop sabem quins gens codifiquen per la proteïna quimèrica (MOZ-ASXH2), fem una cerca al Genome Browser de la pàgina del UCSC i a Ensembl. En el cas de Ensembl, no trobem informació sobre aquesta proteïna com a quimèrica, però a la pàgina del UCSC, obtenim com a resultat una pàgina que ens porta a les seqüències dels dos gens per separat. En aquesta pàgina, podem veure en quin cromosoma es troba cada gen, on comença i on acaba, i un link cap a la pàgina de l'estructura genòmica del gen MOZ i un altre a la pàgina pel gen ASXH2.
Per obtenir les seqüències genòmiques, les dels transcrits i proteiques de cada gen, vam fer una cerca a Ensembl dels 2 gens per separat, i, a partir d'aquï vam, exportar-les en format FASTA.
Per tal de determinar si aquests gens tenen ortòlegs en altres espècies s'han obtingut les dades d'Ensembl, que proporcionava un percentatge d'homologia que es corresponia amb l'obtingut amb Biomart. A més a més, mitjançant el programa Clustalw s'han pogut realitzar els respectius arbres filogenètics.

Abans de fer cap mena d'anàlisi s'han obtingut les següents seqüències:

Per tal de realitzar l'estudi de l'estructura genòmica s'ha dut a terme una recerca a Ensembl (www.ensembl.org) dels dos gens per separat (MOZ i ASXH2), ja que com a gen de fusió no apareix. D'aquesta manera s'ha pogut obtenir informació sobre la localització cromosòmica, el nombre i tipus d'exons, el nombre de transcripts i isoformes associades, etc.
Estudi per separat dels dos gens:
El gen MOZ està situat al cromosoma 8 (p11).

Estructura genòmica del transcrit de MOZ:
Aquest gen té un transcrit (ENST00000265713) constituït per 1 exó no codificant (el primer) i 16 codificants, dels quals el primer i el darrer tenen una porció no codificant. El transcript té una longitud de 7850bps, que donen lloc a 2004 aminoàcids. La llista dels exons en ordre es mostra a continuació (mitjançant els seus ID d'Ensembl):

El gen ASXH2 està situat al cromosoma 2 (p23).

Estructura genòmica del transcrit d'ASXH2:

Aquest gen té dos transcrits:
ENST00000336112: constituït per 12 exons codificants, dels quals el primer i l'últim tenen una porció no codificant. El transcript té una longitud de 8886bps, que donen lloc a 1434 aminoàcids. La llista dels exons en ordre es mostra a continuació (mitjançant els seus ID d'Ensembl):
ENST00000272341: constituït per 7 no exons codificants (els 7 primers) i 6 codificants, dels quals el primer té una porció no codificant. El transcript té una longitud de 3781bps, que donen lloc a 918 aminoàcids. La llista dels exons en ordre es mostra a continuació (mitjançant els seus ID d'Ensembl):
Si observem les identificacions dels diversos exons en cadascun dels transcrits del gen ASXH2, podem veure que hi ha només 3 exons en comú. Això és un indicatiu que els dos transcrits es formen per splicing alternatiu.
Observant les seqüències exòniques dels gens MOZ i ASXH2, podem veure que els gens que estan canviats formen part tant de la regió codificant com de la no codificant.
Això ens pot portar a pensar que les dues proteïnes resultants tenen una seqüència aminoacídica diferent, i per tant que les dues proteïnes tinguin una estructura i/o funció diferent.
Per tal de comprovar si l'splicing alternatiu altera la seqüència de la proteïna, fem un aliniament de les dues seqüències aminoacídiques, i observem el resultat obtingut:
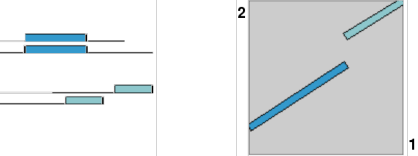
En aquest gràfic les seqüències es representen de la següent manera:
- La seqüència 1 (eix x) correspon a la proteïna resultant del transcrit ENST00000272341
- La seqüència 2 (eix y) correspon a la proteïna resultant del transcrit ENST00000336112.
Analitzant aquestes dades, podem veure que la seqüència 1 és més llarga que la 2, però tot i així el marc de lectura no canvia. La única diferència que hi ha entre els 2 transcrits és que hi ha dues regions en la proteïna 1 no presents en la proteïna 2.
És molt probable que aquestes dues regions no siguin gaire importants per la funció de la proteïna, ja que en una es conserven i en l'altra no. En canvi, les regions comunes en totes dues proteïnes probablement contenen el centre actiu d'aquesta.
S'han dut a terme alineaments de la proteïna quimèrica contra els diferents transcrits per separat, de manera que es pogués observar clarament quins dels dos gens es transcriu primer i quin després i quin dels dos transcrits d'ASXH2 és l'encarregat de donar lloc a la proteïna.
Els resultats es mostren a continuació:
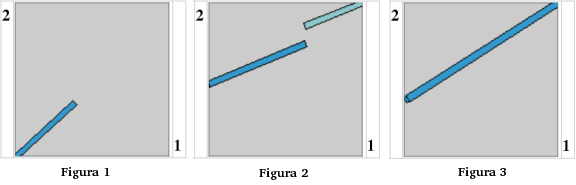
Com es pot observar a la Figura 1, el transcrit al qual dóna lloc el gen MOZ és el que es tradueix en primer lloc per donar la proteïna.
A més a més, a la Figura 2, en el que es mostra l'alineament entre la proteïna quimèrica i el transcrit ENST00000272341 d'ASXH2, s'observa que hi ha una sèrie de bases que són presents a la proteïna quimèrica però que no apareixen en aquest transcrit. D'aquesta manera, es dedueix que aquest transcrit no és el que acaba donant lloc a la proteïna quimèrica juntament amb el produït per MOZ. En canvi, en la Figura 3 es mostra com el transcrit ENST00000336112 sí que dóna lloc a tots els aminoàcids provinents del gen ASXH2 que conformen la proteïna quimèrica.
D'aquesta manera, doncs es pot afirmar que la proteïna quimèrica dels gens MOZ-ASXH2 es forma a partir de l'únic transcrit del gen MOZ i del transcrit ENST00000336112 del gen ASXH2.

Les dades subministrades per Ensembl (www.ensembl.org) inclouen aquells organismes en els quals s'ha trobat homologia (gens ortòlegs) del gen estudiat, a més a més del percentatge d'identitat de l'alineament.
Per confirmar aquestes dades s'ha realitzat un càlcul del tant per cent d'identitat de l'alineament mitjançant Blast, que ha donat el mateix resultat que Ensembl.
Cal destacar, però que hi ha diferents tipus de relacions:
- 1 to 1: ortòlegs. són els que tenen una valors d'identitat més elevats.
- 1 to many: paràlegs. En aquest cas, en general s'obtenen valors força més baixos, fet lògic si es té en compte que sovint es tracta de relacions d'homologia entre espècies molt allunyades filogenèticament d'Homo sapiens. Per tant, s'han inclòs a la taula, però no són ortòlegs realment.
Els resultats obtinguts pel gen MOZ són els següents:
Per tal de confirmar les dades de percentatge d'homologia s'ha realitzat un arbre filogenètic amb el programa MEGA 3.1. Els resultats obtinguts contenen algunes dades que no coincideixen amb els el que esperaríem pels resultats obtinguts d'Ensembl, però tot i així, veiem que l'arbre resultant, s'aproxima bastant a la filogènie de les diferents espècies. El resultat es mostra al següent link:
Veure l'arbre

Els resultats obtinguts pel gen ASXH2 són els següents:
Tal com hem fet amb el gen MOZ, també hem realitzat un arbre filogenètic amb les dades obtingudes a partir de l'Ensembl. Exactament com en el cas anterior, els resultats obtinguts no coincideixen exactament amb el que nosaltres esperàvem, però veiem que la filogènie de les diferents espècies s'aproxima bastant a la realitat excepte en alguns casos.
Veure l'arbre

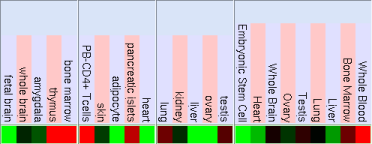
Mitjançant la funció Gene Sorter de la web d'UCSC (www.genome.ucsc.edu) s'ha trobat el patró d'expressió d'amdós gens. Cal tenir en compte el significat dels chips, és a dir, els experiments en que s'analitzen els nivells d'expressió:
- Columnes d'expressió: mostren la ratio d'expressió del gen en els teixits seleccionats o en etapes del cicle cel·lular. El major nivell d'expressió es correspon amb el color vermell i el menor es mostra en verd. Els valors estan en escala logarítmica. El color negre indica que el gen té una valors d'expressió normals en aquest teixit. Aquells espais que apareixen sense colorejar representen manca de dades.
- GNF Expression Atlas 2 Data from U133A and GNF1H Chips: mostra la ID de la prova en l'Atlas de GNF 2 on se superposa la majoria amb el gen escollit. El GNF Atlas 2 es basa en dos chips d'Affymetrix: l'U133A y GNF1H.
- GNF U95 column (Human): mostra les dades de l'Atles d'expressió GNFdel chip U95 d'Affymetrix. Per defecte, la columna mostra la ratio mitjana de l'expressió en un set específic de teixits.
- Max GNF U95 column (Human): mostra l'expressió en nivells absoluts per qualsevol teixit. La majoria dels valors es troben entre 0 i 30.000, però alguns poden arribar a 52.000. Un valor inferior a 20 indica que l'expressió no ha pogut ser detectada en cap teixit a uns nivells significatius (per sobre dels controls d'hibridació creuada).
- U95 ID column (Human): mostra la ID d'Affymetrix del chip HG-U95 que millor es correspon a cada gen.
El patró d'expressió d'aquest gen és en un conjunt de diferents teixits humans es mostra pels següents experiments:
 Expressió màxima
Expressió màxima
 Expressió normal
Expressió normal
 Expressió mínima
Expressió mínima
- GNF Expression Atlas 2 Data from U133A and GNF1H Chips
- Normal Human Tissue cDNA Microarrays
- GNF Expression Atlas 1 Human Data on Affy U95 Chips
- Gladstone Microarray Data Including Stem Cell Tissue
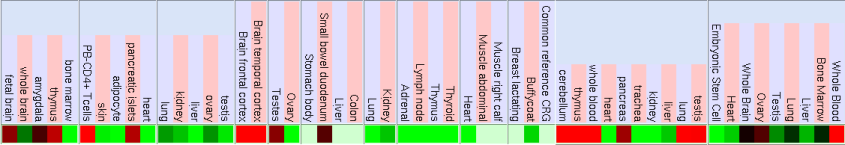
A més a més, mitjançant la funció Visi Gene de la mateixa pàgina web, s'han obtingut les següents imatges de l'expressió del gen MOZ:
Expressió al sistema nerviós central en un embrió de ratolí de 10,5 dies:
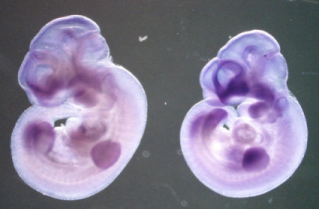
Secció sagital del cervell d'un ratolí de 5 setmanes:
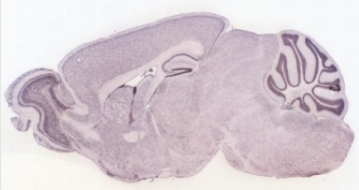
Expressió en diferents secciones del cervell d'un embrió de ratolí de 13,5 dies:
El patró d'expressió d'aquest gen és en un conjunt de diferents teixits humans es mostra pels següents experiments:
 Expressió màxima
Expressió màxima
 Expressió normal
Expressió normal
 Expressió mínima
Expressió mínima
- GNF Expression Atlas 2 Data from U133A and GNF1H Chips
- Gladstone Microarray Data Including Stem Cell Tissue

Per tal de caracteritzar la regió promotora, hem utilitzat dos mètodes i els hem comparat:
- Per una banda, vam realitzar un programa capaç de calcular, a partir d'una matriu de freqüències de nucleòtids per cada posició d'un factor de transcripció, l'score màxim que donava la unió del factor de transcripció a cada posició de la seqüència promotora de cada gen. A més a més, aquest programa ens donava el p-value de cada factor de transcripció en la seqüència promotora donada.
- Per altra banda, vam utiltitzar la pàgina web PROMO, la qual a partir d'una seqüència promotora d'un gen, i la selecció d'una espècie i/o factors de transcripció, dóna com a resultat totes les possibles unions dels diversos factors de transcripció en diferents punts de la seqüència promotora.
Els resultats obtinguts mitjançant el programa pel promotor del gen MOZ són:
| TF |
Score |
Seqüència unió |
Inici |
Final |
p-value |
| AP-1 |
2,9378 |
GAAGTCA |
274 |
280 |
0,08 |
| AR |
2,1172 |
GAACCGC |
42 |
48 |
0,62 |
| c-Myc |
3,4903 |
CACGTG |
732 |
737 |
0,28 |
| NF-AT1 |
3,6660 |
GGAAAGT |
436 |
442 |
0,09 |
| NF-KappaB |
2,4440 |
AGAGTTCCC |
502 |
510 |
0,32 |
| SRF |
-995,7752 |
CCAAGATGG |
981 |
989 |
0,32 |
| YY1 |
2,9867 |
ATGGCG |
986 |
991 |
0,24 |
| RXR-alpha |
2,6311 |
TGAAAG |
285 |
290 |
0,61 |
| HIF-1 |
-995,6667 |
GCCCCACGT |
728 |
736 |
0,47 |
| AhR |
3,5903 |
GCGTGAG |
640 |
646 |
0,07 |
| PU.1 |
3,9140 |
GAGGAAC |
656 |
662 |
0,06 |
| HNF-4 |
-995,2648 |
TGGCCTTT |
713 |
720 |
0,36 |
| NRSF |
-995,9002 |
TCCAGCCCC |
678 |
686 |
0,61 |
Simplement observant els scores obtinguts a través del programa, podem dir que no esperarem que s'uneixin els factors SRF, HIF-1, HNF-4 ni NRSF, ja que el seu valor d'score és molt baix, i indica que la possible seqüència d'unió del factor conté algún nucleòtid no present mai en la seqüència del factor de transcripció, i per tant mai s'hi unirà.
Un altre criteri que podem utilitzar per saber si és probable la unió o no d'un factor de transcripció a la seqüència, és el seu p-value. Aquest valor, és un tant per u que ens indica la significància de l'score obtingut, ja que dóna el nombre de vegades que per atzar obtenim un score superior o igual al que hem obtingut pel factor de transcripció. Per tant, valors baixos de p-value, indicaran que l'score obtingut correspon a una seqüència prou conservada, com per no obtenir el valor de score simplement per atzar.
D'aquesta manera, podríem descartar la possibilitat d'unió dels factors AR, RXR-alpha, HIF-1 i NRSF. Així com podríem sospitar que els factors AP-1, NF-AT1, AhR i PU.1 tenen un valor de p-value prou baix com per ser candidats a unir-se al nostre promotor.
Pel que fa a la resta de factors de transcripció, els valors de p-value no són prou significatius com per arriscar-nos a assegurar o no la seva unió al promotor.
Per comprovar els resultats obtinguts, utilitzarem la pàgina web del PROMO, on s'ens permet seleccionar els factors humans, els llocs d'unió humans i els factors de transcripció utilitzats en el programa realitzat per nosaltres. Un cop li donem la nostra seqüència promotora, li donarem el valor de dissimilarity rate, que permetrà ajustar més o menys la nostra cerca. En la pàgina de resultats, veurem on poden unir-se els diferents factors.
Comparant aquests resultats amb els obtinguts pel nostre programa, podem veure que el PROMO ens mostra molts més possibles llocs d'unió dels factors de transcripció a la nostra seqüència promotora. Entre tots ells, podem comprovar que els factors AP-1, NF-AT1 i c-Myc s'uneixen als mateixos llocs que hem obtingut en el nostre programa, però que tot i això, nosaltres esperàvem veure la unió de Ahr i PU.1, i no és així.
A més a més, nosaltres havíem obtingut un score màxim del factor SRF de -995,7752, i no esperàvem que s'unís al promotor, però en els resultats del PROMO, veiem que ens prediu que sí que s'hi pot unir.
Per tal de veure si els resultats variaven canviant el dissimilarity ratio, el vam disminuir a valors com 10, 5 o fins i tot 1. Els resultats obtinguts van ser la disminució dels possibles llocs d'unió dels diferents factors, i a més a més desapareixien les unions dels factors amb un p-value més baix en contra del que nosaltres esperàvem. Degut a això, ens vam basar en els resultats obtinguts pel valor de dissimilarity ratio 15.
Resultats obtinguts pel promotor del gen ASXH2:
| TF |
Score |
Seqüència unió |
Inici |
Final |
p-value |
| AP-1 |
2,1263 |
AAAGTCA |
403 |
409 |
0,44 |
| AR |
-996,4427 |
GGACTGA |
305 |
311 |
1 |
| c-Myc |
-995,9721 |
CACCTG |
3 |
8 |
0,81 |
| NF-AT1 |
2,6875 |
GGAAAGG |
437 |
443 |
0,5 |
| NF-KappaB |
2,7419 |
AGCTTTCCC |
734 |
742 |
0,23 |
| SRF |
3,5841 |
CCAATATGG |
1035 |
1043 |
0,06 |
| YY1 |
2,2719 |
ATGGTC |
226 |
231 |
0,81 |
| RXR-alpha |
2,4953 |
TGAATC |
250 |
255 |
0,62 |
| HIF-1 |
-995,6601 |
CGCGCTCGT |
945 |
953 |
0,52 |
| AhR |
2,2989 |
GCGTGGC |
876 |
882 |
0,7 |
| PU.1 |
-996,0948 |
GAGGTAC |
883 |
889 |
0,97 |
| HNF-4 |
-995,6078 |
TGGATCTT |
189 |
196 |
0,63 |
| NRSF |
-994,3269 |
TTCAGCTCC |
672 |
680 |
0,07 |
En aquest cas, podem preveure que els possibles factors de transcripció que s'uniran a la nostra seqüència promotora són molt pocs. En primer lloc, podem observar que AR, c-Myc, HIF-1, PU.1, HNF-4 i NRSF tenen un valor d'score molt baix. Això es un primer indici que probablement no s'uniran al promotor. En segon lloc, hem de valorar els p-values dels diferents TF. Per exemple, podem veure que la majoria tenen uns valors molt alts, excepte el NRSF i el SRF. Aquests dos, serien uns bons candidats, però només el SRF ho serà, perquè té un valor d'score positiu enfront el valor d'score tant baix que té NRSF.
Finalment, per valorar els que podem esperar i els que no, farem una valoració entre l'score i el p-value, en aquest cas, podríem esperar l'AP-1, el NF-AT1, l'NF-kappaB i l'SRF.
Per comprovar els factors que s'uniran al promotor i els que no, tal com hem fet abans, utilitzem la pàgina web del PROMO, fent exactament el mateix, però ara donant-li el promotor del gen ASXH2.
Estudiant els resultats obtinguts pel promo i comparant-los amb els obtinguts pel nostre programa, podem comprovar que tant l'AP-1 com el NF-AT1 s'uneixen a la seqüència promotora en els llocs que ens ha predit el nostre programa, però l'SRF que era un molt bon candidat, no s'hi uneix, ni en la seqüència que ens ha predit el programa ni en tot el promotor. En canvi, no esperàvem que s'unís l'YY1 degut al seu p-value tant elevat, però si que ho fa, i en els llocs predits. A més, tampoc experàvem la unió del RXR-alpha al nostre promotor, però veiem que sí que s'hi uneix tot i que no ho fa en la seqüència que haviem predit.
Tot això ens demostra, que la naturalesa és molt capritxosa, i no segueix uns patrons exactes, i sense experimentar totes les possibilitats, no podem assegurar al 100% si un factor de transcripció s'uneix a un promotor o no, i a més a més tampoc podem assegurar en quin lloc ho fa.
El cas del factor de transcripció NRSF, havíem predit un lloc d'unió en tots dos promotors, que tot i tenir un score molt negatiu, tampoc ho hem pogut comprovar, perquè el promo no ens donava la opció de seleccionar aquest factor de transcripció per testar la seva unió als promotors.

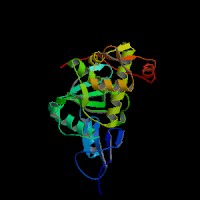
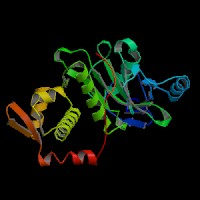
Per tal de veure els dominis de la proteïna, vam cercar a la base de dades InterPro la ID Q76L81 corresponent a la proteïna quimèrica. En aquesta pàgina vam veure que la proteïna que estem estudiant consta de 5 dominis diferents tal i com es mostra en la figura inferior.

Si fem la cerca d'aquests dominis en la base de dades d'InterPro buscant les referències, obtindrem que els dominis que conté la proteïna quimèrica són:
- Domini RING-finger (imatge). És un tipus especialitzat de domini zinc-finger, format per 40-60 residus que s'uneixen a dos àtoms de zinc, i probablement està involucrat en la mediació d'interaccions proteïna-proteïna.
- Domini PHD. És un homeodomain finger semblant al motiu zinc finger, i es troba en proteïnes nuclears que es creu que estan involucrades en la regulació transcripcional mediada per cromatina. Tot i que encara té una funció desconeguda, per analogia es creu que pot estar implicat en interaccions proteïna-proteïna i ser important per la unió o activitat de complexes importants en l'activació o repressió transcripcional.
- Domini MOZ_SAS. Tal com hem explicat, Moz és una proteïna zinc finger de la leucèmia monocítica, i la proteïna SAS de S.cerevisiae està implicada en el silenciament del locus Hmr. Es creu que aques proteïnes són homòlogues a les acetiltransferases, però aquesta similaritat no està soportada per anàlisis estandards de seqüències.
- Domini H15. La histona enllaçant H1 és un component essencial de l'estructura de la cromatina. Aquesta histona uneix nucleosomes formant una estructura d'ordre superior. Pel que fa a la histona H5, fa la mateixa funció que l'H1, i la reemplaça en certs tipus cel·lulars. El plegament és similar al domini d'unió al DNA de la proteïna CAP, proveïnt així un possible model d'unió GH5 al DNA.
- Domini Wing_hlx_DNA_bd. Les proteïnes winged helix DNA-binding comparteixen un motiu relacionat amb unió a DNA helix-turn-helix, on les "ales" (wings) són loops o petites fulles beta. Aquests motius consisteixen en dos 2 ales (W1,W2), tres hèlix alfa (H1, H2, H3) i 3 fulles beta (S1, S2, S3), ordenades de la següent forma: H1-S1-H2-H3-S2-W1-S3-W2. L'hèlix de reconeixement de DNA, fa que els contactes específics de seqüències de Dna siguin amb el solc major de l'hèlix de DNA mentre les "ales" fan diferents contactes amb DNA usualment amb el solc menor o l'esquelet del DNA. Algunes proteïnes que contenen aquests dominis, mostren un seguit de residus hidrofòbics que es creu que medien interaccions proteïna-proteïna.
Per finalitzar amb l'estudi de la funció de la proteïna quimèrica, vam fer una cerca a la base de dades del geneontology, mitjançant la cerca de les identificacions que vam trobar referides a la nostra proteïna. Les diferents ontologies obtingudes en la cerca estan relacionades en la següent taula juntament amb les corresponents identificacions i links de cadascuna de les ontologies.
| ID GO |
Ontologia |
Nom |
| GO:0000786 |
Component cel·lular |
Nucleosoma |
| GO:0005634 |
Component cel·lular |
Nucli |
| GO:0003677 |
Funció molecular |
Unió a DNA |
| GO:0005515 |
Funció molecular |
Unió a proteïna |
| GO:0016740 |
Funció molecular |
Activitat transferassa |
| GO:0008270 |
Funció molecular |
Unió a ions de zinc |
| GO:0016568 |
Procés biològic |
Modificació de cromatina |
| GO:0006334 |
Procés biològic |
Ensamblatge de nucleosomes |
| GO:0006355 |
Procés biològic |
Regulació de la transcripció dependent de DNA |

Pàgines web:
Biomart: http://www.biomart.org/
BLAST: http://130.14.29.110/BLAST/
Clustalw: http://www.ebi.ac.uk/clustalw/
Ensembl: http://www.ensembl.org
Genome Browser: http://genome.ucsc.edu/cgi-bin/hgGateway
InterPro: http://www.ebi.ac.uk/interpro/
NCBI: http://www.ncbi.nlm.nih.gov/
Programa PROMO: http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3
UCSC: http://genome.ucsc.edu/
UniProt: http://www.uniprot.org/
PubMed:http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=PubMed
GeneOntology:http://www.geneontology.org/
VisiGene:http://genome.ucsc.edu/cgi-bin/hgVisiGene?command=start
Articles:
Toshihiko Imamura, Naoki Kakazu, Shigeyoshi Hibi, Akira Morimoto, Yoko Fukushima, Ikuko Ijuin, Satoshi Hada, Issei Kitabayashi, Tatsui Abe, Shinsaku Imashuku. Rearrangement of the MOZ Gene in Pediatric Therapy-Related Myelodysplastic Syndrome With a Novel Chromosomal Translocation t(2;8)(p23;p11). Genes, chromosomes & cancer. 2003; 36:413-19.





































 Expressió màxima
Expressió màxima Expressió normal
Expressió normal Expressió mínima
Expressió mínima






