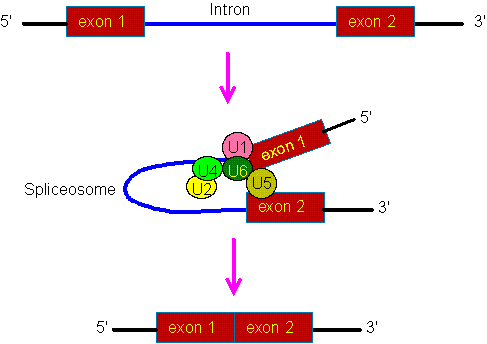
El proceso de splicing consiste en la eliminación de intrones y la
unión de exones dando lugar a un tránscrito primario (pre-mRNA). Los
intrones contienen señales reconocidas por un complejo proteico, el
spliceosoma, encargado de llevar a cabo el splicing. El spliceosoma
está formado por cinco snRNAs (small nuclear RNAs) y sus
proteínas asociadas. El mRNA se obtiene a partir de dos reacciones
enzimáticas, en las cuales el intrón es eliminado y los dos exones
vecinos son unidos. En la siguiente figura vemos un esquema de la
formación del spliceosoma durante el proceso de splicing del
RNA.
RNA Splicing

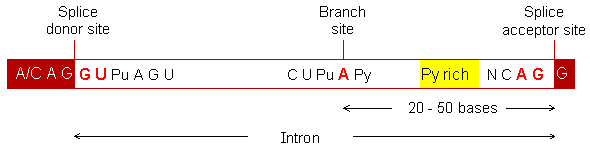
La mayoría de intrones tienen las secuencias de reconocimiento
conservadas, se inician en 5' con la secuencia GU y finalizan en 3' con
AG, conocidas como donor site y acceptor site respectivamente. Por otra
parte, para el reconociemiento de intrones, es necesaria otra secuencia
localizada 20-50 bases upstream del acceptor site, ésta es conocida
como branch site. Su secuencia conseso para los vertebrados es
"CU(A/G)A(C/U)", donde A se encuentra conservada en todos los genes. Se
ha visto que en más de un 60% de los casos, la secuencia exónica es
(A/C)AG en el donor site, y G en el acceptor site. En la siguiente
imagen podemos ver la estructura típica exón-intrón en vertebrados.
En el genoma de levaduras, la presencia de intrones en genes nucleares es poco frecuente,(5% tienen intrones), principalmente en el extremo 5' de los ORFs, sin presentar una elevada conservación en su secuencia.
Gran parte de los intrones en levadura presentan el hexámero GTATGT en el extremo 5' y preferentemente TAG o CAG, pero nunca GAG, en el extremo 3'. Adicionalmente muchos de estos intrones también presentan la secuencia TACTAAC en el branch site.
Aquellos intrones que difieren de este patrón consenso, deben estar regulados por señales adicionales que promueven el reconocimiento y el splicing en el intrón. Este mecanismo debe ocurrir por la unión de moléculas a las señales intrónicas.
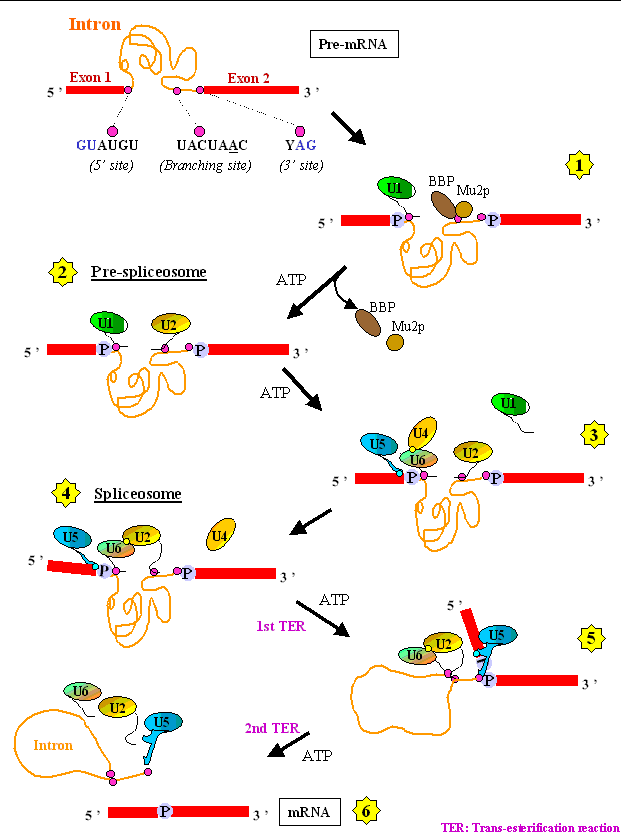
La eliminación de intrones desde los pre-mRNAs involucra un
mecanismo molecular similar en todos los eucariotas. En muchos de estos
casos, este proceso está mediado por el "spliceosoma
U2-dependiente", un complejo ribonucleico compuesto por cinco small
nuclear RNAs (snRNAs) filogenéticamente conservadas (U1,U2,U4,U5 y U6),
ensambladas dentro de pequeñas partículas ribonucleo-proteícas
(snRNPs), además de a un conjunto de proteínas transitorias asociadas
(RNA helicasas, SR splicing factors, etc.). Según el modelo
tradicional, el spliceosoma es un complejo altamente dinámico, el cual
junta los intrones del mRNA a través de un proceso secuencial
("A stepwise assembled complex"). Sin embargo, trabajos recientes han
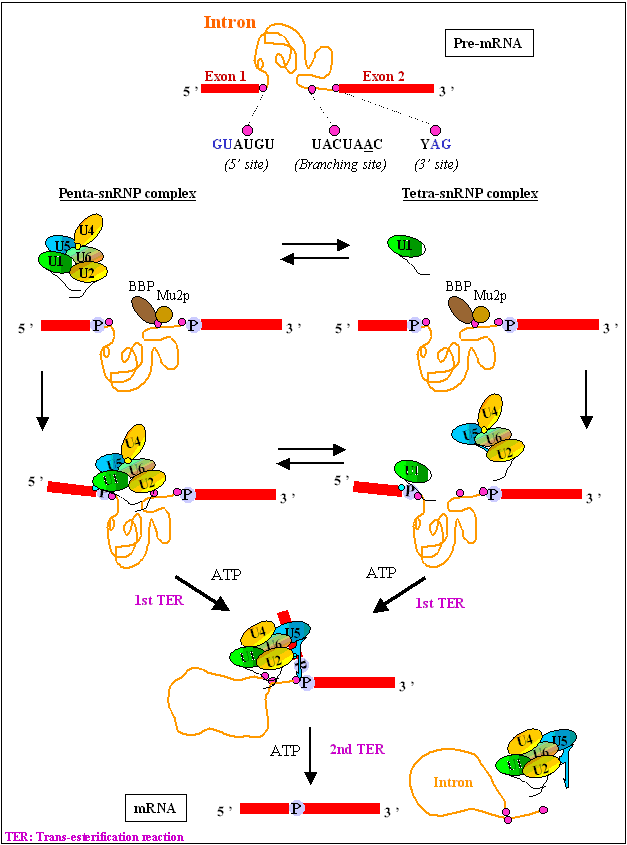
aportado nuevos conocimientos sobre el mecanismo de ensamblaje del
splicing en los eucariotas. Se ha demostrado, que en
levaduras, el snRNPs spliceosoma forma un pre-complejo penta-snRNP
(45S) previamente a la unión al tránscrito de mRNA ("A preassembled
complex").
| Modelo clásico: A stepwise assembled complex |
| Modelo moderno: A preassembled complex |
El spliceosoma está organizado, al menos en parte, por la complementariedad de bases del RNA entre los snRNAs y algunos sitios dianas específicos localizados en 5' y 3' terminales del intrón (5' y 3' splice sites) y en una posición interna cerca del 3' terminal del intrón (branch site).
La reacción de splicing es autocatalítica y se lleva a cabo vía dos reacciones de transesterificación consecutivas. En la primera reacción el splice site 5' es cortado y el intrón del 5' terminal es ligado al branch site concomitantemente. En la segunda reacción, el corte del splice site 3' deja al intrón en una estructura de lazo, y los exones 5' y 3' son ligados.
El objetivo de nuestro proyecto es la búsqueda de señales conservadas que puedan actuar como responsables del reconocimiento y del splicing, en aquellos intrones donde las secuencias de los splice y branch site difieren de la consenso.
