 |

En primer lloc, ens ha interessat estudiar com es distribueixen els SNPs en els cromosomes; per a fer-ho, hem utilitzat el programa contachr.pl , que ens calcula el nombre de SNPs que cauen en cadascun dels cromosomes d'humà. D'aquesta manera, podrem valorar si realment es correspon amb la longitud del cromosoma i conseqüentment amb el nombre de gens que aquest conté.
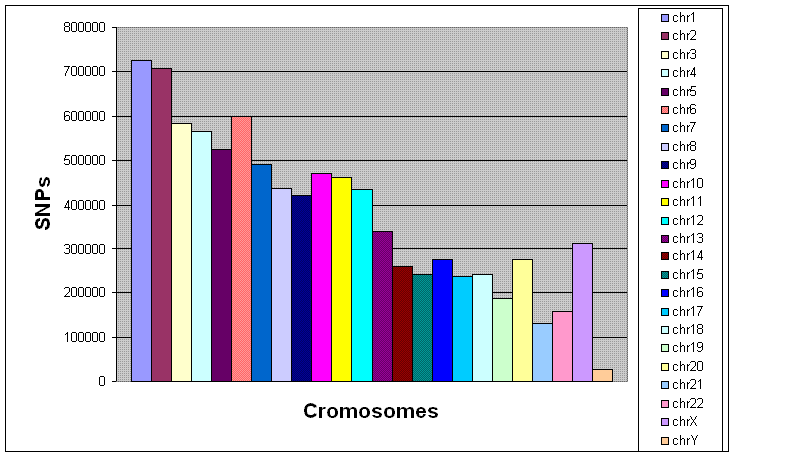
Així, en el gràfic es pot observar que, per línia general, a mesura que disminueix la longitud de cada cromosoma en qüestió també decreix el % d'SNPs que s'hi troben.
Per exemple en els cromosomes pels quals s'han descrit més nombre de gens, els cromosomes 1 i 2; s'hi troba un % d'SNPs molt més elevat que no pas en els cromosomes 22 i Y, la densitat gènica dels quals és bastant baixa.
Tot i això, s'observen algunes exepcions: els cromosomes 6, 10, 11 i 12 i 21 són més rics en SNPs del que esperaríem pel seu tamany. No trobem cap explicació biològica al fet que aquests cromosomes s'escapin de la línia general de distribució, doncs assumim que la selecció natural actua amb igual força en cada cromosoma. Probablement, hom pot pensar que els esforços destinats a trobar SNPs poden haver estat diferencials per cromosomes; de manera que potser aquesta és la causa de veure desviacions a la regla general.
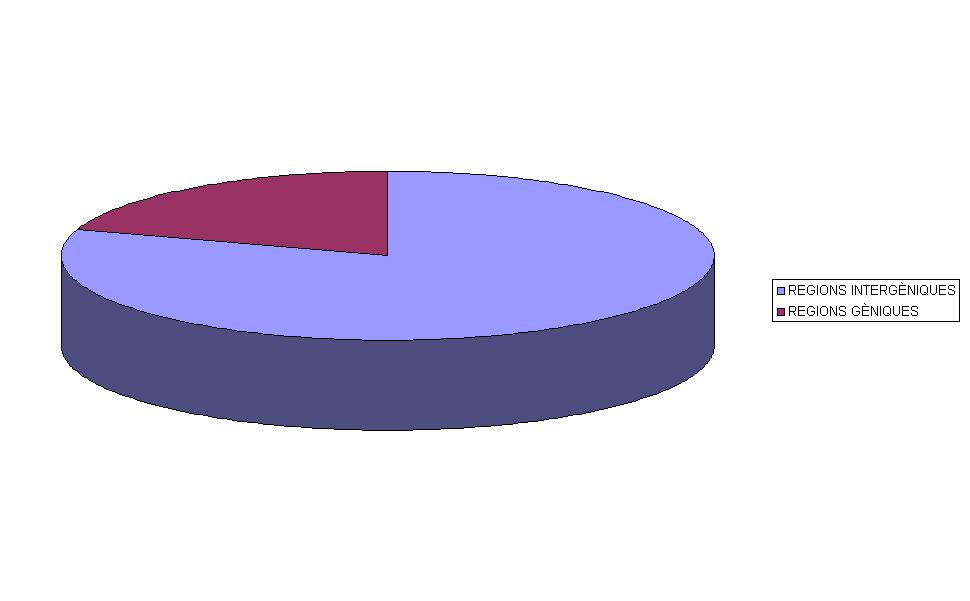
Seguidament, utilitzant el nostre programa per buscar la localització dels SNPs segons la regió genòmica, obtenim els següents resultats:
| Regions intergèniques | Regions gèniques |
| 66,9654086 | 16,7651775 |
 |
| Introns | 15,5998984 |
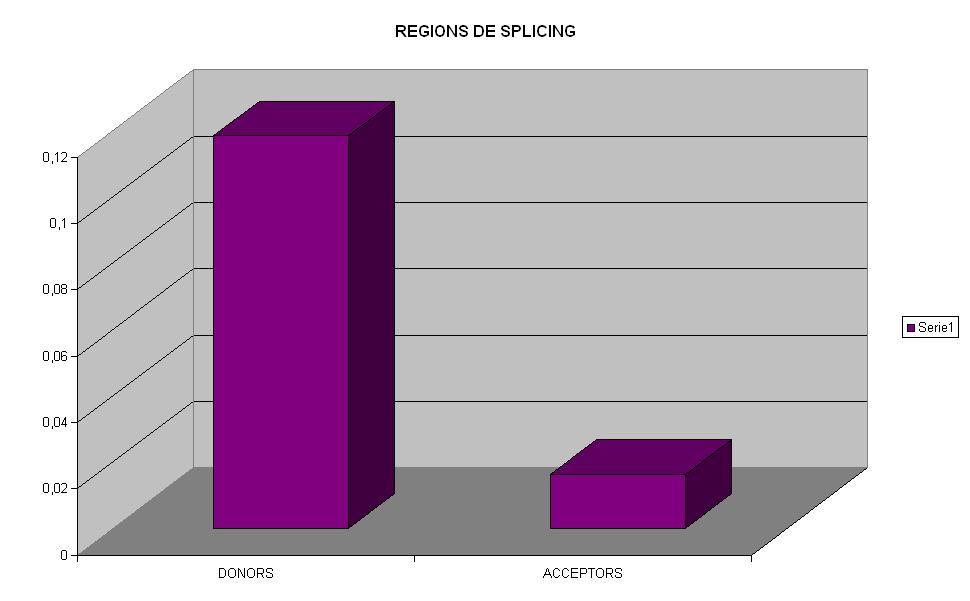
| Donors | 0,1185211 |
| Acceptors | 0,0164916 |
| CDS | 0,5146799 |
| 5'UTR | 0,2042447 |
| 3'UTR | 0,3113418 |
 |
| Donors | 0,1185211 |
| Acceptors | 0,0164916 |
 |
D'aquests resultats en podem extreure les següents conclusions:
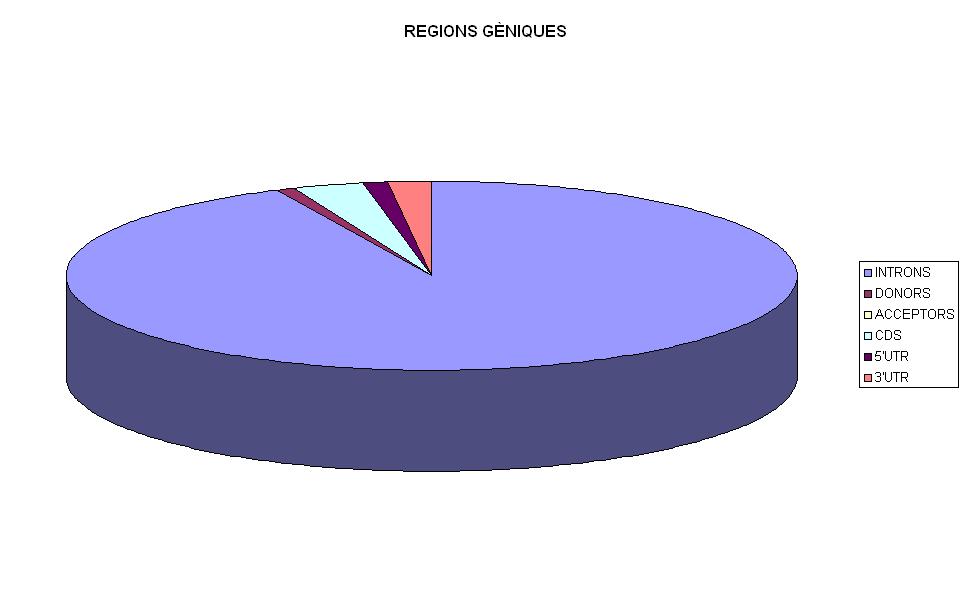
En primer lloc observem una distribució molt més àmplia de SNPs en les regions intergèniques (67%) que en les regions gèniques (16%), una distribució pràcticament igual a la que després observarem en gens no relacionats amb malalties. Esperem trobar aquests valors perquè és lògic trobar més variacions en aquelles regions del genoma que no contenen gens i que no han de codificar per a proteïnes importants per a les funcions biològiques.
Si ens centrem en les regions gèniques, observem, com caldria esperar, que les regions que presenten un major percentatge de SNPs són les regions intròniques, que no codifiquen per proteïna.
Sí que s'observa una diferència interessant entre les regions 5'UTR i 3'UTR, ja que esperaríem valors similars. Trobem menys % de SNPs en la regió 5'UTR i això podria ser degut a que és aquesta regió gènica on hi té lloc la unió de la maquinària per a dur a terme la transcripció del gen. Per tant, això implicaria que petits canvis nucleotídics en aquesta part del genoma podríem comportar la mala unió d'aquestes proteïnes i la no transcripció del gen. Aquest podria ser el motiu per el qual la regió 5'UTR es trobar més conservada pel que fa a la presènica de polimorfismes.
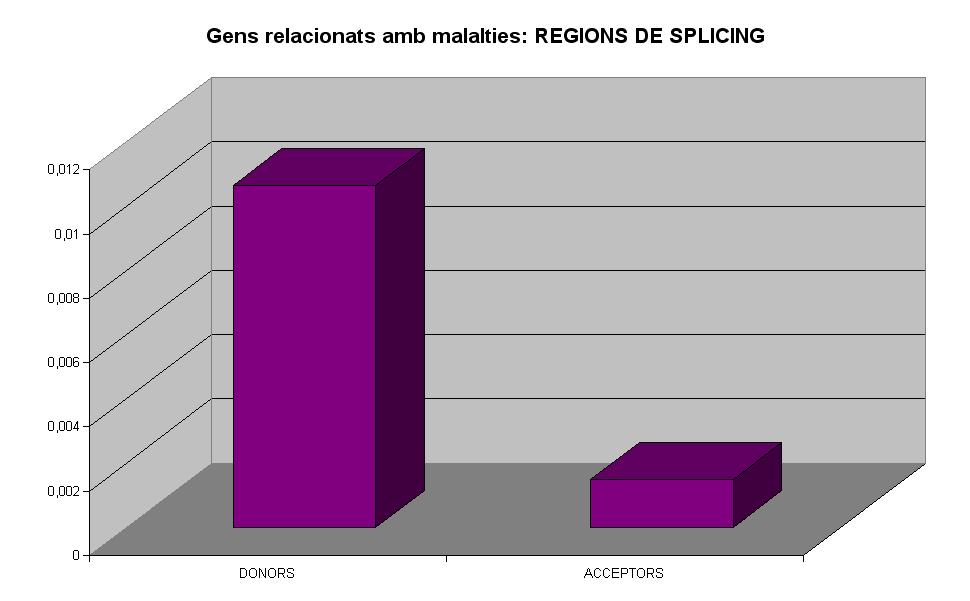
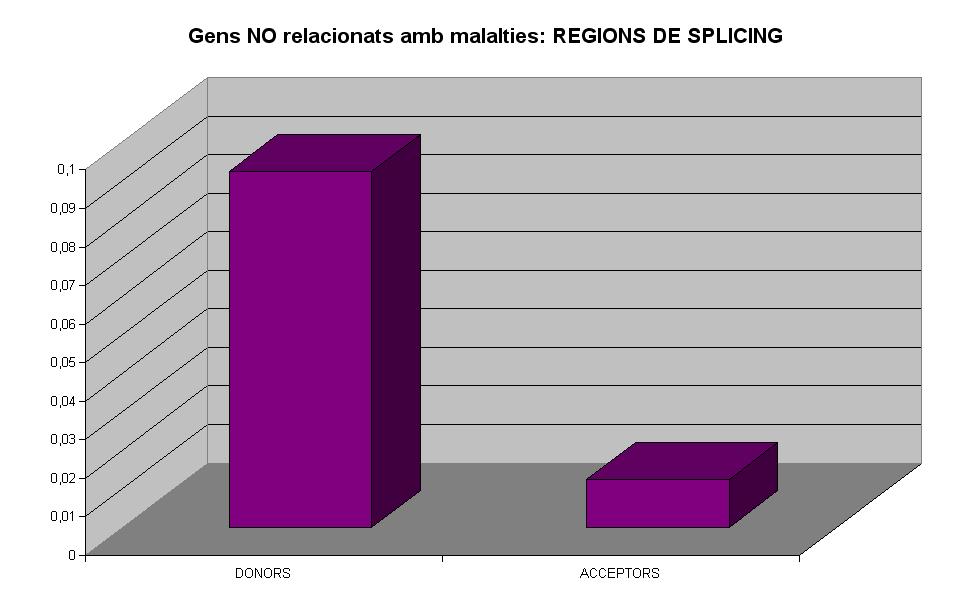
També podem observar un fet similar en les regions de splicing. Els acceptors es troben més conservats pel que fa a canvis nucleotídics que els donors. Això ens pot portar a pensar que la seqüènica que determina l'acceptor és molt més important i necessita que molts més nucleòtids és trobin conservats al llarg de la població per tal que el mecanisme de splicing es pugui donar correctament.
Distribució de SNPS en gens relacionats i no relacionats amb malalties
Ens ha motivat analitzar si es poden observar canvis en la distribució dels SNPs en gens que han estat relacionats amb algun tipus de disfunció biològica o malaltia i amb els que no ho han estat.
Amb aquest propòsit hem utilitzat el programa ppro.pl però ara, el fitxers d'entrada han estat: en primer lloc, els gens relacionats amb malalties i en segon lloc els gens no relacionats amb malalties.
Per a cadascun dels fitxers d'entrada hem obtingut un output: relacionats.out , i norelacionats.out,gràcies als quals hem pogut elaborar els següents gràfics.
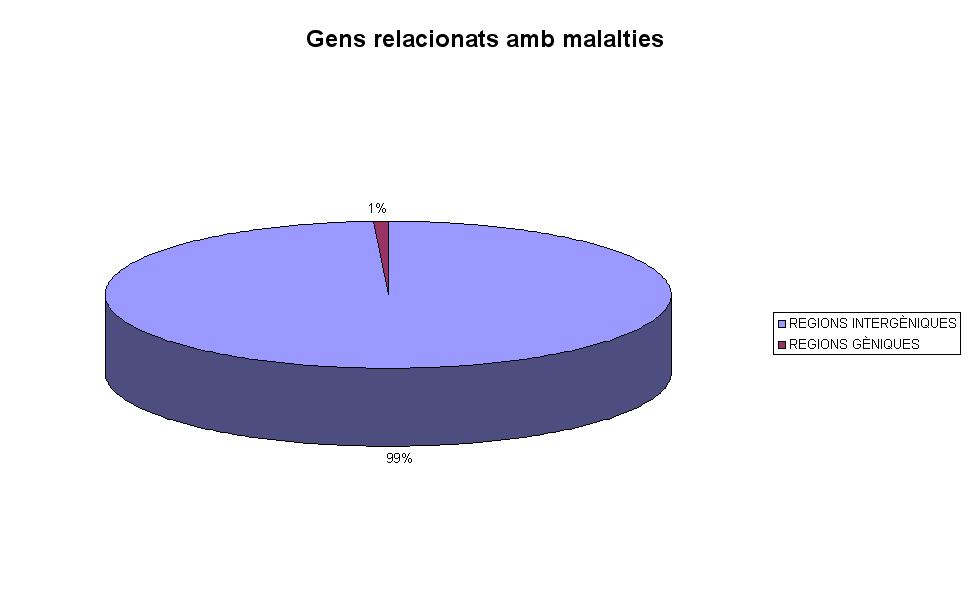
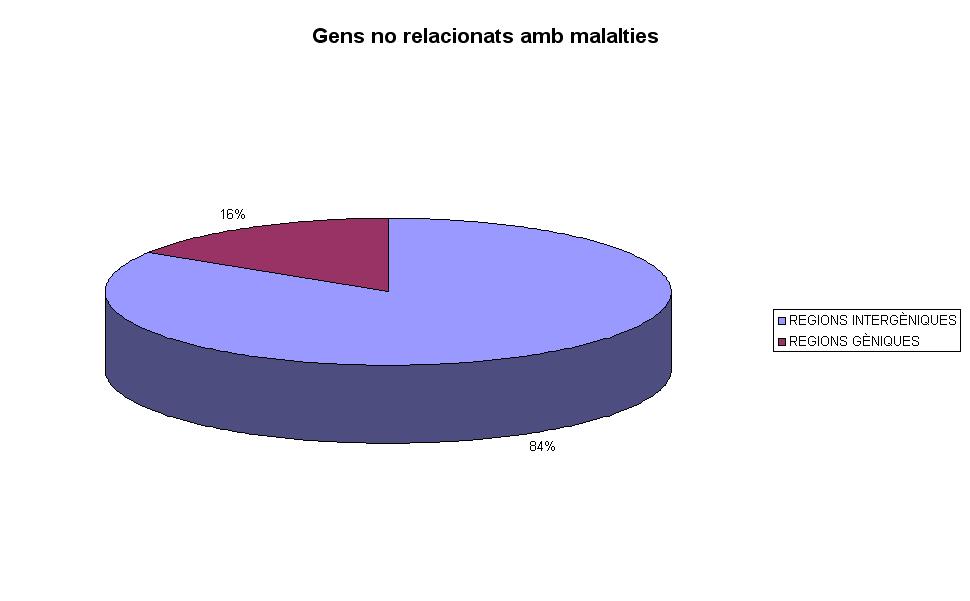
Pel que fa a la proporció de SNPs en regions intergèniques i en regions que contenen gens veiem que s'observa variació.
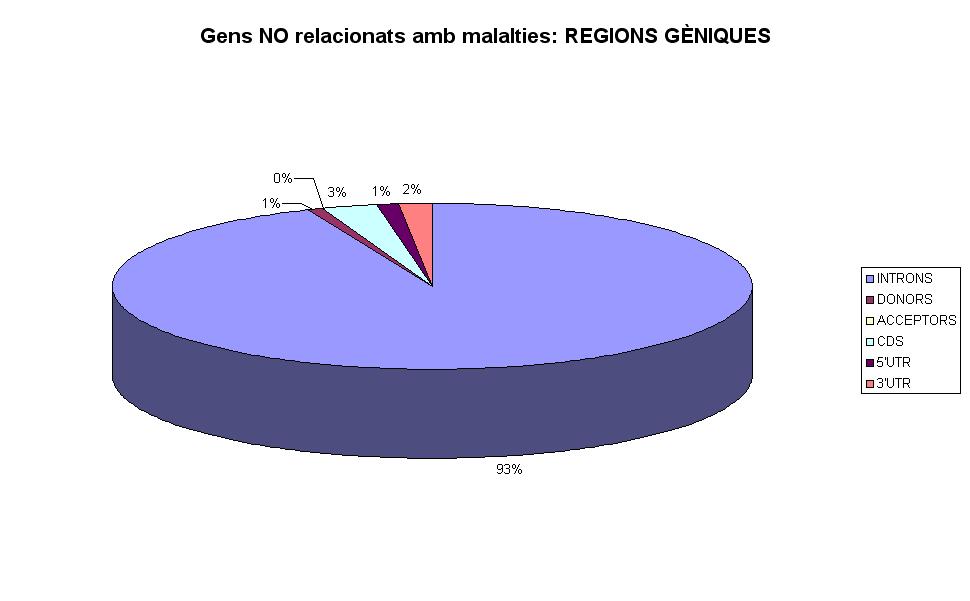
Els gens relacionats amb malalties presenten tan sols un 1% de SNPs en regions gèniques mentre que en gens no relacionats amb cap alteració biològica hi trobem un 16%.
D'aquesta manera, veiem que els gens que estan relacionats amb malalties, d'entrada presenten menys % SNPs en regions que contenen gens i pràcticament el 99% dels SNPs cauen en regions intergèniques.
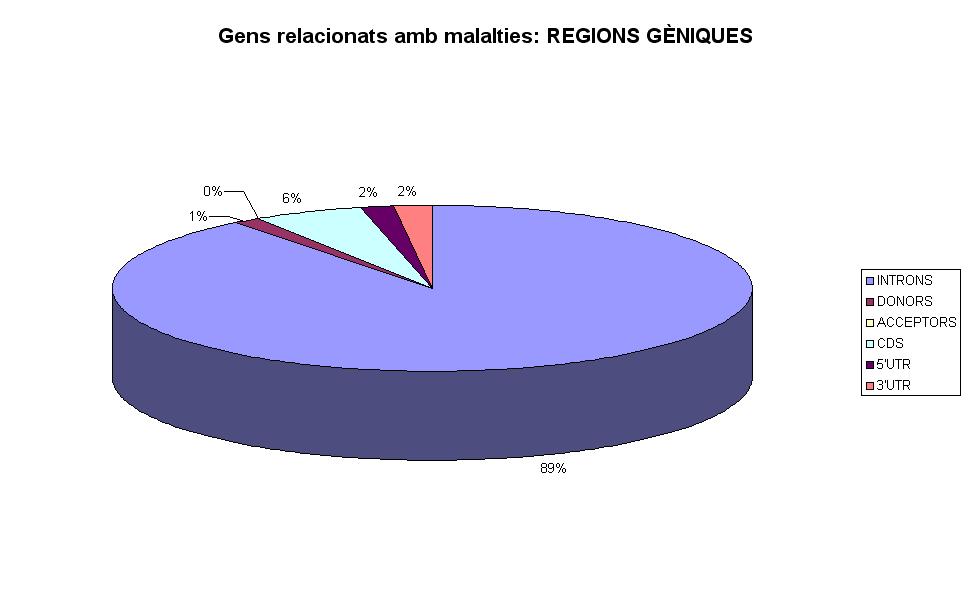
D'altra banda, i centrant-nos ja en com es distribueixen els SNPs en les 2 classes de gens diríem que:
- el % d'SNPs que cauen en introns és major en gens no relacionats amb malalties (93%) que no pas amb els relacionats amb malalties (89%).
- el % d'SNPs trobats a llocs d'splicing és major (4%) en gens relacionats amb malalties que no pas en aquells que no estan relacionats amb malalties (3%) i en els 2 casos s'observa més percentatge d'SNPs en donor splice sites en comparació amb els acceptor splice sites.
- el % de SNPs presents en exons és major en gens relacionats amb malalties, 7%, enfront un 4% en gens no relacionats amb malalties.
Analitzant la regió dins els exons que presenta més SNPs s'observa:
- En els 2 casos la proporció de SNPs dins regions exòniques és de major a menor : CDS > 3'UTR > 5'UTR.
- Cal destacar que en el cas de gens no relacionats amb malalties, tan sols la meitat dels SNPs situats en exons els trobem en CDSs en canvi en gens relacionats amb malalties pràcticament tots els SNPs estan situats en CDS en comparació amb els presents a 5'UTR i 3'UTR.
Una vegada analitzats els resultats obtinguts podem extreure una sèrie de conclusions:
Primerament que els gens relacionats amb malalties presenten menys polimorfismes de nucleòtid únic en regions gèniques. Això vertaderament ens sorpren molt perquè hom esperaria trobar tot el contrari. Pensavem trobar un % més baix d'SNPs en regions intergèniques en aquests gens perquè suposavem que probablement el fet de tenir polimorfismes en regions gèniques podria ser el causant d'algunes malalties i per tant, n'hauriem de trobar més en gens que han estat relacionats amb malaties.
En segon lloc, és interessant fixar-nos en el fet que els gens que han estat relacionats amb malalties tenen un % d'SNPs més elevat en llocs d'splicing.
L'splicing no és res més que el processament que pateix el pre-ARNm resultant de la transcripció, després de ser modificat amb el capping, la metilació i la cua de poliadenilació, per tal de donar lloc a un ARNm madur. Gràcies a les regions donadores i acceptores d'splicing, les quals estan altament conservades, tota la maquinària que participa en aquest procés pot extreure els introns del transcrit primari i així obtenir l'ARN madur, el qual pot estar format per diferents combinacions d'exons, que serà posteriorment traduït a proteïna.
Així, un elevat grau de polimorfisme en les regions donadores i acceptores de splicing pot conduir a un mal processament de l'ARN inmadur i conseqüentment a la formació d'una proteïna no funcional.
Alguns exemples de malalties relaciones amb presència de SNPs a donor sites i aceptor sites serien:
Amb aquest estudi hem pogut corroborar aquest fenomen; un grau de polimorfisme elevat en llocs d'splicing comporta producció de proteïnes no funcionals i per tan, veiem més proporció de SNPs en donor i acceptor sites en gens que han estat relacionats amb malalties.
D'altra banda, també podem corroborar que els gens relacionats amb malalties presenten un grau de polimorfisme més elevat en regions codificants per proteïnes, fet suposable donat que polimorfimes en CDS poden provocar canvis en el marc de lectura (introducció de codons stop, canvis en codons que comportin canvis en l'aa... ) que condueixin a la traducció de proteïnes que no poden realitzar la seva funció correctament i per tan que comportin disfuncions fisiològiques.
Finalment i a mode de conclusió diríem que els gens que han estat relacionats amb malaltia presenten més variacions en regions amb més importància biològica, principalment les implicades en la síntesi i el processament proteic, que no pas els gens que no han estat relacionats amb malaties.

 |  |  |