Obtenció d'elements repetitius
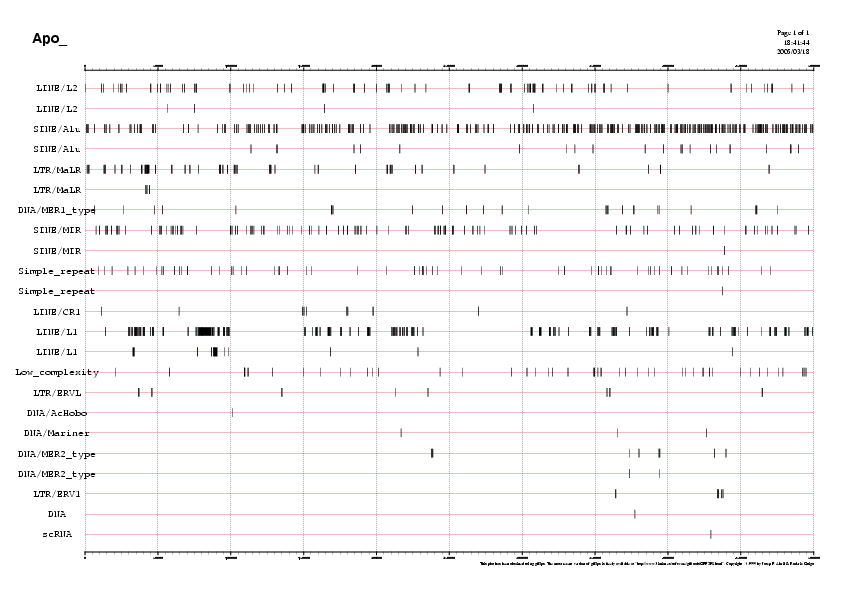
L'arxiu Apo.seq.tbl mencionat anteriorment, conté la distribució de les repeticions al llarg de la seqüència.
Aquesta arxiu també ens presenta el contingut de GC que conté la seqüència i que és d'un 43.78%, així com també ens indica que en un 41,73%,la seqüència ha esdevingut emmascarada. Aquest elevat percentatge d'elements repetitus permetrà un anàlisi més ràpid de la regió.
Per poder visualitzar la distribució de les repeticions,es va passar l'arxiu Apo.seq.out a format gff, després a format ps i, per últim, a png .
El passos que es van seguir són els següents:
- Es van exportar els programes al nostre directori per poder treballar amb ells amb la comanda:
export PATH=/disc8/soft/perl/bin/:/disc8/bin/:$PATH
- Es va passar l'arxiu a format gff amb la següent comanda i es van mirar les repeticions a amb el gff web server.
grep Apo_ Apo.seq.out | \
> awk 'BEGIN{ OFS="\t" }
> { print $5, $11, "repeat", $6, $7, ".", ".", "."; }
> ' > Apo.seq.out.gff
- A continuació es va transformar l'arxiu a format ps mitjançant el programa gff2ps, posant al shell la següent comanda:
gff2ps Apo.seq.out.gff > Apo.seq.out.ps
- El pas de format ps a png es va realitzar amb l'ordre:
convert -antialias -rotate 90 Apo.seq.out.ps Apo.seq.out.png
- Per últim, es visualitzen els elements repetitius de la seqüència ENm003 Apo Cluster amb el programa kview:
kview Apo.seq.out.png
Inici