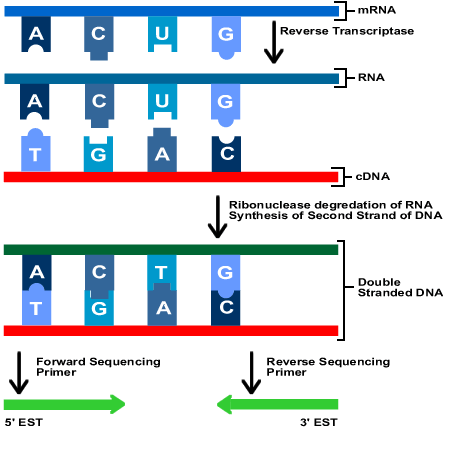
|
INTRODUCCIÓN:
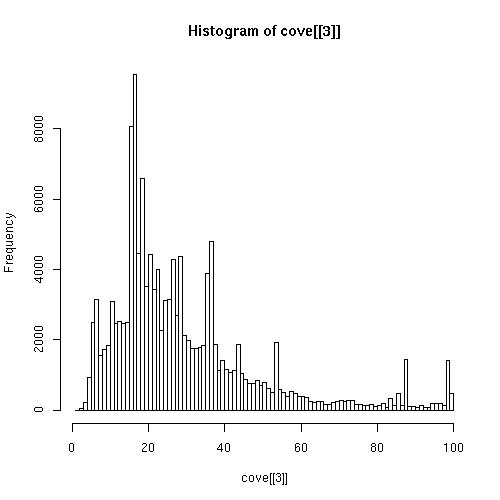 |
GRÁFICO 1
A partir del listado de EST-coverage y utilizando las comandas correspondientes del UNIX, conseguimos respresentar la frecuencia de EST en función del coverage que presentan. En esta gráfica vemos que la mayor parte de los EST se distribuyen alrededor de un valor random, que es del 20% de coverage, con ciertas variaciones. El pequeño grupo de secuencias que se halla a la derecha del valor random, es el que presenta un puntuación mayor, y por lo tanto son los que están más relacionadas con nuestras secuencias humanas. Por esto hemos definido el umbral a partir de un 80% de coverage, ya que creemos que estos serán los mejores EST para trabajar. |
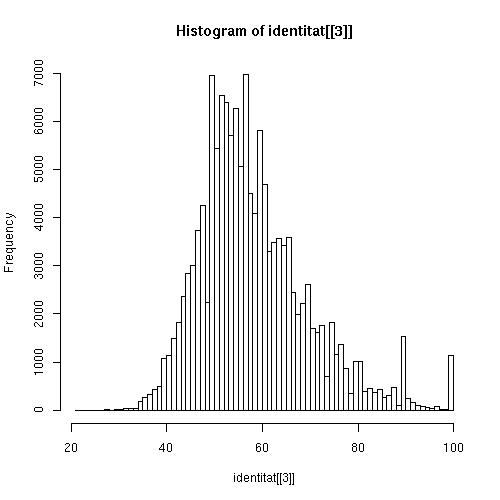 |
GRÁFICO 2
A partir del fichero que contiene EST-porcentaje de identidad y, utilizando también comandas del UNIX, representamos la frecuencia de EST que presentan un determinado porcentaje de identidad. En esta gráfica los mayores porcentajes de identidad se situan alrededor de 50. Por ello, hemos decidido establecer el umbral a partir del 60% de identidad. |
Otro fichero que queremos destacar es el que contiene los genes humanos con el EST que se ha alineado e información sobre la función y localización de este gen y, por lo tanto, que podría corresponder al EST.
En la siguiente tabla presentamos los 9 ESTs finales con su correspondiente identificador de gen humano y con su posible función asociada. Clicando sobre el gen podemos acceder al fichero del que proceden y clicando sobre Link obtenemos más información sobre el gen.
| BB160005A20G10 | ENSG00000013503.1 | RNA Polymerase III, Subunit 2 (RPC2) | |
| BB160013A10A03 | ENSG00000186907.1 | Reticulon 4 receptor-like 2 (RTN4RL2) | |
| BB160014B20B10 | ENSG00000129315.1 | Cyclin T1 | |
| BB160019A10F10 | ENSG00000170364.1 | Set domain and mariner transposase fusion gene | |
| BB160020B20F06 | ENSG00000065108.1 | Ganglioside induced differentiation associated protein 2 | |
| BB170008A10C08 | ENSG00000080802.4 | CCR4-NOT transcription complex, subunit 4 (CNOT4) | |
| BB170016B10H03 | ENSG00000021574.2 | Spastin
Fidgetin-like 1 (FIGNL1) |
|
| BB170029B10G04 | ENSG00000066926.1 | Ferrochelatase, mitochondrial precursor (Protoheme ferro-lyase)(Heme synthetase) | |
| BB170031A20F07 | ENSG00000115760.1 | Baculoviral IAP repeat-containing protein 6(Ubiquitin-conjugating BIR-domain enzyme apollon) |
CONCLUSIONES:
En este proyecto hemos trabajado con ESTs del cerebro de Apis mellifera, por lo tanto, en un principio esperábamos en los resultados, que la mayor parte de los ESTs se asociaran con genes humanos expresados en cerebro. Hemos comprobado que en realidad no es así. Nuestros ESTs se han alineado con cDNA que se halla en múltiples partes del organismo; obtenemos genes de expresión genital, endocrina, respiratoria, cardiovascular, urinaria, alimentaria, dermal, linforeticular o musculoesquelética. Probablemente esto sucede debido a la corta longitud de los ESTs utilizados. Al ser pequeños se alinean con cDNAs que no son su secuencia homóloga, sino que comparten alta similitud de bases.
El análisis del fichero que contiene únicamente los genes de cerebro, los ESTs y la anotación nos indica que no hay especificidad de lugar, es decir, que estos genes también se encuentran en otras zonas del organismo. Además el número de genes era demasiado elevado para hacer un estudio detallado.
Debido a esto hemos decidido profundizar en las anotaciones del gen y buscar genes específicos de áreas concretas del cerebro. Antes de hacer esto eliminamos los genes relacionados con neoplasias ya que nos podrían dar comparaciones funcionales erróneas.
Las zonas utilizadas para estudiar la expresión han sido cerebelo, líquido cefaloraquídeo, encéfalo, diencéfalo, médula oblongata, meninges, cerebro medio, sistema ventricular...Sólo en encéfalo hemos encontrado genes que se expresan exclusivamente allí y en ninguna otra parte cerebral.
De todos los ESTs analizados, son nueve los que se hallan en encéfalo. Estos presentan diferentes funciones, algunas de ellas todavía en investigación.
A continuación presentamos un listado del EST con su correspondiente identificador humano y la función que presenta este gen. De este modo se puede inferir la posible función de la secuencia que representa el EST.
- BB160005A20G10-ENSG0000013503.1: presentan un 94% de identidad y un 93,67% de coverage. Se trata de una RNApolimerasa dependiente de DNA que cataliza la transcripción de DNA a RNA usando los ribonucleotidos coma sustrato. En humano se localiza en el cromosoma 12.
- BB160013A10A03-ENSG00000186907.1: presentan un 80% de identidad y 81,03% de coverage. Es una secuencia homóloga al receptor NOGO-66 y se halla en el cromosoma 11.
- BB160014B20B10-ENSG00000129315.1: presentan un 76% de identidad y un 98,72% de coverage. Es la subunidad reguladora de la kinasa dependiente de ciclina CDK9 y por lo tanto es muy importante en ciclo celular. Se encuentra en el cromosoma 12 humano.
- BB160019A10f10-ENSG00000170364.1: tienen un 66% de identidad y un 89,58% de coverage. Se trata de un dominio SET y un gen de fusión a un transposón de tipo mariner. Se halla en el cromosoma 3.
- BB160020B20F06-ENSG00000065108.1: presentan un 72% de identidad y un coverage del 94,67%. Se trata de una proteina que induce diferenciación neuronal asociada a la proteina 2; presenta un ortólogo en ratón. Se encuentra en el cromosoma 1.
- BB170008A10C08-ENSG00000080802.4: presentan un porcentaje de identidad de 86% y 81,90% de coverage. Es la subunidad 4 de un complejo de transcripción que regula negativamente el proceso; esto se ha encontrado en levaduras. Se halla en el cromosoma 7.
- BB170016B10H03-ENSG00000021574.2: tienen un 80% de identidad y un 85,37% de coverage. Es probablemente un ATPasa involucrada en el ensamblje o función de complejos proteicos nucleares.Se halla en el cromosoma 2.
- BB170016B10H03-ENSG00000132436.1:presentan un porcentaje de identidad del 67% y 80,98% de coverage. Se trata de la proteina FIGNL1 codificada en el cromosoma 7.
- BB0023B10G04-ENSG00000066926.1: tienen un 72% de identidad y 86,71% de coverage. Se trata de una ferrocatalasa que cataliza la inserción de hierro en la porfirina 9. Se halla en el cromosoma 18.
- BB170031A20F07-ENSG00000115760.1: presentan un 87% de identidad y un 99,48%de coverage. Se trata de una proteina que protega a la célula frente la inducción de apoptosis. Se localiza en el cromosoma 2 humano.
REFERENCIAS:
http://www.ncbi.nlm.nih.gov/About/primer/est.htm
http://hgsc.bcm.tmc.edu/projects/honeybe
http://titan.biotec.uiuc.edu/bee/honeybee_project.ht
http://www.genome.org/cgi/content/abstract/12/4/55
http://www.molinicos.org/agricultura/La%20abeja%20IV.htm
http://www.ldc.usb.ve/~vtheok/webmaestro
http://html.conclase.net/tutorial/html
- Hu,P., Wu,S., Sun,Y., Yuan,C.-C., Kobayashi,R., Myers,M.P., Hernandez,N.,
Characterization of human RNA polymerase III identifies orthologues for Saccharomyces cerevisiae RNA polymerase III subunits.
(2002) Mol. Cell. Biol. 22:8044-8055
- Wei,P., Garber,M.E., Fang,S.-M., Fischer,W.H., Jones,K.A.,A novel CDK9-associated C-type cyclin interacts directly with HIV-1 Tat and mediates its high-affinity, loop-specific binding to TAR RNA. (1998) Cell 92:451-462
- Bieniasz,P.D., Grdina,T.A., Bogerd,H.P., Cullen,B.R., Analysis of the effect of natural sequence variation in Tat and in cyclin T on the formation and RNA binding properties of Tat-cyclin T complexes.(1999) J. Virol. 73:5777-5786
-Liu,H. Nakagawa,T. Kanematsu,T. Uchida,T. Tsuji,S. Isolation of 10 differentially expressed cDNAs in differentiated Neuro2a cells induced through controlled expression of the GD3 synthase gene.J. Neurochem. 72:1781-1790 (1999)
CONTACTA CON NOSOTRAS:
Las autoras de este proyecto somos Montse Roura y Marta Valiente, estudiantes de cuarto curso de Biología de la Universidad Pompeu Fabra de Barcelona.
Si tienes cualquier duda o quieres algun tipo de información puedes contactar con nosotras, escribinedo un mail a cualquiera de estas dos direcciones:
Muchas gracias por tu visita!!