Como el título de nuestro trabajo indica, la finalidad de éste consiste en obtener genes ortólogos de ratón o humano a partir de sus correspondientes en la especie de la que partimos; es decir, si introducimos un gen o tránscrito de ratón obtendríamos su ortólogo en humano y viceversa, si introducimos un gen o tránscrito de humano obtendríamos su ortólogo en ratón.
El tema nos resultó interesante ya que como es sabido, un 99% de genes de ratón tienen un homólogo en el genoma de humano y existe una fuerte conservación de la estructura exónica (de genes ortólogos) entre ambas especies. Es decir, en un 86% de los casos, los genes tienen el mismo número de exones y en un 91% los exones ortólogos tienen la misma longitud mientras el 99% conservan la fase, observándose algunos casos de introducción / deleción de intrones.
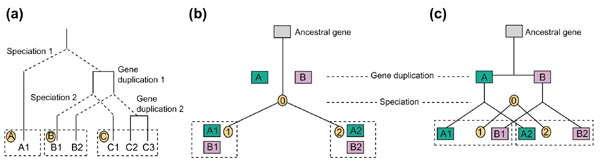
Por todo ello, consideramos conveniente explicar como introducción ciertos términos que suelen llevar a confusión, como son los conceptos de ortología y paralogía. Hay diferentes enfoques de la cuestión (W.M. Fitch, E.V. Koonin, B. Modrek & Ch.J.Lee, J. Gerlt & P. Babbitt). En general se consideran genes ortólogos aquellos homólogos en diferentes especies que catalizan la misma reacción (es decir, con una misma función), mientras que genes parálogos serían aquellos homólogos en una misma especie que no son capaces de catalizar la misma reacción (es decir, con una función distinta). Ortología y paralogía diferirían en que la primera procedería de la especiación mientras que la segunda procedería de la duplicación génica. A continuación, los siguientes esquemas visualizan la diferencia:

(a)Diagrama simplificado de los subtipos homólogos (mostrando tanto ortólogos como parálogos). Eventos de especiación producirían las especies A, B y C. Los genes A1, B1, B2, C1, C2 y C3 descienden de un gen ancestral siguiendo los correspondientes eventos de especiación y duplicación génica. (b,c)Descencencia evolutiva de un gen ancestral dando lugar a parálogos y ortólogos consecuencia de la duplicación en la especie 0, y luego la especiación de las especies 1 y 2. Diagrama (b) muestra el resultado de las relaciones entre parálogos y ortólogos según Koonin. Diagrama (c) es la versión del diagrama b según Roy A. Jensen utilizando el diagrama de Fitch.
Sin embargo, ha de tenerse en cuenta que es posible la adquisición de nuevas propiedades en lo que respecta a los distintos caminos evolutivos, pues se ha comprobado la posibilidad de que existan genes ortólogos con diferentes propiedades catalíticas o reguladoras y que igualmente existan parálogos con una misma función.
Es por ello que algunos bioquímicos han sugerido una clasificación alternativa, según estén en una o diferente especie con una misma o diferente función (sin embargo nosotras no profundizaremos más en este tema).
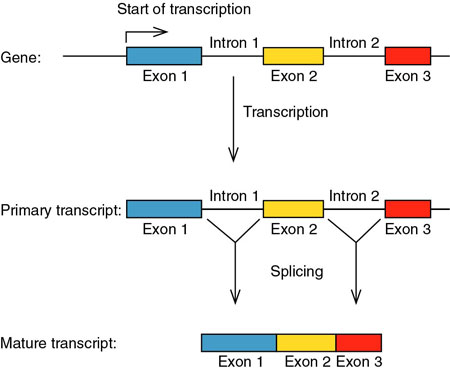
Sin embargo, este proceso no siempre ocurre de una única manera, sino que, por el contrario, existen diferentes combinaciones al procesar el pre-mRNA que darán lugar, a partir de un mismo tránscrito (pre-RNA) y por lo tanto, a partir de un mismo gen, a diferentes RNAs maduros, generando una gran variedad de proteínas. A este tipo de splicing se le denomina splicing alternativo y es importante tenerlo en cuenta debido a que entre un 40-60% de genes humanos son sometidos a este tipo de splicing.
Es por ello que nuestro trabajo ha ido complicándose, pues en la segunda parte de éste hemos intentado obtener, a partir de los tránscritos que proceden de un mismo gen ortólogo de una u otra especie, aquéllos que realmente han sido procesados de una misma manera, teniendo en cuenta la variación que otorga el mecanismo de splicing alternativo. Al parecer, la comparación de pares de exones ortólogos que han sufrido splicing alternativo en humano y ratón muestran una fuerte semejanza. Esto se debe a que el exón que ha sufrido splicing alternativo en ambas especies debe haber existido en su ancestro común, generándose previamente a la divergencia evolutiva posterior de ambas especies. Mientras que la mayor parte de los exones de genomas de humano y ratón están fuertemente conservados en ambas especies, análisis de genes ortólogos en humano y ratón indican que, en aquellas formas generadas por splicing alternativo, también existen exones presentes únicamente en una de ambas especies, producto de recientes creaciones o pérdidas con un efecto positivo, negativo o neutro. Todo ello indica que el splicing alternativo en estos genomas puede ir asociado con el incremento del cambio evolutivo.