La utilització de mètodes estadístics i computacionals en aquest camp permet obtenir una primera aproximació senzilla del problema a l'ordinador.
L'objectiu d'aquest treball és desenvolupar un programa en llenguatge Perl que, donada una seqüència de DNA dins un fitxer de text en format FASTA, construeixi un fitxer en format GFF que contingui les prediccions dels possibles exons de la seqüència.
INTRODUCCIÓ
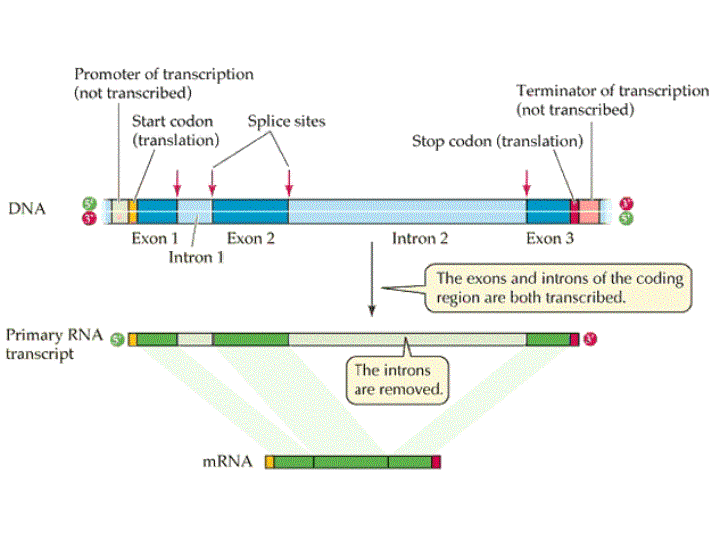
Els components estructurals bàsics d'un gen inclouen: el promotor, l'start codon, introns, exons, i l'stop codon.
La informació genètica del DNA és copiada a RNA mitjançant el procés de transcripció. Els trànscrits de RNA que dirigeixen la síntesi de molècules proteiques són els mRNAs.
L'ordre dels nucleòtids en el DNA indica la seqüència d'aminoàcids d'una proteïna a sintetitzar. Aquest missatge està xifrat en la disposició de les bases A, G, C i T. El mRNA expressat amb les bases A, C, G i U ha de ser traduït a la seqüència d'aminoàcids, determinats pels diferents codons (triplets de bases), que són la unitat d'informació en el mRNA.
La informació completa per a la síntesi d'una cadena polipeptídica es denomina cistró (exons + introns).
Els exons són parts del cistró que contenen informació genètica per a una proteïna, i un intró és una porció de DNA que no posseeix informació per a la síntesi proteica.
Splicing
Es basa en l'eliminació d'introns i posterior unió dels exons en un trànscrit primari, gràcies a RNPnp.
Els introns normalment contenen senyals clares que determinaran el processament de les seqüències genòmiques, ja que la majoria comencen amb la seqüència GT i acaben amb la seqüència AG (de 5' a 3'). Es tracta del donor site i l'acceptor site respectivament. Una altra seqüència important és el branch point localitzat 20-30 bases upstream de l'acceptor site (tot i que no existeix cap seqüència consens).
A més a més, a vegades aquestes senyals poden estar emmascarades per una proteïna reguladora, resultant en splicing alternatiu o en casos més extranys, un pre-mRNA pot contenir diversos senyals ambigus d'splicing que també portarien a la mateixa situació.
Els introns normalment contenen senyals clares que determinaran el processament de les seqüències genòmiques, ja que la majoria comencen amb la seqüència GT i acaben amb la seqüència AG (de 5' a 3'). Es tracta del donor site i l'acceptor site respectivament. Una altra seqüència important és el branch point localitzat 20-30 bases upstream de l'acceptor site (tot i que no existeix cap seqüència consens).
ESQUEMA DEL PROCÉS D'SPLICING