Estudi de la proteïna predita
A partir dels programes utilitzats per a la predicció de gens de la nostra seqüència i de la comparació amb les bases de dades de ESTs, arribem a la conclusió que els gens més probables són el 4, el 5 i el 6 (geneid). Tanmateix estendrem l'estudi a les possibles proteïnes obtingudes amb els altres gens predits. Per això, utilitzarem el blastp (server) de NCBI
Cal dir que els dos programes de predicció de gens utilitzats en l'estudi proteic han estat geneid i genscan, ja que aquests han estat els que ens han oferit els millors resultats.
Els passos que hem seguit per a l'obtenci&oactue; de les prediccions proteiques han estat:
1) En el blastp , enganxem la seqüència d'aminoàcids que prediu el geneid per a cada exó, i seleccionem:
- Choose database: swissprot
- Number of: descriptions:1000 alignments:1000
- Alignment view: pairwise with identities
- Select from: all organisms
- Layout: one window
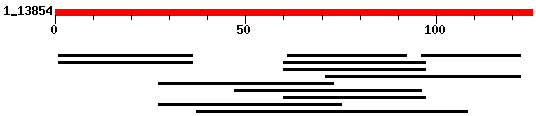
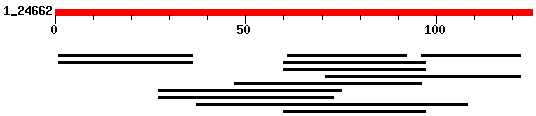
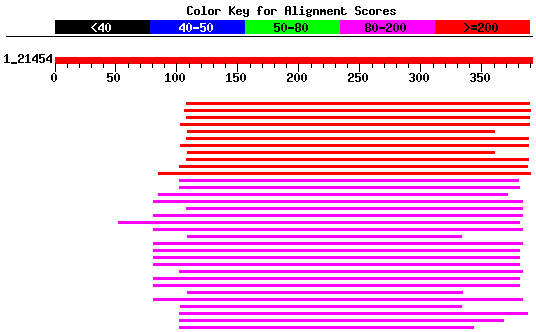
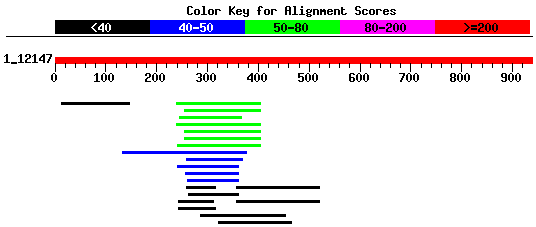
Per tal de facilitar el seguiment de l'estudi proteic mostrem en primer lloc el gràfic on s'observen les prediccions de geneid i genscan juntament amb les validacions d'ESTs (gràfic).El fet d'observar els ESTs situats en forward al gràfic no implica que aquests només estiguin validant els gens que es troben en aquest strand, sinó que simplement és un defecte de la representació. A la base de dades d'ESTs no sabem si estan en forward o reverse.
2 ) Els resultats obtinguts del blastp són:
PRIMERA PROTEÏNA (gen 1)
PROTEÏNA 1 GENEID
PROTEÏNA 1 GENSCAN
Un cop hem fet el blastp amb la predicció de geneid per al primer gen, obtenim 12 hits per aquesta. Ens adonem que l'aliniament no és gaire bo, doncs apareixen e-values molt grans (considerem que els e-values són acceptables si es troben propers a zero, o un nombre elevat a un exponencial negatiu gran). A més, s'observa que els fragments aliniats amb la proteïna són petits, i presenten una gran quantitat de gaps i poques identitats. Les proteïnes que s'alinien pertanyen a organismes de grups taxonòmics molt diferents, com per exemple, pollastre, e.coli, rata...
Decidim doncs provar-ho amb la predicció de genscan i obtenim uns aliniaments exactament iguals, amb els mateixos hits, com es pot comprovar. Semblen aparàixer diferents dominis, per exemple dos P2Y purinoceptor, 3 Sensor protein rcsC,etc.
SEGONA PROTEÏNA (gen 2)
PROTEÏNA 2 GENEID

PROTEÏNA 2 GENSCAN
En aquest cas hem obtingut 1 únic hit. Si observem l'aliniament veiem és bastant curt i s'observen bastants gaps.A més es suficient amb fixar-nos amb l' e-value, el qual es troba en un valor molt alt, fet que reflexa poca similaritat.
Pel que fa a l'aliniament amb la proteïna predita per genscan, observem 4 hits, els quals tampoc s'alinien per complet amb la nostra proteïna, però aporten una mica més d'informació. Per exemple 3 hits coincideixen en l'identificació d'un domini de proteïna ADAM (domini desintegrina i metaloproteinasa).
TERCERA PROTEÏNA (gen 3)
PROTEÏNA 3 GENEID
Un dels pitjors aliniaments , ha estat amb la proteïna 3, on només hem obtingut 3 hits, els quals tenen uns e-values bastant alts. Així doncs, en aquest cas la predicció de la proteïna no ens ajuda a determinar la presència d'un gen. Cal dir, però, que la predicció que es va fer en el seu moment amb els programes geneid i genscan tampoc ens van donar uns resultats gaire esperançadors, per tan era d'esperar obtenir uns resultats com aquests. De fet no hem pogut mostrar l'aliniament obtingut amb genscan, ja que no ens ha trobat similaritats significants, el qual era d'esperar. Un cop més ens corrobora que la possibilitat de trobar un gen en aquella zona és bastant baix.
PROTEÏNA QUATRE (gen 4)
PROTEÏNA 4 GENEID
PROTEÏNA 4 GENSCAN
Si mirem els hits que ha trobat el blastp amb la predicció de geneid, trobem proteïnes amb un e-value d'entre 9e-69 i 2e-34, els quals són uns valors més acceptables. En quant a la taxonomia que presenten les proteïnes, hi ha molta variació. Així, per exemple, en el cas del primer hit, es troba a Caenorhabditis elegans, el segon en llevadura, també es troba en ratolí...
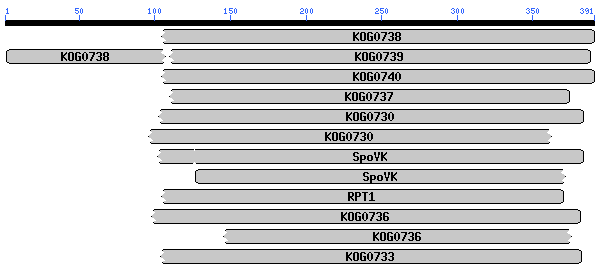
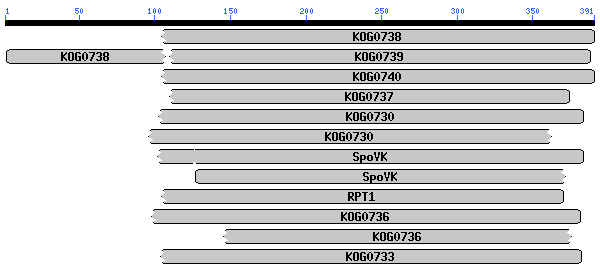
De l'anàlisi dels aliniaments, ens es possible veure alguns residus conservats tan en aquells casos en qué l'aliniament té un score més alt com en aquells en que és inferior. Per exemple, la regió compresa entre els residus 148 i 171 de la proteïna problema, es troba molt ben conservada en tots els aliniaments. Podria ser doncs, que aquesta regió tingués importància funcional.
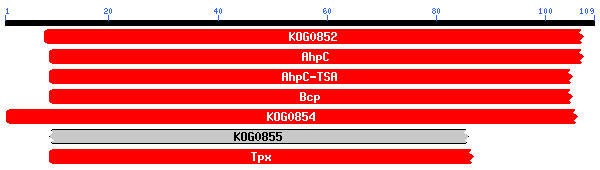
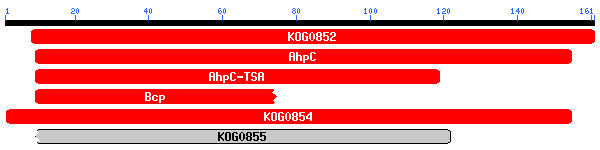
Analitzem aquests dominis conservats i com podem veure a les figures, i en els aliniaments obtinguts amb les proteïnes predites amb genscan i geneid , veiem que el que té un score més alt és el domini KOGO738, el qual té funció ATPasa. Els altres dominis alineats també ho són a excepció d'alguns Factors peroxisoma o proteosoma.
Els dos primers aliniaments són la mateixa proteïna, però s'alinia en dos trossos diferents. Al comprovar l'aliniament amb la nostra query veiem que el primer fragment de query va de 104-391 i la KOGO738 de 207 a 491 , i en el segon fragment la query s'alinia de 1 a 108 al igual que KOGO738. Ens adonem que en la query hi ha un solapament de 4 aminoàcids (104-108) i en la KOGO738 es perd una regió entre (108-207). Això ens pot portar a pensar que en la predicció hem perdut un exó, ja que la resta de l'aliniament és prou bo o que es tracta d'un pseudogen.
Però si investiguem una mica més podem arribar a saber a quin exó ha esdevingut el truncament (es a dir la localització de l'aa 104 en la nostra seqüència). Això ho sabrem realitzant una sèrie de càlculs.
El que hem obtingut és que els nucleòtids finals de l'exó 2 codifiquen per l'aa 104. El fet que es doni a la part final del gen fa pensar que la nostra predicció a passat per alt un exó, o que aquest s'ha perdut al llarg de l'evolució.
Una manera de comprovar aquestes hipòtesis ha estat mirant els ESTs i observar si algun d'ells ens permet predir un gen entre l'exó 2 i 3. I tal com podem observar al gràfic dels ESTs i les prediccions, veiem que entre els exons esmentats els ESTs validen la presència d'un altre exó que els programes genscan, geneid no prediuen. Per tant la nostra primera hipotesi és correcta.
Per altra banda hem utilitzat el programa GeneWise on hem enfrontat la nostra proteïna amb la subseqüència hg16_dna_261662-338899 (inclou els els gens esmentats). Aquest programa ens alinia la nostra proteïna tenint en compte l'estructura d'introns-exons. El resultat del genewise ens mostra 4 exons, pero a la regió de l'aa 110-120 torna a apareixer un intró.
També hem realitzat un tblastn tblastn .
Per altra banda hem entrat al link de KOGO738, on trobem aliniaments de la proteïna amb altres organismes. Comprovem que aquest domini es troba en Eucariotes i que és semblant en Homo Sapiens a la subunitat p60 de la katanina (proteïna de citoesquelet). En Arabidopsis Thaliana també s'assembla a katanina i ATPasa. Això no és d'estranyar ja que la katanina té activitat ATPasa. Un altre organisme on es troba un aliniament es en Drosophila.Així verifiquem que aquest domini es troba conservat a nivell Eucariota.
PROTEÏNA CINC (gen 5):
PROTEÏNA 5 GENEID
PROTEÏNA 5 GENSCAN
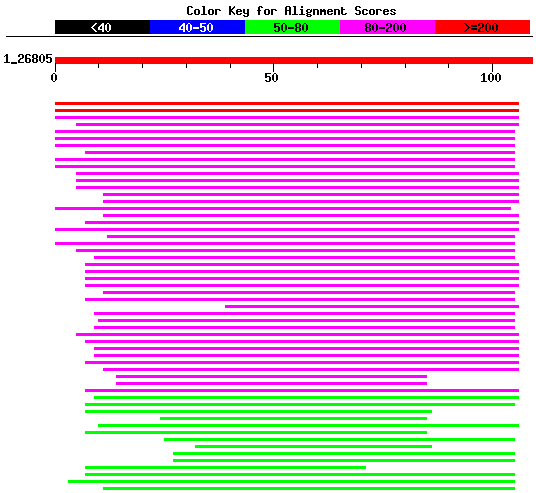
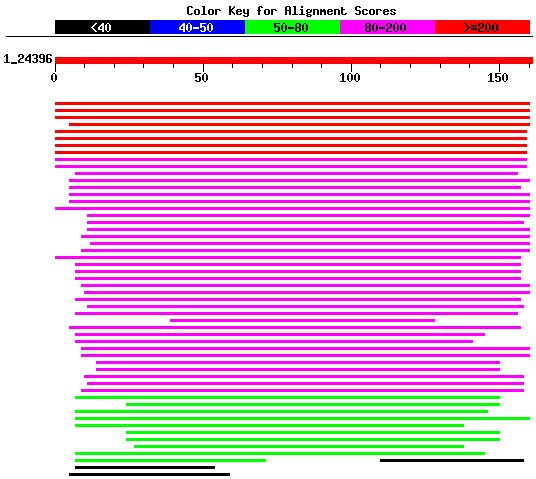
En aquest cas obtenim un conjunt d'aliniaments amb uns e-values prou significatius. A més la majoria d'ells es troben solapats, i els scores del primers són bastant alts (sobre 200). El primer hit és amb una Peroxiredoxin (98% de conservació) al igual que la majoria dels altres hits. Això fa pensar que probablement aquesta proteïna tindrà algun domini comú amb la Peroxiredoxina. A més en la imatge anterior podem veure que els dominis fan referència a aquesta funció. El que està clar és que la nostra proteïna predita només seria una part de la proteïna amb funció peroxiredoxina humana, ja que aquesta última la formen més de 200 aa.
Els resultats obtinguts amb el genscan com es pot observar són exactament iguals.
Cal dir que el genscan ens predeia un sisè exó peró no estava validat per cap EST.
PROTEÏNA SIS
PROTEÏNA 6 GENEID
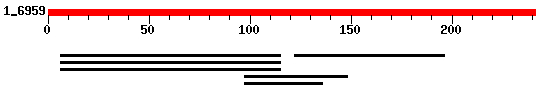
PROTEÏNA 8 GENSCAN
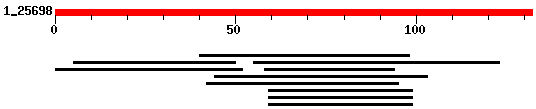
PROTEÏNA 9 GENSCAN
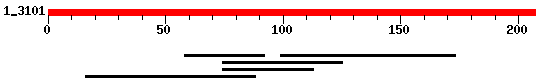
La predicció de genscan en aquesta subseqüència ens donava 3 gens (7, 8 i 9). I en la predicció de geneid, el 8 i 9 de genscna, els unia en un únic gen (6).
El blastp del gen 6 predit en geneid, ens dóna 6 hits el millor amb un e-value de 0.1 i correspon al receptor del factor de creixement d'insulina humana, i el 3r i 4t de rata i ratolí respectivament. Aquests es troben aliniats. També s'observen alguns hits de enzim alpha-glucan branching.
Per tal de comparar fem el blastp amb els gens 8 i 9 per separat i observem els hits. En el primer tan sols tenim 10 hits,i el millor presenta un e-value de 0.21 i es tracta del precursor de la Glicoproteïna B. Els altre hits estan poc conservats i les puntuacions són baixes. I el més important és que no coincideix cap amb els hits obtinguts amb la proteïna predita per geneid .
En l'aliniament amb el gen 9 en canvi, tot i nomes tenir 5 hits 4 d'ells coincideixen amb la proteïna de geneid. El millor hit es la HI0523 amb un e-value de 0.44, i en l'altre era de 0.6. Així doncs aquestes dues prediccions podem dir que estan validant la mateixa proteïna.
Amb la finalitat de millorar l'estudi de la regió del gen 8 (genscan) farem el següent:
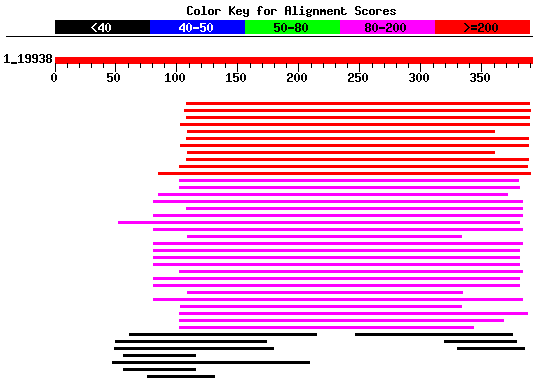
La regió que correspon al gen 9 predit per genscan i part del 6 predit per geneid, no estan validats per ESTs, en canvi la regio del gen 8 predita per genscan si (gràfic) . Hem decidit fer una ampliació d'aquesta zona utilitzant les següents comandes:
-gawk '$4>8000 && $5<90000'hg16_dna_369503-500000.spliced_rescored_modificat.gff | gff2ps -f -S 20000 - > test.ps
-convert -rotate 90 test.ps test.jpg
-kview test.jpg
- Choose database: swissprot
- Number of: descriptions:1000 alignments:1000
- Alignment view: pairwise with identities
- Select from: all organisms
- Layout: one window
Per tal de facilitar el seguiment de l'estudi proteic mostrem en primer lloc el gràfic on s'observen les prediccions de geneid i genscan juntament amb les validacions d'ESTs (gràfic).El fet d'observar els ESTs situats en forward al gràfic no implica que aquests només estiguin validant els gens que es troben en aquest strand, sinó que simplement és un defecte de la representació. A la base de dades d'ESTs no sabem si estan en forward o reverse.
2 ) Els resultats obtinguts del blastp són:
PRIMERA PROTEÏNA (gen 1)
Un cop hem fet el blastp amb la predicció de geneid per al primer gen, obtenim 12 hits per aquesta. Ens adonem que l'aliniament no és gaire bo, doncs apareixen e-values molt grans (considerem que els e-values són acceptables si es troben propers a zero, o un nombre elevat a un exponencial negatiu gran). A més, s'observa que els fragments aliniats amb la proteïna són petits, i presenten una gran quantitat de gaps i poques identitats. Les proteïnes que s'alinien pertanyen a organismes de grups taxonòmics molt diferents, com per exemple, pollastre, e.coli, rata... Decidim doncs provar-ho amb la predicció de genscan i obtenim uns aliniaments exactament iguals, amb els mateixos hits, com es pot comprovar. Semblen aparàixer diferents dominis, per exemple dos P2Y purinoceptor, 3 Sensor protein rcsC,etc. |
SEGONA PROTEÏNA (gen 2)
|
En aquest cas hem obtingut 1 únic hit. Si observem l'aliniament veiem és bastant curt i s'observen bastants gaps.A més es suficient amb fixar-nos amb l' e-value, el qual es troba en un valor molt alt, fet que reflexa poca similaritat.
Pel que fa a l'aliniament amb la proteïna predita per genscan, observem 4 hits, els quals tampoc s'alinien per complet amb la nostra proteïna, però aporten una mica més d'informació. Per exemple 3 hits coincideixen en l'identificació d'un domini de proteïna ADAM (domini desintegrina i metaloproteinasa). |
TERCERA PROTEÏNA (gen 3)
| PROTEÏNA 3 GENEID |
 |
Un dels pitjors aliniaments , ha estat amb la proteïna 3, on només hem obtingut 3 hits, els quals tenen uns e-values bastant alts. Així doncs, en aquest cas la predicció de la proteïna no ens ajuda a determinar la presència d'un gen. Cal dir, però, que la predicció que es va fer en el seu moment amb els programes geneid i genscan tampoc ens van donar uns resultats gaire esperançadors, per tan era d'esperar obtenir uns resultats com aquests. De fet no hem pogut mostrar l'aliniament obtingut amb genscan, ja que no ens ha trobat similaritats significants, el qual era d'esperar. Un cop més ens corrobora que la possibilitat de trobar un gen en aquella zona és bastant baix.
PROTEÏNA QUATRE (gen 4)
PROTEÏNA 4 GENEID
PROTEÏNA 4 GENSCAN




| PROTEÏNA 4 GENEID | PROTEÏNA 4 GENSCAN |
 |
 |
 |
 |
Si mirem els hits que ha trobat el blastp amb la predicció de geneid, trobem proteïnes amb un e-value d'entre 9e-69 i 2e-34, els quals són uns valors més acceptables. En quant a la taxonomia que presenten les proteïnes, hi ha molta variació. Així, per exemple, en el cas del primer hit, es troba a Caenorhabditis elegans, el segon en llevadura, també es troba en ratolí...
De l'anàlisi dels aliniaments, ens es possible veure alguns residus conservats tan en aquells casos en qué l'aliniament té un score més alt com en aquells en que és inferior. Per exemple, la regió compresa entre els residus 148 i 171 de la proteïna problema, es troba molt ben conservada en tots els aliniaments. Podria ser doncs, que aquesta regió tingués importància funcional.
Analitzem aquests dominis conservats i com podem veure a les figures, i en els aliniaments obtinguts amb les proteïnes predites amb genscan i geneid , veiem que el que té un score més alt és el domini KOGO738, el qual té funció ATPasa. Els altres dominis alineats també ho són a excepció d'alguns Factors peroxisoma o proteosoma.
Els dos primers aliniaments són la mateixa proteïna, però s'alinia en dos trossos diferents. Al comprovar l'aliniament amb la nostra query veiem que el primer fragment de query va de 104-391 i la KOGO738 de 207 a 491 , i en el segon fragment la query s'alinia de 1 a 108 al igual que KOGO738. Ens adonem que en la query hi ha un solapament de 4 aminoàcids (104-108) i en la KOGO738 es perd una regió entre (108-207). Això ens pot portar a pensar que en la predicció hem perdut un exó, ja que la resta de l'aliniament és prou bo o que es tracta d'un pseudogen.
Però si investiguem una mica més podem arribar a saber a quin exó ha esdevingut el truncament (es a dir la localització de l'aa 104 en la nostra seqüència). Això ho sabrem realitzant una sèrie de càlculs.
El que hem obtingut és que els nucleòtids finals de l'exó 2 codifiquen per l'aa 104. El fet que es doni a la part final del gen fa pensar que la nostra predicció a passat per alt un exó, o que aquest s'ha perdut al llarg de l'evolució.
Una manera de comprovar aquestes hipòtesis ha estat mirant els ESTs i observar si algun d'ells ens permet predir un gen entre l'exó 2 i 3. I tal com podem observar al gràfic dels ESTs i les prediccions, veiem que entre els exons esmentats els ESTs validen la presència d'un altre exó que els programes genscan, geneid no prediuen. Per tant la nostra primera hipotesi és correcta.
Per altra banda hem utilitzat el programa GeneWise on hem enfrontat la nostra proteïna amb la subseqüència hg16_dna_261662-338899 (inclou els els gens esmentats). Aquest programa ens alinia la nostra proteïna tenint en compte l'estructura d'introns-exons. El resultat del genewise ens mostra 4 exons, pero a la regió de l'aa 110-120 torna a apareixer un intró.
També hem realitzat un tblastn tblastn .
Per altra banda hem entrat al link de KOGO738, on trobem aliniaments de la proteïna amb altres organismes. Comprovem que aquest domini es troba en Eucariotes i que és semblant en Homo Sapiens a la subunitat p60 de la katanina (proteïna de citoesquelet). En Arabidopsis Thaliana també s'assembla a katanina i ATPasa. Això no és d'estranyar ja que la katanina té activitat ATPasa. Un altre organisme on es troba un aliniament es en Drosophila.Així verifiquem que aquest domini es troba conservat a nivell Eucariota.
PROTEÏNA CINC (gen 5):
| PROTEÏNA 5 GENEID | PROTEÏNA 5 GENSCAN |
 |
 |
 |
 |

La regió que correspon al gen 9 predit per genscan i part del 6 predit per geneid, no estan validats per ESTs, en canvi la regio del gen 8 predita per genscan si (gràfic) . Hem decidit fer una ampliació d'aquesta zona utilitzant les següents comandes:
-gawk '$4>8000 && $5<90000'hg16_dna_369503-500000.spliced_rescored_modificat.gff | gff2ps -f -S 20000 - > test.ps
-convert -rotate 90 test.ps test.jpg
-kview test.jpg
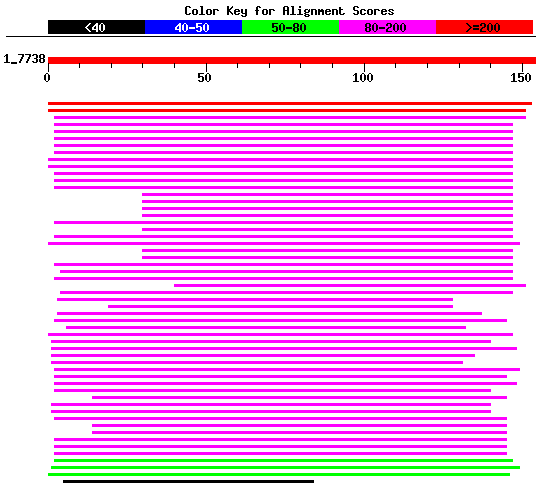
La imatge obtinguda es mostra a continuació
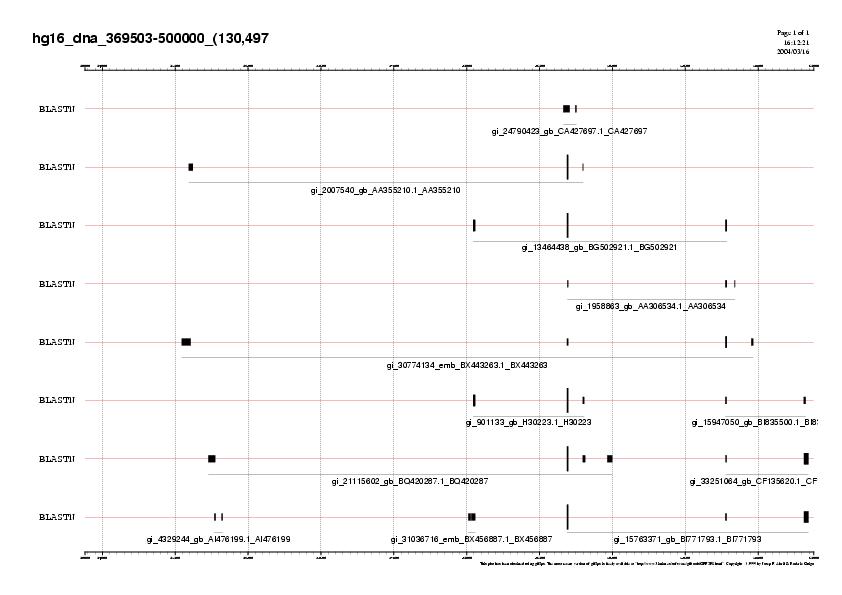
Ara podem observar amb major claredat els ESTs,i amb la finalitat de dur a terme un estudi més exhaustiu, busquem el DNA de l' EST amb un score major gi_30774134_emb_BX443263.1_BX443263 . I duem a terme un BLASTX tenint en compte tots els organismes. El resultat del BLASTX és:
Els hits més alts corresponen a subfamilies Alu, les quals no apareixien en el blastp del gen 8. Això aporta nova informació, el qual obre una nova via d'estudi.
El gen 7 predit amb el genscan no està suportat per cap EST.
PROTEÏNA SET
PROTEÏNA 7 GENEID
| PROTEÏNA 7 GENEID |
 |
La proteïna 7 predita per geneid, tal i com s'observa en l'aliniament , ens aporta bastanta informació.El primer hit obtingut, amb un score de 287 i un 97% d'identitat, ens permet dir que aquesta &eactue;s Ubiquitin-conjugating enzyme E2-18 kDa UbcH7 (Ubiquitin-protein ligase) (Ubiquitin-protein). I tots els altres hits són de proteïnes de la mateixa família com era d'esperar.En aquest cas podem afirmar amb seguretat que aquesta proteïna es troba en la nostra seqüència, i podem obtenir més informació en el link de NCBI (gi|1717860|sp|P51966|UBC7_HUMAN).