Obtenció de la seqüència de DNA
El nostre treball es basa en l'anàlisi d' una de les seqüències que formen part del projecte ENCODE, concretament la NT_024524.13. El primer que hem fet és obtenir la seqüència en la base de dades ncbi (NT_024524.13). Tal i com es pot veure els esquemes que segueixen, està localitzada al cromosoma 13 d'Homo sapiens, concretament entre les posicions 11500016-12000015.

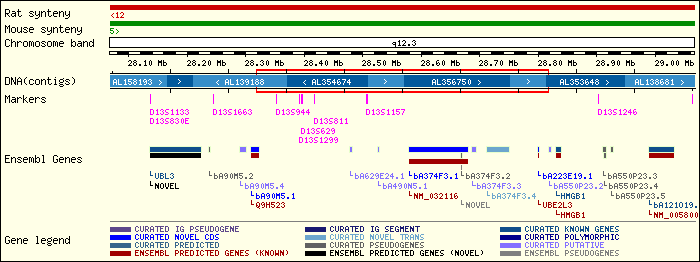
Aquesta seqüència està en format fasta, que és el que es necessita per a l'anàlisi que realitzarem. Tanmateix, el format fasta té un símbol ">" a l'inici de la seqüència. Introduïrem aquest símbol usant l'emacs NT_024524.fa .
Una altra manera d'enregistar la seqüència que també serà útil és en format tabular. Per exemple, es pot usar el següent transcrit per obtenir el format tabular:
- awk ' { printf "%s", $0 } '
Li direm seq1.tbl.
A continuació, usarem l'emacs per introduir un tabulador ("t") entre l'identificador i la seqüència propiament dita. Aquest fitxer obtingut serà utilitzat per:
- Calcular la longitud de la seqüència:
- awk '{print lenght($2)} seq1.tbl
- aquesta ordre ens retorna el valor de 500000, que és el nombre de nucleòtids de la nostra seqüència
- Calcular el contingut G+C:
- awk '{print$2}' seq1.tbl | fold -1 | sort | uniq -c | gawk '{print $2, 41/500000}'
- I el resultat obtingut és:
- A : 0,294306
- C : 0,21887
- G : 0,212097
- T : 0,27473
- i el contingut G+C : 43,10 %



- awk ' { printf "%s", $0 } '
- awk '{print lenght($2)} seq1.tbl
- aquesta ordre ens retorna el valor de 500000, que és el nombre de nucleòtids de la nostra seqüència
- awk '{print$2}' seq1.tbl | fold -1 | sort | uniq -c | gawk '{print $2, 41/500000}'
- I el resultat obtingut és:
- A : 0,294306
- C : 0,21887
- G : 0,212097
- T : 0,27473
- i el contingut G+C : 43,10 %
- A : 0,294306