Validació dels gens predits a la bdd de ESTs
Un cop hem predit els possibles gens de la nostra seqüència mitjançant els
programes anomenats a l'apartat anterior, ara és moment d'intentar
validar aquestes prediccions. I buscar nous mètodes per validar-les. Un
d'aquests possibles mètodes podria ser enfrontar la nostra seqüència a
una base de dades d'ESTs humans mitjançant el programa Megablast.
Els ESTs (Expressed Sequence Tags) són petits fragments de DNA (entre 200 i
500 nucleòtids) generats per la seqüenciació d' un o ambdós extrems
d'un gen (5' i 3'). Aquests poden ser utilitzats per identificar gens
desconeguts i trobar la seva posició dins el genoma.
El programa Megablast l'hem utilitzat amb els paràmetres següents:
- Base de dades : est-human
- Select from: Homo Sapiens [ORNG]
- Word size: 16
- Number of descriptions: 1000
- Number of aligments: 1000
- Aligment view: Pairwise with identities
- Select from: Homo sapiens
- Layout: one window
- Formatting options: At the bottom
Un cop s'ha dut a terme el blast, obtenim un output (seq1fm1.blast.est), i a partir d'aquest el transformem a format gff mitjançant el programa parseblast:
- parseblast -G seq1fm1.blast.est > seq1fm1.blast.est.gff
Posteriorment obtenim el format ps amb el gff2ps, i el visualitzem amb kview:
- gff2ps seq1fm1.blast.est.gff > repeat.seq1fm1.blast.est.ps
- convert -rotate 90 seq1fm1.blast.est.ps seq1fm1.blast.est.jpg
- kview seq1fm1.blast.est.jpg
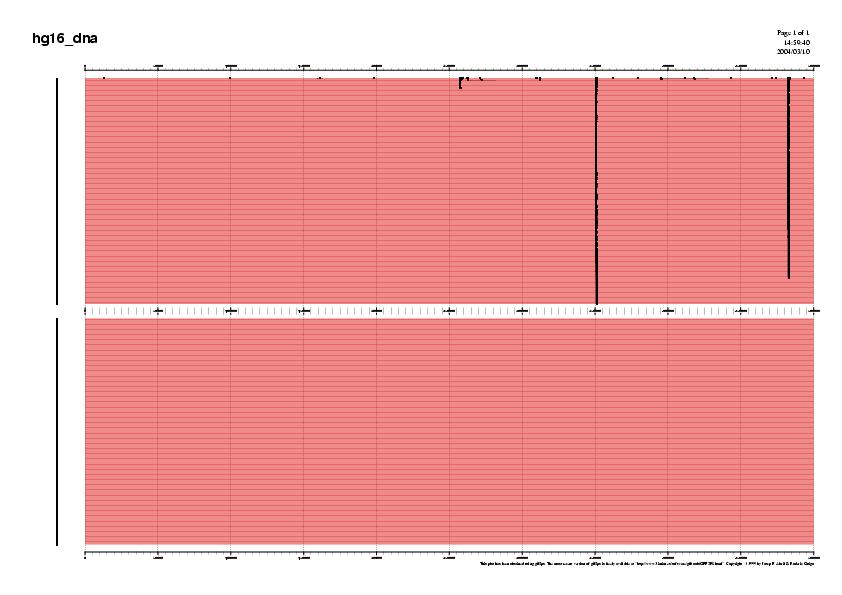
La imatge mostra una gran quantitat d'ESTs aliniats a una determinada posició, i a causa de la gran quantitat d'aquests elements (1000) i a
la llargada de la seqüència (500Kb) no es pot apreciar la informació
que ens proporcionen. Però de fet, els ESTs considerats una bona
evidència per l'existència d'un gen són els spliced, i per tal
d'obtenir-los hem d'identificar aquells hsp's que apareixen més d'un
cop en el format gff. Únicament necessitarem un petit programa de Perl
que es troba dins getsplicedhsp.awk (awk script)
- awk -f getsplicedhsp.awk seq1fm1.blast.est.gff > seq1fm1.blast.est.spliced.gff
I posteriorment fem una representació dels ESTs spliced juntament amb
les prediccions de gens que havíem obtingut amb els programes geneid i
genscan. D'aquesta manera podem comprovar si existeixen ESTs que
validen tals prediccions. Les comandes són:
- gff2ps seq1fm1.blast.est.spliced.gff seq1m.geneid.gff seq1m.genscan.gff > seq1fm1.est.3.genepredictions.ps
- convert -rotate 90 seq1fm1.est.3.genepredictions.ps seq1fm1.est.3.genepredictions.jpg
- kview seq1fm1.est.3.genepredictions.jpg
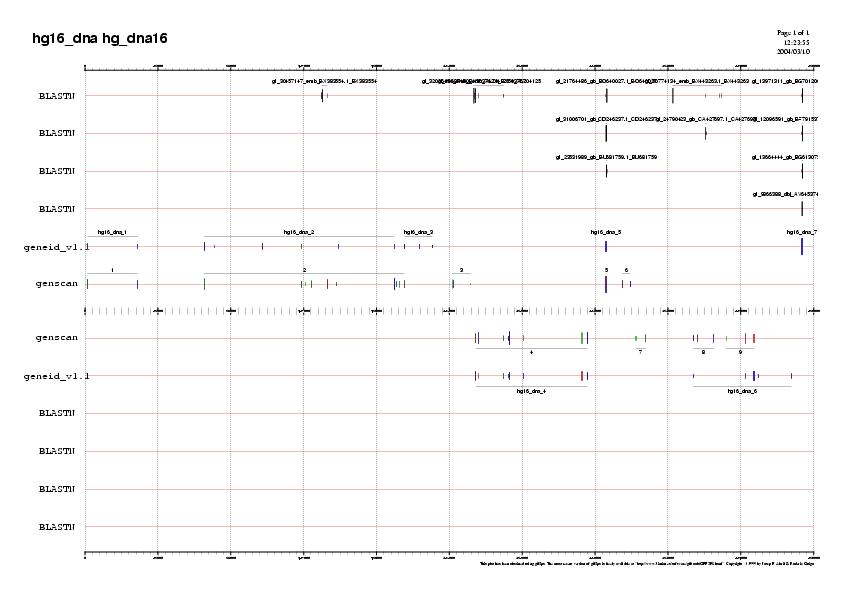
Desprès d'haver realitzat el spliced, s'observa una millora notable, ja que el nombre d'ESTs ès molt menor, i ens permeten validar alguns dels gens predits. Excepte el gen 1 predit per genscan i geneid, tots els altres presenten algun exó validat per ESTs. Com a exemple el cas del gen 5 predit per ambdós programes, que es troba clarament soportat, com a mínim per 3 ESTs diferents.
Un aspecte que ens pot cridar l'atenció del gràfic és que tots els ESTs es troben en forward. En realitat, cal recordar que les bases de dades d'ESTs no ens informen de el strand d'aquests, i a l'hora de realitzar el dibuix surt per defecte.

- Base de dades : est-human
- Select from: Homo Sapiens [ORNG]
- Word size: 16
- Number of descriptions: 1000
- Number of aligments: 1000
- Aligment view: Pairwise with identities
- Select from: Homo sapiens
- Layout: one window
- Formatting options: At the bottom
Un cop s'ha dut a terme el blast, obtenim un output (seq1fm1.blast.est), i a partir d'aquest el transformem a format gff mitjançant el programa parseblast:
- parseblast -G seq1fm1.blast.est > seq1fm1.blast.est.gff
Posteriorment obtenim el format ps amb el gff2ps, i el visualitzem amb kview:
- gff2ps seq1fm1.blast.est.gff > repeat.seq1fm1.blast.est.ps
- convert -rotate 90 seq1fm1.blast.est.ps seq1fm1.blast.est.jpg
- kview seq1fm1.blast.est.jpg

La imatge mostra una gran quantitat d'ESTs aliniats a una determinada posició, i a causa de la gran quantitat d'aquests elements (1000) i a
la llargada de la seqüència (500Kb) no es pot apreciar la informació
que ens proporcionen. Però de fet, els ESTs considerats una bona
evidència per l'existència d'un gen són els spliced, i per tal
d'obtenir-los hem d'identificar aquells hsp's que apareixen més d'un
cop en el format gff. Únicament necessitarem un petit programa de Perl
que es troba dins getsplicedhsp.awk (awk script)
- awk -f getsplicedhsp.awk seq1fm1.blast.est.gff > seq1fm1.blast.est.spliced.gff
I posteriorment fem una representació dels ESTs spliced juntament amb
les prediccions de gens que havíem obtingut amb els programes geneid i
genscan. D'aquesta manera podem comprovar si existeixen ESTs que
validen tals prediccions. Les comandes són:
- gff2ps seq1fm1.blast.est.spliced.gff seq1m.geneid.gff seq1m.genscan.gff > seq1fm1.est.3.genepredictions.ps
- convert -rotate 90 seq1fm1.est.3.genepredictions.ps seq1fm1.est.3.genepredictions.jpg
- kview seq1fm1.est.3.genepredictions.jpg

Desprès d'haver realitzat el spliced, s'observa una millora notable, ja que el nombre d'ESTs ès molt menor, i ens permeten validar alguns dels gens predits. Excepte el gen 1 predit per genscan i geneid, tots els altres presenten algun exó validat per ESTs. Com a exemple el cas del gen 5 predit per ambdós programes, que es troba clarament soportat, com a mínim per 3 ESTs diferents.
Un aspecte que ens pot cridar l'atenció del gràfic és que tots els ESTs es troben en forward. En realitat, cal recordar que les bases de dades d'ESTs no ens informen de el strand d'aquests, i a l'hora de realitzar el dibuix surt per defecte.

Posteriorment obtenim el format ps amb el gff2ps, i el visualitzem amb kview:
- gff2ps seq1fm1.blast.est.gff > repeat.seq1fm1.blast.est.ps
- convert -rotate 90 seq1fm1.blast.est.ps seq1fm1.blast.est.jpg
- kview seq1fm1.blast.est.jpg
- convert -rotate 90 seq1fm1.blast.est.ps seq1fm1.blast.est.jpg
- kview seq1fm1.blast.est.jpg

La imatge mostra una gran quantitat d'ESTs aliniats a una determinada posició, i a causa de la gran quantitat d'aquests elements (1000) i a
la llargada de la seqüència (500Kb) no es pot apreciar la informació
que ens proporcionen. Però de fet, els ESTs considerats una bona
evidència per l'existència d'un gen són els spliced, i per tal
d'obtenir-los hem d'identificar aquells hsp's que apareixen més d'un
cop en el format gff. Únicament necessitarem un petit programa de Perl
que es troba dins getsplicedhsp.awk (awk script)
- awk -f getsplicedhsp.awk seq1fm1.blast.est.gff > seq1fm1.blast.est.spliced.gff
I posteriorment fem una representació dels ESTs spliced juntament amb
les prediccions de gens que havíem obtingut amb els programes geneid i
genscan. D'aquesta manera podem comprovar si existeixen ESTs que
validen tals prediccions. Les comandes són:
- gff2ps seq1fm1.blast.est.spliced.gff seq1m.geneid.gff seq1m.genscan.gff > seq1fm1.est.3.genepredictions.ps
- convert -rotate 90 seq1fm1.est.3.genepredictions.ps seq1fm1.est.3.genepredictions.jpg
- kview seq1fm1.est.3.genepredictions.jpg

I posteriorment fem una representació dels ESTs spliced juntament amb
les prediccions de gens que havíem obtingut amb els programes geneid i
genscan. D'aquesta manera podem comprovar si existeixen ESTs que
validen tals prediccions. Les comandes són:
- gff2ps seq1fm1.blast.est.spliced.gff seq1m.geneid.gff seq1m.genscan.gff > seq1fm1.est.3.genepredictions.ps
- convert -rotate 90 seq1fm1.est.3.genepredictions.ps seq1fm1.est.3.genepredictions.jpg
- kview seq1fm1.est.3.genepredictions.jpg
