Señales de splicing
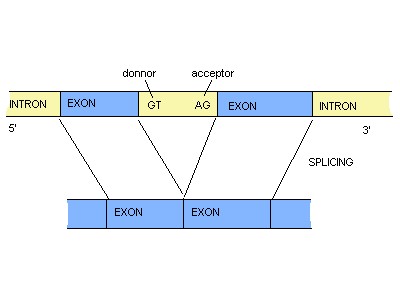
Iniciales: Delimitados por un codón inicial (ATG) y un donnor site.
Internos: Delimitados por un acceptor site y un donnor.
Terminales: Delimitados por un donnor site y un codón STOP (TGA, TAG y TAA).
Singles: Delimitados por un codón inicial y un STOP.
Sesgo codificante
El uso de codones en los exones que se traducen a proteína no es aleatorio, sino que existe un sesgo en el uso de codones concretos en función del organismo. Este hecho puede ser utilizado para puntuar los exones según los codones que utiliza y ver si un exón tiene más posibilidades de ser un exón real en este organismo. Si un exón utiliza más codones de nuestra tabla le daremos una puntuación mayor.