Predicció de gens
Per tal de realitzar la predicció de gens "ab initio" hem introduit la seqüència emmascarada en format fasta en els programes geneid i genscan. Les prediccions obtingudes s'han guardat en els següents fitxers:
Per tal de visualitzar les prediccions d'una forma gràfica i poder comparar millor els resultats d'ambdós programes s'ha utilitzat de nou el programa gff2ps. Donat que l'output del geneid ja està en format gff primer s'ha convertit l'output obtingut amb el genscan a format gff. Les comandes utilitzades són les següents:
-
- gawk 'BEGIN{OFS="\t"}$2 ~ /Term|Intr|Init/ {print "AC091491", "genscan", $2, start=($4<$5 ? $4 : $5), end=($5<$4 ? $4 : $5),$13,$3,$7, $1}' AC091491.genscan | sed 's/\.[0-9][0-9]$//' > AC091491.genscan.gff
- ./gff2ps_v0.98c AC091491.geneid.gff AC091491.genscan.gff >AC091491.genepredictions.ps
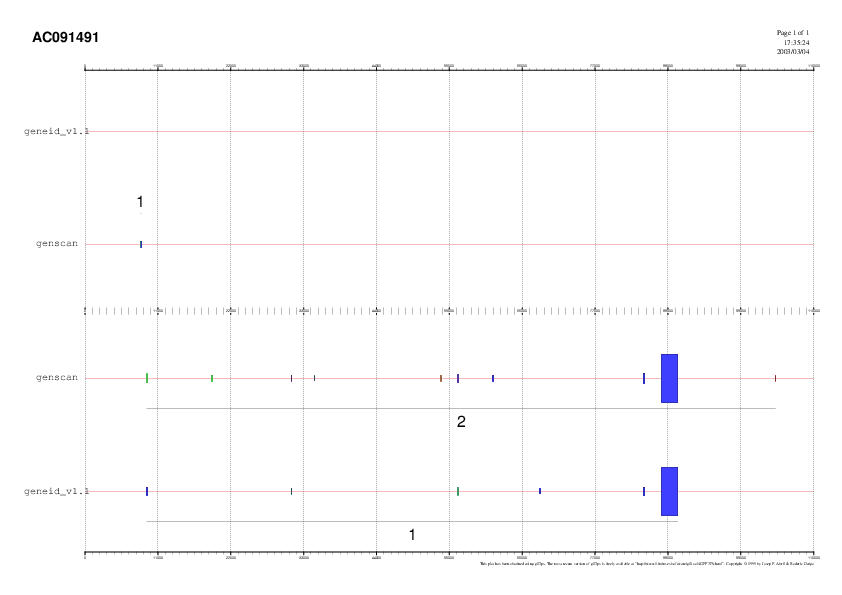
Fig 3: Resultats de la predicció de gens per genscan i geneid
Predicció del Genscan: segons aquest programa la nostra seqüència contindria un gen complet en revers constituit per 10 exons entre els quals trobem un first i un terminal. Alguns d'aquests exons tenen un score molt baix i inclús negatiu com ara el sisè internal. També ens prediu un terminal en forward que formaria part d'un altre gen.
Predicció del Geneid: en aquest cas es prediu un gen en reverse que coincidiria en alguns dels exons amb la predicció del gen en reverse del genscan. Aquest gen contindria 5 exons internals i un terminal amb bons scores excepte en el tercer internal. A diferència de la predicció anterior no s'ha obtingut un first i per tant el gen estaria incomplet. Tampoc veiem cap predicció en forward.
Validació de les prediccions
Per tal de reforçar els resultats de les prediccions obtingudes amb els anteriors programes hem obtingut els ESTs presents en la seqüència mitjançant el programa Megablast. Aquest ens compara la seqüència amb una base de ESTs humans i ens dóna un fitxer amb els ESTs homòlegs (AC091491.blast.est) a diferents regions de la seqüència. Amb aquest resultat podem configurar un gràfic que ens representa la localització dels ESTs trobats en la nostra seqüència, processant el fitxer amb el programa parseblast, per tal d'obtenir el fitxer amb format gff
Comandes:
-
- parseblast -Gi AC091491.blast.est | gawk 'BEGIN{OFS="\t"}{$2=$9;$6=$7=".";print}' > AC091491.blast.est.gff
- ./gff2ps_v0.98c AC091491.blast.est.gff > AC091491.blast.est.ps
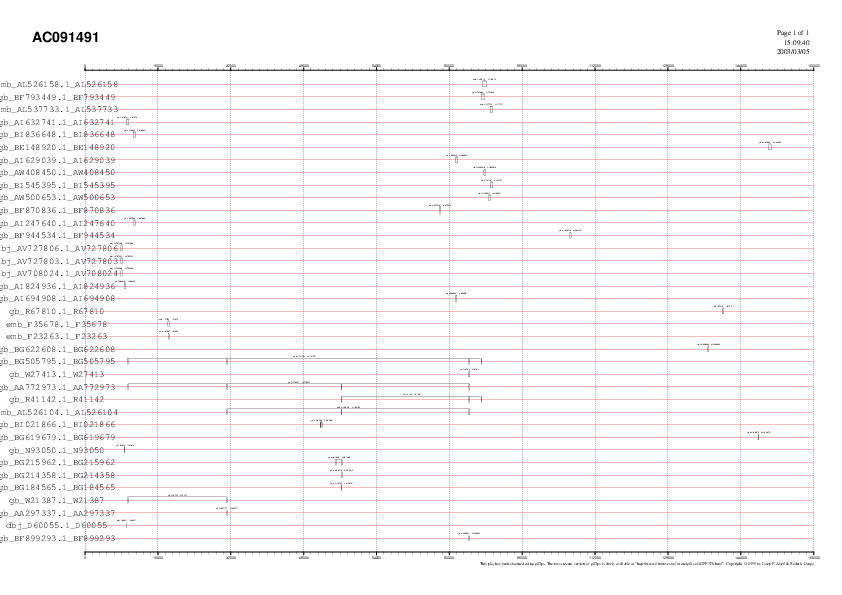
Fig 4: ESTs obtinguts per Megablast
Per tal de visualitzar els ESTs que es consideren una bona evidència a l'hora de validar l'existència de gens processarem el fitxer de tal manera que es quedi amb els hps que apereguin més d'una vegada en el fitxer obtingut amb format gff, ja que seran els que hauran passat per un procÚs d'splicing. Per tal de dur a terme aquest procès utilitzem un programa en awk (awk script), juntament amb les següents comandes:
-
- gawk -f getsplicedhsp.awk AC091491.blast.est.gff > AC091491.blast.est.spliced.gff
- ./gff2ps_v0.98c AC091491.blast.est.spliced.gff AC091491.geneid.gff AC091491.genscan.gff >AC091491.est+genepredictions.ps
Finalment podem visualitzar el resultat mitjançant el ghostview:
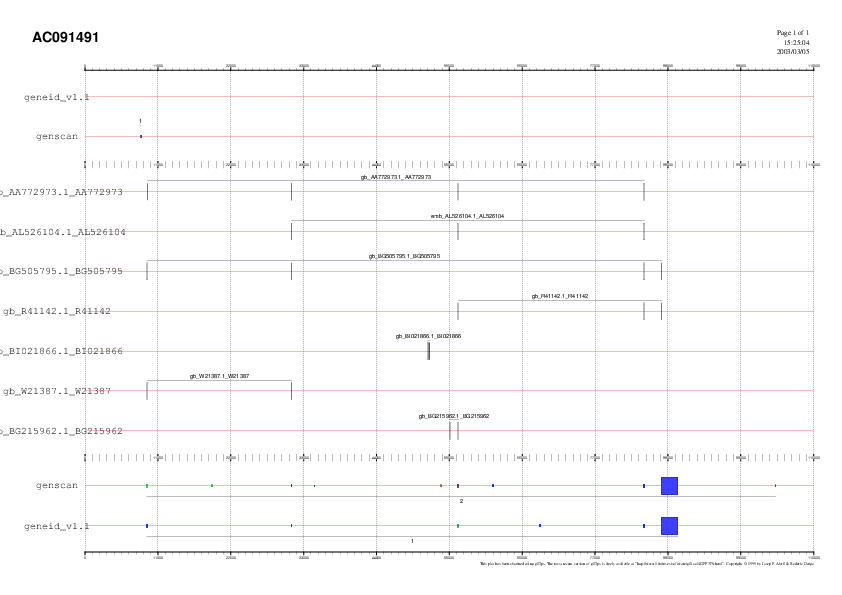
Fig 5: Prediccions dels gens + spliced ESTs
Com es pot observar, cinc dels ESTs concorden amb les posicions de 5 exons predits amb els dos programes. Observem que la predicció del geneid per aquesta seqüència està més suportada per la presència de ESTs en tots els exons que prediu excepte en un d'ells, el tercer, el qual presentava un score molt baix. D'altra banda no s'observa cap EST que reforci el primer exó del gen predit pel programa genscan. Això juntament amb el seu score baix, ens indica que podria tractar-se d' un fals positiu. A més a més veiem que es pot donar splicing alternatiu ja que el tercer EST té una forma d'splicing diferent a la de la resta d'ESTs.