Extensió de la seqüència
Donat que la predicció del geneid, més fiable que la del genscan tal com s'observa amb la validació per ESTs, no contempla l'exó first, s'ha procedit a extendre la seqüència 90000 pb upstream mitjançant el servidor UCSC, que ens proporciona la seqüència en format fasta ( expAC091491.fa ). Un cop obtinguda la seqüència extesa s'ha seguit el mateix protocol que per a la seqüència inicial: emmascarament i predicció de gens.
Identificació de les regions repetitives i emmascarament
Tal com s'ha fet anteriorment per a la seqüència inicial, introduim la seqüència extesa en format fasta al programa RepeatMasker a través del servidor EMBL RepeatMasker server . Obtenim els següents fitxers:
- Seqüència extesa emmascarada (expAC091491.masked.fa)
-
Resum del contingut de repeticions (expAC091491.repeat.seq.tbl)
- Anotació dels repeats de la seqüència extesa emmascarada (expAC091491.seq.out)
-
- grep hg13_dna expAC091491.seq.out | awk 'BEGIN{OFS="\t"}{print $5, $11, "repeat", $6,$7,".", ".", "."}' > expAC091491.seq.out.sencera.gff
- ./gff2ps_v0.98c expAC091491.seq.out.sencera.gff > expAC091491.seq.out.sencera.ps
- gv expAC091491.seq.out.sencera.ps
Per fer-nos una millor idea del grau d'emmascarament de la nostra seqüència extesa s'ha procedit a representar els elements repetitius agrupats mitjançant les següents comandes:
-
- grep hg13_dna expAC091491.seq.out | awk 'BEGIN{OFS="\t"}{if($11~/low-complexity/){SRC="low-comlexity";}else{SRC ="repeat"; }; print $5, SRC, "repeat", $6,$7,".", ".", "."}' > expAC091491.seq.out.gff
./gff2ps_v0.98c expAC091491.seq.out.gff > expAC091491.seq.out.ps
- gv expAC091491.seq.out.ps
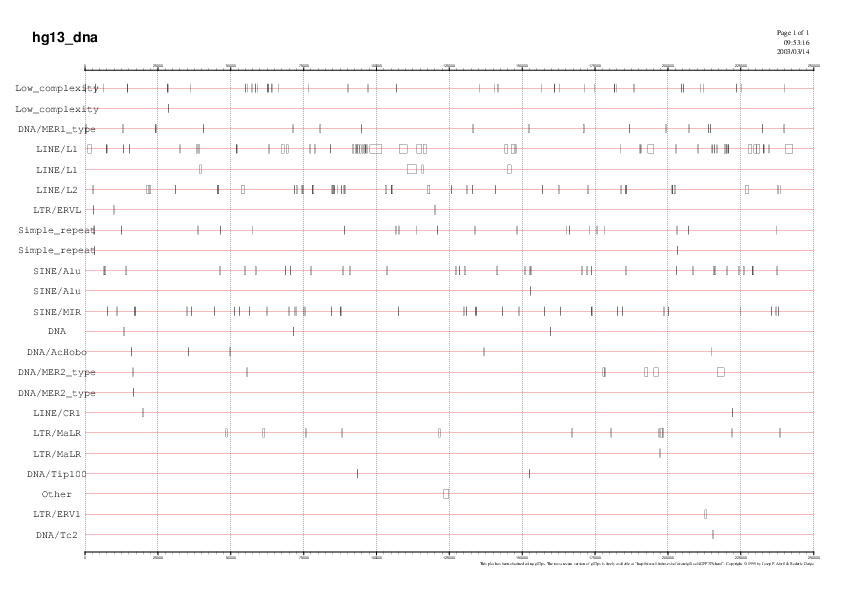 Fig 6: Distribució dels elements repetitius en la seqüència extesa |
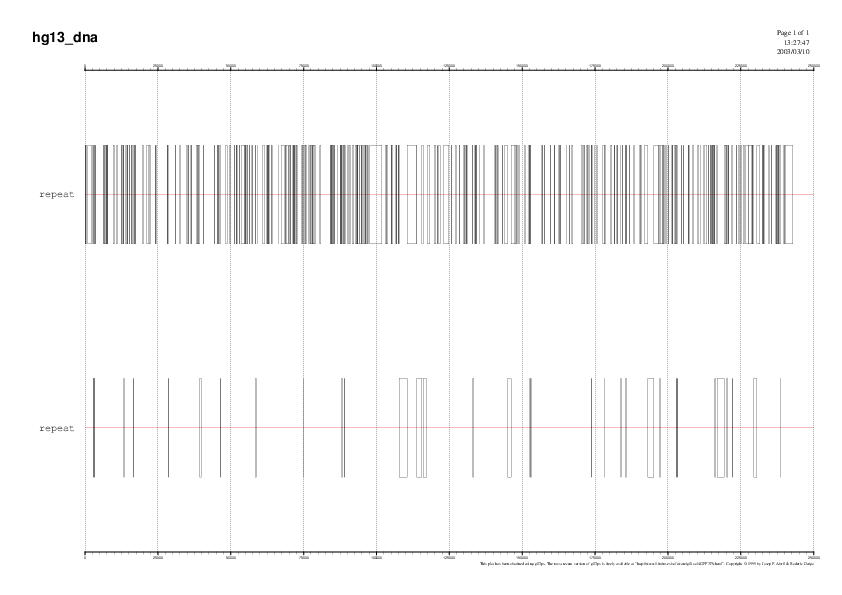 Fig 7: Distribució dels elements repetitius agrupats |
Predicció de gens "ab initio" amb la seqüència extesa
De nou s'ha fet la predicció de gens amb els programes geneid i genscan, aquest cop introduint la seqüència extesa en format fasta emmascarada. Els resultats obtinguts es mostren a continuació:
- Resultats del geneid per a la seqüència extesa emmascarada
- Resultats del genscan per a la seqüència extesa emmascarada
-
- gawk 'BEGIN{OFS="\t"}$2 ~ /Term|Intr|Init/ {print "AC091491", "genscan", $2, start=($4<$5 ? $4 : $5), end=($5<$4 ? $4 : $5),$13,$3,$7, $1}' expAC091491.genscan | sed 's/\.[0-9][0-9]$//' > expAC091491.genscan.gff
-
- ./gff2ps_v0.98c expAC091491.geneid.gff expAC091491.genscan.gff expAC091491.genepredictions.ps
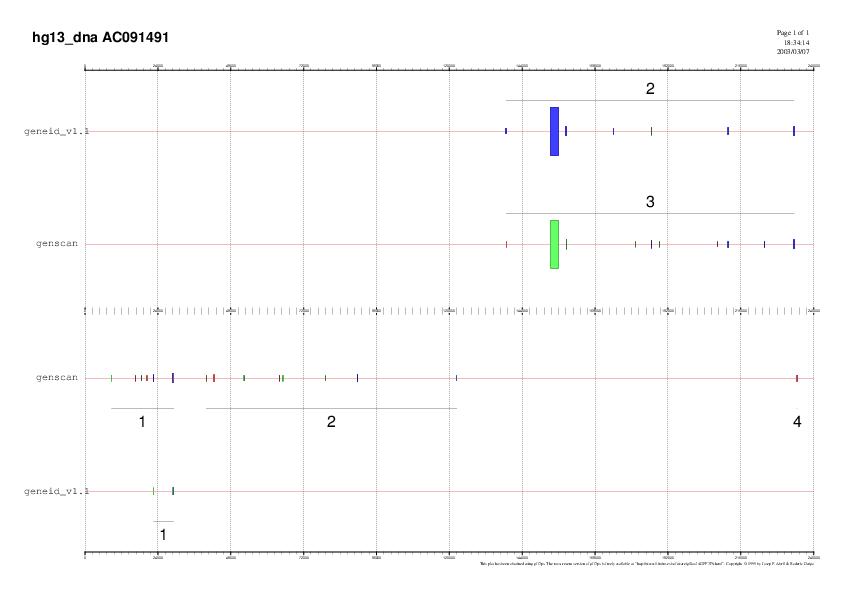
| Fig 8: Representació de la predicció de gens amb genscan i geneid per a la seqüència extesa. Com podem observar, els programes mostren el gen predit anteriorment en forward, tot i que abans el trobavem en reverse. Això és degut a que el nostre contig en ensamblar-se dins del cromosoma ho fa de forma inversa de manera que obtenim la seqüència complementària. Els exons d'aquest gen es corresponen amb els exons predits amb la seqüència sense extendre, excepte que en aquesta ocasió el programa geneid ha predit un exó first que coincideix amb la predicció del genscan. A més a més podem veure que s'han predit nous gens en reverse que no són objecte del nostre estudi. |
Validació de la predicció de gens amb la seqüència extesa
De nou s'ha procedit a validar la predicció de gens mitjançant ESTs humans obtinguts amb el programa Megablast. L'output d'aquest programa s'ha guardat en un fitxer expAC091491.blast.est que posteriorment s'ha processat amb el programa parseblast per tal de convertir-lo a format gff:
-
- parseblast -Gi expAC091491.blast.est | gawk 'BEGIN{OFS="\t"}{$2=$9;$6=$7=".";print}' > expAC091491.blast.est.gff
- ./gff2ps_v0.98c AC091491.blast.est.gff > expAC091491.blast.est.ps
A continuació s'ha procedit a obtenir els ESTs amb més d'un hsp i visualitzar els resultats amb ghostview, juntament amb les prediccions dels gens i els repeats, mitjançant les següent comandes:
-
- gawk -f getsplicedhsp.awk expAC091491.blast.est.gff > expAC091491.blast.est.spliced.gff
- ./gff2ps_v0.98c expAC091491.blast.est.spliced.gff expAC091491.geneid.gff expAC091491.genscan.gff expAC091491.seq.out.gff >expAC091491.spliced+repeats+genepredictions.ps
- gv expAC091491.spliced+repeats+genepredictions.ps
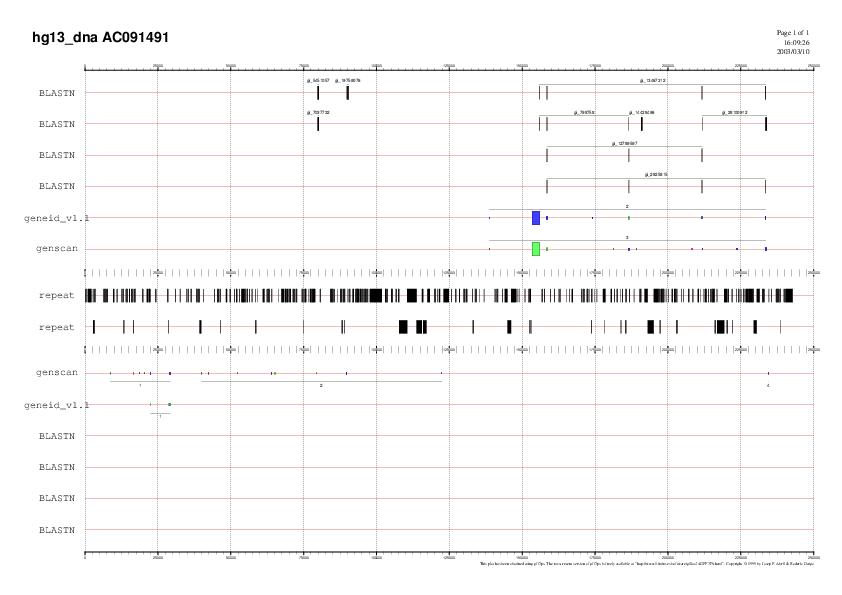
| Fig 9. Com podem observar els ESTs que validen els exons del nostre gen son els mateixos que els obtinguts anteriorment amb la seqüència no extesa. A més a més s'observen ESTs que suporten alguns dels exons dels gens predits en reverse. |