Arbre ADH1 i ADH3
Per comparar
les distribucions filogenètiques d’amdues isoformes (ADH1 i ADH3), hem
analitzat la filogènia limitada a aquestes dues ADHs, en els organismes
utilitzats en l’anàlisi anterior. En aquest cas, però, hem utilitzat el
programa Protpars, a més de fer-ho amb Neighbour.
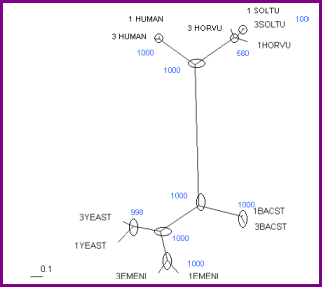 Neighbour:
Neighbour:
Els resultats obtinguts ens indiquen que l’evolució d’aquestes dues isoformes s’ha produït de manera diferent per cada espècie, és a dir, que primer es van separar les espècies i després van anar evolucionant les ADH independentment.
Les branques de
l’arbre ens mostren uns valors de bootstrap molt alts, per tant, aquesta
distribució és molt correcta.
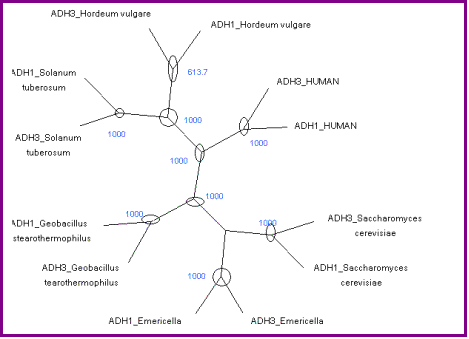 Protpars:
Protpars:
Utilitzant
aquest programa també veiem la mateixa distribució filogenètica de l’ADH1 i
l’ADH3, és a dir, l’evolució independent de cada isoforma en funció de cada
espècie.
En aquest cas,
els valors del bootstrap també són molt alts.
![]() Es
confirma que la proteïna és universal: es troba en totes les espècies degut a
la seva existència prèvia a la diversificació de les espècies; per aquest
motiu, les diferents espècies també presenten isoformes diferents de la
proteïna, en funció dels requeriments metabòlics afavorits per la Selecció
Natural, al llarg de l’evolució.
Es
confirma que la proteïna és universal: es troba en totes les espècies degut a
la seva existència prèvia a la diversificació de les espècies; per aquest
motiu, les diferents espècies també presenten isoformes diferents de la
proteïna, en funció dels requeriments metabòlics afavorits per la Selecció
Natural, al llarg de l’evolució.
![]() Quan es va fer un Psi-blast amb l’adh3 humana
per trobar les Adhs a diferents organismes va ser curiós que el blast donava
primer l’adh1 i després l’adh3 en ordre de similaritat de seqüència i amb el
mateix score.
Quan es va fer un Psi-blast amb l’adh3 humana
per trobar les Adhs a diferents organismes va ser curiós que el blast donava
primer l’adh1 i després l’adh3 en ordre de similaritat de seqüència i amb el
mateix score.
S’observa que en els altres organismes la nomenclatura és
la mateixa que en humans i això no concorda amb els arbres obtinguts, perquè
lògicament les seqüències amb el mateix nom s’haurien d’agrupar conjuntament; una
explicació a aquesta confusió podria ser que les seqüències d’organismes
s’haurien acumulat a Blast sota el nom de les adh humanes, amb les que
comparteixen un alt percentatge de similaritat. En tot cas, podem afirmar que
les seqüències adh1 i adh3 són paràlogues
i per aquest motiu s’agrupen juntes dins de cada espècie.