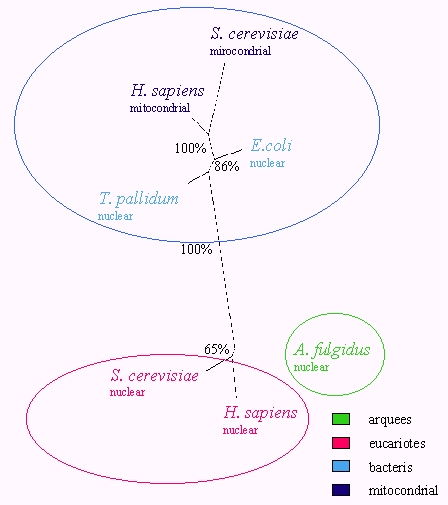
Fig.1 Arbre sense arrel mitjançant Neighbour-Joining
per a la LeuRS [1].
Els números en % indiquen la robustesa
de les branques per 1000 rèpliques de bootstrap .
La relació entre els nucleòtids (codons) i els aminoàcids s’estableix mitjançant les reaccions que porten a terme les aminoacil-tRNA sintetases. Com a conseqüència de la universalitat i antiguitat d’aquesta funció, es pensa que l’aparició del codi genètic precedeix la separació dels organismes en eucariotes i procariotes. Per aquest motiu, l’anàlisi filogenètic de gairebé totes les sintetases reflexa la distribució filogenètica de l’arbre universal de la vida. Però aquesta distribució no s'esdevé en l'anàlisi de la triptofanil i la tirosil-tRNA sintetases. La WRS i la YRS eucariotes estan més relacionades filogenèticament entre elles que no pas amb les corresponents WRS i YRS procariotes. Aquests resultats indiquen que les actuals WRS i YRS podrien haver aparegut després de la separació entre eucariotes i procariotes.
Aquesta família de proteïnes ha tingut molt temps per evolucionar
i incorporar modificacions. Així ho demostra la gran heterogeneïtat
entre els membres que componen aquesta família. Després de
la separació amb Drosophila melanogaster, s’han produït
diverses duplicacions dels gens donant lloc a un nombre variable de còpies
per cada gen. Algunes d’aquestes còpies ja s’han perdut, però
d'altres s’han conservat.
Les aminoacil-tRNA sintetases són un grup d’enzims encarregats d’activar els aminoàcids i transferir-los a les molècules del seu tRNA corresponent (primer pas en la biosíntesi de proteïnes). Són per tant un element clau en la traducció, ja que enllacen el món de les proteïnes amb el dels àcids nucleics. La importància d’aquestes molècules no només rau en la funció que exerceixen, sinó que és possible que el seu anàlisi pugui desvetllar algun dels secrets més profunds del codi genètic [5].
Cada cèl·lula conté 20 aminoacil t-RNA sintetases, una per cada aminoàcid (en eucariotes generalment hi ha dues aminoacil-tRNA sintetases per cada aminoàcid: una forma citosòlica i una forma mitocondrial). La funció que realitzen és comú per totes les proteïnes d'aquesta família, però són extremadament diverses en termes de grandària i estructura quaternària.
Les aminoacil t-RNA sintetases són unes molècules que
contenen empremtes gèniques que s’extenen més enllà
de l’ancestre universal de tota la vida, és per aquest motiu que
el seu anàlisi és un reflex de la dinàmica de l’evolució
en general. La seva filogènia molecular concorda amb la filogènia
acceptada de tots els organismes (Fig.1).

Fig.1 Arbre sense arrel mitjançant Neighbour-Joining
per a la LeuRS [1].
Els números en % indiquen la robustesa
de les branques per 1000 rèpliques de bootstrap .
Les sintetases es classifiquen en dues classes en funció dels
seus dominis estructurals comuns i les seves homologies de seqüència
[3]. Totes elles comparteixen en comú la seva funció general
i un domini estructural anomenat WHEP implicat en la unió d'ATP.
En la següent taula es pot veure la classificació de les aminoacil-tRNA
sintetases.
|
|
|
|
|
|
|
Isoleucina Valina Arginina Cisteïna Metionina |
Treonina Alanina Glicina Prolina Histidina |
|
|
|
|
Glutamina Lisina-I |
Asparragina Lisina-II |
|
|
|
|
Triptòfan |
|
Recentment es va poder observar que un grup d'aquesta família
de proteïnes, concretament les de classe I, tenien en comú
una regió conservada anomenada 'HIGH' (formada pel tetrapèptid
His-Ile-Gly-His) situada a la part N-terminal de la proteïna.
Aquesta regió 'HIGH' forma part del lloc d'unió a adenilat.
Les 10 aminoacil-tRNA sintetases de classe I contenen un plegament de Rossmann,
caracteritzat pel motiu conservat 'HIGH' i el motiu 'KMSKS'. Les altres
10 aminoacil-tRNA sintetases de classe II estan caracteritzades per tres
seqüències motiu degenerades.
A causa del seu paper central en l’establiment del codi genètic, es creu que les aminoacil-tRNA sintetases es troben entre les primeres proteïnes que van aparèixer en l’evolució. D’acord amb l’origen antic d’aquests enzims, les anàlisis filogenètiques de les seqüències de les aminoacil-tRNA sintetases mostren que aquests enzims s’agrupen segons la seva especificitat d’aminoàcid i no segons la seva posició en l’arbre filogenètic universal. Això indica que les aminoacil-tRNA sintetases van aparèixer i evolucionar abans que l’arbre de la vida es dividís en els tres dominis actualment reconeguts (Archaea, Bacteria, Eukarya).
En aquest treball es presenta una anàlisi de les relacions filogenètiques
entre les triptofanil i tirosil-tRNA sintetases eucariotes i procariotes,
les quals mostren un patró filogenètic totalment diferent
a les relacions evolutives que presenten la resta d’aminoacil-tRNA sintetases.
També s’exposa un anàlisi exhaustiu dels gens humans que
codifiquen per aquesta família de proteïnes.
- Caracteritzar i conèixer una família de proteïnes molt antiga que porta milions d’anys evolucionant.
- Esbrinar les seves relacions evolutives a través de l’anàlisi filogenètic dels seus components.
- Comparar la filogènia entre la triptofanil i tirosil-tRNA sintetases (dos enzims que presenten un alt grau de similaritat estructural, la qual cosa suggereix un ancestre comú recent) i la resta de les aminoacil-tRNA sintetases.
- Fer ús de les eines informàtiques per tal d’analitzar
els gens que codifiquen per aquestes proteïnes: dominis conservats,
estructura exònica, etc.
1.-Obtenció de les seqüències
Les seqüències s’han obtingut a partir d’un document
que llista les entrades de Swissprot
pels 20 tipus d’aminoacil-tRNA sintetases. Aquest document també
indica a quina classe pertany cada sintetasa (classe I o classe II) i si
hi ha resolta l’estructura tridimensional d’algun dels membres de cada
tipus.
L'obtenció dels membres de la família mitjançant
la cerca per similaritat de seqüències amb el BLASTP
detecta molt pocs membres degut als milions d’anys d’evolució que
porten aquestes molècules. Per aquest motiu totes les seqüències
s’han obtingut a partir de Swissprot.
2.-Anàlisi filogenètic
2.1.- Aliniament de les seqüències
Les seqüències han estat aliniades mitjançant el
Clustalw.
En la realització del 2n arbre s’han aliniat els motius conservats
'KMSKS' característics de les aminoacil-tRNA sintetases de classe
I. Com que el Clustalw no feia una bona aliniació d’aquest motiu
conservat, s’ha utilitzat el programa Jalview
per fer un aliniament
"manual"
a partir del patró de l’aliniament dels motius conservats que feia
Pfam
i de les referències de l'índex bibliogràfic
[4].
2.2.- Construcció d’arbres
Els arbres filogenètics s’han construït utilitzant el mètode
de màxima parsimònia, a partir del paquet informàtic
Phylip versió 3.5. La representació gràfica dels arbres
sense arrel ha estat realitzada mitjançant el Treeview,
i la robustesa de les branques ha estat obtinguda pel Seqboot, mitjançant
anàlisis de bootstrap de 100 rèpliques.
3.-Informació genòmica: dominis conservats, estructura gènica i localització cromosòmica
Per a l’estudi dels dominis conservats de les aminoacil-tRNA sintetases
s'han utilitzat les bases de dades Prosite
i
Interpro.
Per determinar la localització cromosòmica de les aminoacil-tRNA
sintetases s’ha fet servir Ensembl.
1. Distribució filogenètica
La primera anàlisi s’ha realitzat a partir de l’aliniament múltiple de les seqüències senceres de diferents aminoacil-tRNA sintetases de classe I, incloses la triptofanil i tirosil-tRNA sintetases eucariotes i procariotes. L’arbre resultant (fig. 2) agrupa la WRS i la YRS segons el seu origen eucariota o procariota i no segons la seva especificitat d’aminoàcid. En canvi, les altres dues sintetases analitzades, la metionil i l’arginil-tRNA sintetasa sí que mostren una agrupació definida per la seva especificitat d’aminoàcid.
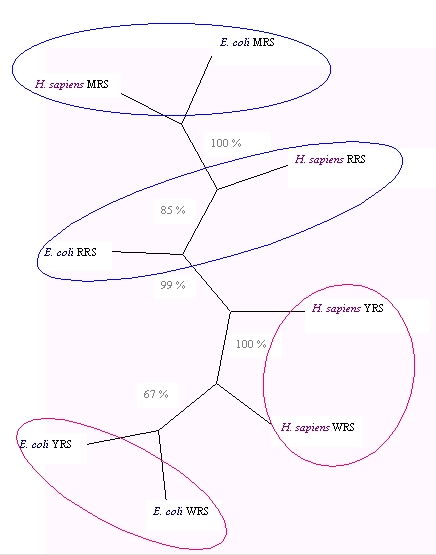
La segona anàlisi s’ha realitzat a partir de l’aliniament de les regions conservades que contenen el motiu “KMSKS” característic de les aminoacil-tRNA sintetases de classe I. Totes les seqüències aquí analitzades són WRS i YRS i inclouen sintetases provinents de genomes mitocondrials i sintetases de l’arquea A. fulgidus. L’arbre resultant mostra una clara distribució de les sintetases segons si són eucariotes-arqueas o bacterianes-mitocondrials, la qual cosa dóna suport a la hipòtesi que les WRS i les YRS actuals van aparèixer després de la separació entre eucariotes-arqueas i bacteris.
D’acord amb la teoria endosimbiòtica, les WRS i YRS mitocondrials
s’agrupen amb les sintetases d’origen bacterià. En canvi, les sintetases
de l’arquea s’agrupen amb les sintetases eucariotes, en concordància
amb la hipòtesi que el nucli de les cèl·lules eucariotes
prové d’una endosimbiosi ancestral amb un arquea (fig. 3).
![]() YRS i WRS eucariotes i arqueas.
YRS i WRS eucariotes i arqueas.
![]() YRS i WRS bacterianes i mitocondrials.
YRS i WRS bacterianes i mitocondrials.
Fig. 3 Arbre sense arrel de màxima parsimònia obtingut
a partir de l’aliniament dels motius
conservats 'KMSKS' de 18 seqüències de WRS i YRS. Els números
corresponen al percentatge
obtingut per bootstrap a cada branca particular (100 rèpliques).
Els noms complets de les espècies que s’hi mostren són:
Neurospora
crassa, Podospora anserina,
Bacillus caldotenax, Archaeoglobus
fulgidus, Bacillus
stearothermophilus, Bacillus
subtilis,
Escherichia coli,
Saccharomyces
cerevisiae,
Bos taurus, Homo
sapiens, Oryctolagus cuniculus
i Mus musculus.
Abrevacions
de les sintetases: YRS, tirosil-tRNA sintetasa; WRS, triptofanil-tRNA
sintetasa; mit, mitocondrial.
2. Estudi dels gens que codifiquen per les aminoacil-tRNA sintetases humanes
A continuació s’exposa una anàlisi exhaustiu dels gens
humans que codifiquen per aquesta família de proteïnes. El
genoma humà conté 20 aminoacil-tRNA sintetases distribuïdes
en diferents cromosomes. El nombre de gens que codifiquen per cada aminoacil-tRNA
sintetasa és força variable observant-se casos amb una sola
còpia del gen i casos amb 6 còpies. La llargada dels gens
oscil·la entre 1.4 Mb (CRS) i 228.1 Mb (E-PRS). El nombre d’exons
de cada gen també és molt variable, entre 1 (ARS) i 34 (IRS)
exons. També és força destacable el fet que el gen
que codifica per la prolil-tRNA sintetasa és comú amb un
dels gens que codifica per la glutamil-tRNA sintetasa.
ANÀLISI DELS GENS HUMANS QUE CODIFIQUEN PER A LES AMINOACIL-tRNA SINTETASES
| Aminoacil-tRNA sintetases humanes | |||||
|---|---|---|---|---|---|
| . |
(Número d'accés) |
|
|
d'exons |
(Número d'accés) |
|
AARS |
P54136 |
|
|
|
|
|
P49589 |
|
|
|
|
|
|
O14563 P07814 |
228075312 - 228140152 bp (228.1 Mb) en cromosoma 1 |
|
29 |
|
|
|
P47897 |
|
|
|
|
|
|
P41252 |
|
|
|
|
|
|
Q9NSE1 |
|
|
|
|
|
|
P56192 |
68354941 - 68357494 bp (68.4 Mb) en cromosoma 12 |
|
4 |
|
|
|
P54577 |
|
|
|
|
|
|
P23381 |
86169124 – 86210654 bp (86.2 Mb) en cromosoma 14 |
|
11 |
|
|
|
P26640 |
36334346 - 36346588 bp (36.3 Mb) en cromosoma 6 |
|
29 |
|
|
|
AARS |
P49588 |
71478163 – 71508567 bp (71.5 Mb) en cromosoma 16 75130215 – 75132228 bp (75.1 Mb) en cromosoma 16 75140359 - 75142388 bp (75.1 Mb) en cromosoma 16 75149404 – 75152498 bp (75.1 Mb) en cromosoma 16 71693865 - 71739976 bp (71.7 Mb) en cromosoma 16 |
|
20 5 4 3 2 |
|
|
P14868 |
|
|
|
|
|
|
O43776 |
57507702 – 57508757 bp (57.5 Mb) en cromosoma 18 |
q21.3 |
2 |
|
|
|
P41250 |
55930988 – 55931263 bp (55.9 Mb) en cromosoma 17 |
|
1 |
|
|
|
P12081 |
144001946 – 144019718 bp (144.0 Mb) en cromosoma 5 143855521 - 143873309 bp (143.9 Mb) en cromosoma 5 143872918 – 143880764 bp (143.9 Mb) en cromosoma 5 |
|
13 13 13 |
|
|
|
Q15046 |
69047553 – 69047897 bp (69.0 Mb) en cromosoma 16 |
q22.3 |
1 |
|
|
|
O95363 |
|
|
|
|
|
|
P07814 |
|
|
|
|
|
|
P49591 |
|
|
|
|
|
|
P26639 |
99436321 – 99523695 bp (99.4 Mb) en cromosoma 15 |
|
19 |
|
|
Veient aquesta taula ens adonem que el gen que codifica per la glutamil-tRNA
sintetasa és comú amb el gen que codifica per la prolil-tRNA
sintetasa. Aquest gen localitzat al cromosoma 1 expressa un enzim bifuncional.
A continuació s'analitzen els dominis d'aquesta proteïna amb
característiques tan peculiars.
2.1 El cas de la E-P-tRNA sintetasa: un enzim bifuncional
S’ha observat que els genomes de certs organismes no contenen la prolil-tRNA sintetasa, la qual és essencial per dur a terme els processos de traducció de l’RNAm a proteïna. Malgrat tot, aquests organismes contenen un enzim que és capaç de realitzar l’activitat de dues aminoacil-tRNA sintetases simultàniament. La glutamil-prolil-tRNA sintetasa és un enzim bifuncional que pot aminoacetilar el seu tRNA amb àcid glutàmic o prolina. Aquest enzim només és present en organismes eucariotes superiors.
En humans i en Drosophila l’activitat de la glutamil-tRNA sintetasa
(GluRS) i la prolil-tRNA sintetasa (ProRS) està continguda en una
sola cadena polipeptídica, tot i que aquests enzims provenen de
classes diferents i han evolucionat per camins diferents. La glutamil-prolil-tRNA
sintetasa està composta per 1440 aminoàcids que provenen
de 29 exons. Els exons que codifiquen per la glutamil-tRNA sintetasa i
la prolil-tRNA sintetasa estan situats als extrems oposats del gen, separats
per una o més còpies del domini WHEP característic
d’aquesta familia de proteïnes.
2.2 El domini conservat WHEP-TRS
Les diferents aminoacil-tRNA sintetases d’eucariotes superiors comparteixen en comú un domini conservat de 46 aminoàcids anomenat WHEP-TRS [2]. Aquest domini es troba entre una i sis vegades en cada enzim. En els enzims multifuncionals de mamífers se’n troben 3 còpies localitzades entre la regió N-terminal on hi ha la glutamil-tRNA sintetasa i el domini C-terminal on hi ha la prolil-tRNA sintetasa. Aquest domini WHEP també es troba en la regió N-terminal d’altres aminoacils com la triptofanil-tRNA sintetasa, la histidil-tRNA sintetasa i la glicil-tRNA sintetasa. Aquest domini WHEP conté una regió central alfa-hèlix que té un paper molt important en l’associació de les aminoacil-tRNA sintetases en complexes multienzimàtics.
El fet que diferents gens al llarg de l’evolució s’hagin fusionat donant lloc a proteïnes de fusió com la glutamil-prolil-tRNA sintetasa sembla que és una característica exclusiva dels eucariotes superiors. Aquest tipus d’organització estructural que dóna lloc a complexes multisintètics és un mecanisme que permet incorporar diferents activitats catalítiques en un sol enzim.
La seqüència patró basada en les 29 primeres posicions
del domini WHEP s’exposa a continuació:
|
|
|
| Seqüència patró | [QY]-G-[DNEA]-x-[LIV]-[KR]-x(2)-K-x(2)-[KRNG]-[AS]-x(4)-[LIV]-[DENK]-x(2)-[IV]-x(2)-L-x(3)-K |
2.3 Estructura exònica
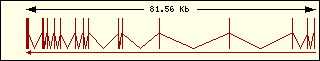
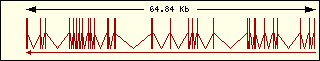
L'anàlisi de l'estructura exònica de les aminoacil-tRNA sintetases humanes mostra que són molt diferents entre elles, la qual cosa recolza el fet que fa molt temps que aquests enzims estan evolucionant, cadascun pel seu camí.
Aminoacil-tRNA
sintetases humanes |
|
|||
Classe
I |
Classe
II |
|
||
|
Arginina
 |
Alanina
 |
|||
|
Cisteïna
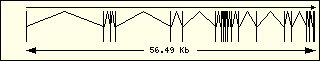 |
Àcid
aspàrtic
 |
|||
|
Àcid
glutàmic
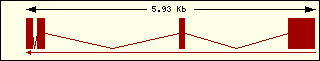  |
Asparagina
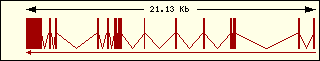 |
|||
|
Glicina
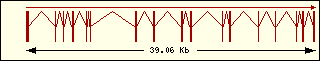 |
||||
|
Glutamina
|
|
|||
|
Isoleucina
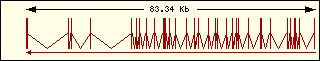 |
Histidina
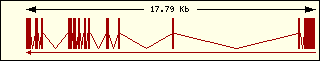 |
|||
|
Leucina
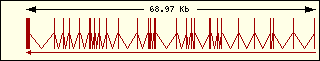 |
Lisina
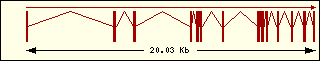 |
|||
|
Metionina
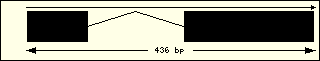 |
Fenilalanina
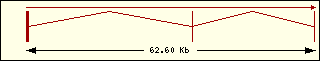 |
|||
|
Tirosina
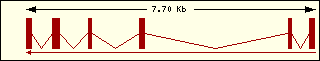 |
Prolina
 |
|||
|
Triptofan
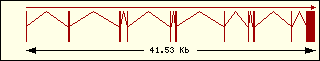 |
Serina
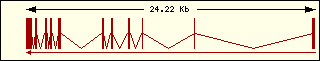 |
|||
|
Valina
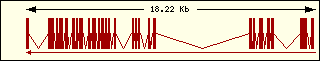 |
Treonina
 |
|||
2.4 Variabilitat
nucleotídica (SNPs) dins l’espècie humana:
Després de buscar a OMIM i a SNPs al NCBI, no hem trobat cap
malaltia, ni cap mutació, que afecti, en el sentit estricte de la
paraula, les aminoacil-tRNA sintetases. Això pot estranyar en un
principi. Però, tenint en compte que estem parlant d’una família
de proteïnes que es remunten als orígens de la vida, i que
es troben a tots els organismes vius -des de bacteris, fins els mateixos
humans i arqueas- i que, com sabem, tenen un paper rellevant en l’establiment
del codi genètic, hem de suposar que el seu paper com a enzims,
és tan important per a la vida que qualsevol mutació que
les afecti és incompatible amb la mateixa vida, o si més
no, amb la vida, tal i com nosaltres l’entenem.
Anàlisi filogenètic
Una possible explicació d’aquests resultats és que després de l’aparició de l’ancestre de la cèl·lula eucariota, almenys un dels gens que codificaven per les YRS o WRS primitives es va perdre en la branca dels Archaea-Eukarya. Això s’hagués pogut solucionar pel reemplaçament del gen perdut per un al·lel duplicat d’una altra sintetasa (de fet, tal com s'ha explicat, hi han hagut vàries duplicacions d’aquestes molècules al llarg de la història evolutiva de les espècies). Però aquesta hipòtesi requeriria algun tipus d’explicació que actualment es desconeix sobre com un enzim funcional i essencial es pot reemplaçar per la duplicació d’un altre enzim, funcionalment diferent.
Una altra hipòtesi seria que una sintetasa ancestral fos capaç d’interaccionar amb ambdós aminoàcids i pogués unir selectivament la tirosina i el triptofan al seu tRNA corresponent (sintetases bifuncionals). Aquesta sintetasa després de la separació entre bacteris d’eucariotes-arqueas, s’hauria duplicat independentment en ambdós branques. El punt dèbil d’aquesta teoria és que requereix un esdeveniment de doble duplicació i divergència.
Tanmateix, aquests resultats demostren esdeveniments
de gran dinamisme més tardans a la separació dels bacteris
amb els eucariotes-arqueas, que reflexen que malgrat la importància
d'aquestes molècules s'han esdevingut grans canvis.
Estudi dels gens que codifiquen per les aminoacil-tRNA sintetases humanes
De l’anàlisi de la localització
cromosòmica i l’estructura exònica dels gens de les aminoacil-tRNA
sintetases humanes se’n poden extreure unes quantes conclusions: la més
evident és l’enorme diversitat que hi ha entre els diferents membres
de la família, fàcilment explicable tenint en compte que
es tracta d'una de les famílies de proteïnes més antigues
i que per tant han tingut molt temps per evolucionar i incorporar modificacions.
En segon lloc, en els casos on el nombre de gens que codifiquen per una
de les aminoacils-tRNA sintetases és superior a 1, s’observa que
el nombre de còpies sempre és parell (2, 4 i 6). Aquest fet
podria evidenciar un origen d’aquest nombre de còpies per successives
duplicacions al llarg de la història de l’evolució. En el
cas de la histidil-tRNA sintetasa on s’observen 4 còpies del gen
-totes elles localitzades en el cromosoma 5- les duplicacions s’haurien
esdevingut fa més de 990 MA, després de la separació
entre Drosophila melanogaster i Primats, ja que en el genoma de
Drosophila
melanogaster només se’n troba una còpia.
1. Bacardit M., Coll M., Gabernet N. Hostes vingueren i a sorgir ens empenyeren! Origen endosimbiòtic dels mitocondris i arbre sense arrel de tots els organismes vius, a partir de les aminoacil t-RNA sintetases. Pràctiques d’evolució, 2001.
2. Cerini C., Kerjan P., Astier M., Gratecos D., Mirande M., Semeriva M. 1991. A component of the multisynthetase complex is a multifunctional aminoacyl-tRNA synthetase. EMBO J Dec;10(13):4267-77.
3. Eriani G., Delarue M., Poch O., Gangloff J., and Moras D. 1990. Partition of tRNA synthetases into two classes based on mutually exclusive sets of sequence motifs. Nature 347:203-206.
4. Ribas de Pouplana, Ll., Frugier M., Quinn C., and Schimmel P. 1996. Evidence that two present-day components needed for the genetic code appeared after nucleated cells separated from eubacteria. Proc. Natl. Acad. Sci. USA 93:166-170.
5. Woese, C., Olsen G.J., Ibba M., Söll
D. 2000. Aminoacyl-tRNA synthetases, the genetic code, and the evolutionary
process. Microbiology and Molecular Biology Reviews 64: 202-236.
Per qualsevol dubte o aclariment no ho dubtis
i escriu-nos un e-mail: merce.bacardit01@campus.upf.edu
montserrat.coll02@campus.upf.edu
nuria.gabernet01@campus.upf.edu